大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
DNA甲基化在调控基因表达和睾丸发育中发挥重要作用。WGBS 和 RNA-Seq 数据综合分析可以绘制DNA甲基化与基因表达关系的全面图谱,从而鉴定受甲基化影响的关键基因。如对小鼠睾丸组织中 WGBS 和 RNA-Seq 的分析揭示了与类固醇产生和精子发生相关的基因和通路,揭示了PM2.5如何通过甲基化破坏雄性生殖功能。对胚胎天鹅绒山羊皮肤样本(胚胎第65天和胚胎第120天天鹅绒山羊)中的 WGBS 和 RNA-Seq 分析鉴定了对早期卵泡发育至关重要的基因和机制。然而,DNA 甲基化对猪睾丸发育过程中基因表达的作用仍不清楚。阐明这种作用对于了解雄性生殖发育中的基因表达变化至关重要。
近日,湖北省农业科学院畜牧兽医研究所梅书棋和彭先文共同通讯在《International Journal of Molecular Sciences》(Int J Mol Sci)(IF:5.6)期刊发表题为“Comprehensive Analysis of Methylome and Transcriptome to Identify Potential Genes Regulating Porcine Testis Development”的研究成果。研究聚焦于猪睾丸发育过程中的DNA甲基化模式,以及这些模式如何与基因表达相关联。通过对猪发育过程中的睾丸组织进行WGBS和RNA-Seq分析,筛选出关键候选差异甲基化基因(DMG)及其富集信号通路。为DNA甲基化探索及其潜在调控机制提供重要见解,促进对雄性发育生物学的更深入理解,还为未来的生殖健康研究和治疗提供了新视角。

本研究使用RNA测序(RNA-Seq)和全基因组亚硫酸盐测序(WGBS)技术,检测了60日龄(60d)和180日龄(180d)的猪睾丸组织中的差异表达基因(DEGs)及其甲基化水平。研究揭示了DNA甲基化主要发生在胞嘧啶-鸟嘌呤(CG)中,分析鉴定出106282个差异甲基化区域(DMRs),对应于12385个差异甲基化基因(DMGs)。进一步对RNA-Seq和WGBS数据的综合分析揭示了1083个与DEGs表达呈负相关的DMGs。GO分析显示这些基因在精子发生、生殖细胞发育和精子细胞分化中显著富集。富集基因的筛选揭示高甲基化抑制ADAM30、ADAM3A、DPY19L2、H2BC1、MAK、RPL10L、SPATA16和YBX2的表达,而低甲基化则增强CACNA1I、CADM1、CTNNB1、JAM2和PAFAH1B3的表达。此外,通过亚硫酸盐测序PCR(BSP)检测关键基因ADAM3A、ADAM30、YBX2、JAM2、PAFAH1B3和CTNNB1的甲基化状态。本研究为猪睾丸发育的表观遗传调控机制提供了见解。
研究方法:
- 使用WGBS技术对60日龄(60d)和180日龄(180d)的猪睾丸组织样本进行DNA甲基化分析。
- 利用RNA-Seq技术分析同一组织样本的基因表达。
- 通过生物信息学工具对WGBS和RNA-Seq数据进行分析,鉴定差异甲基化区域(DMRs)和差异表达基因(DEGs)。
- 进行GO富集分析和KEGG通路分析,以揭示DMRs相关基因的功能。
- 使用实时定量PCR(RT-qPCR)和亚硫酸盐测序PCR(BSP)验证关键基因的表达和甲基化状态。
结果图形
(1)猪睾丸组织中的DNA甲基化模式
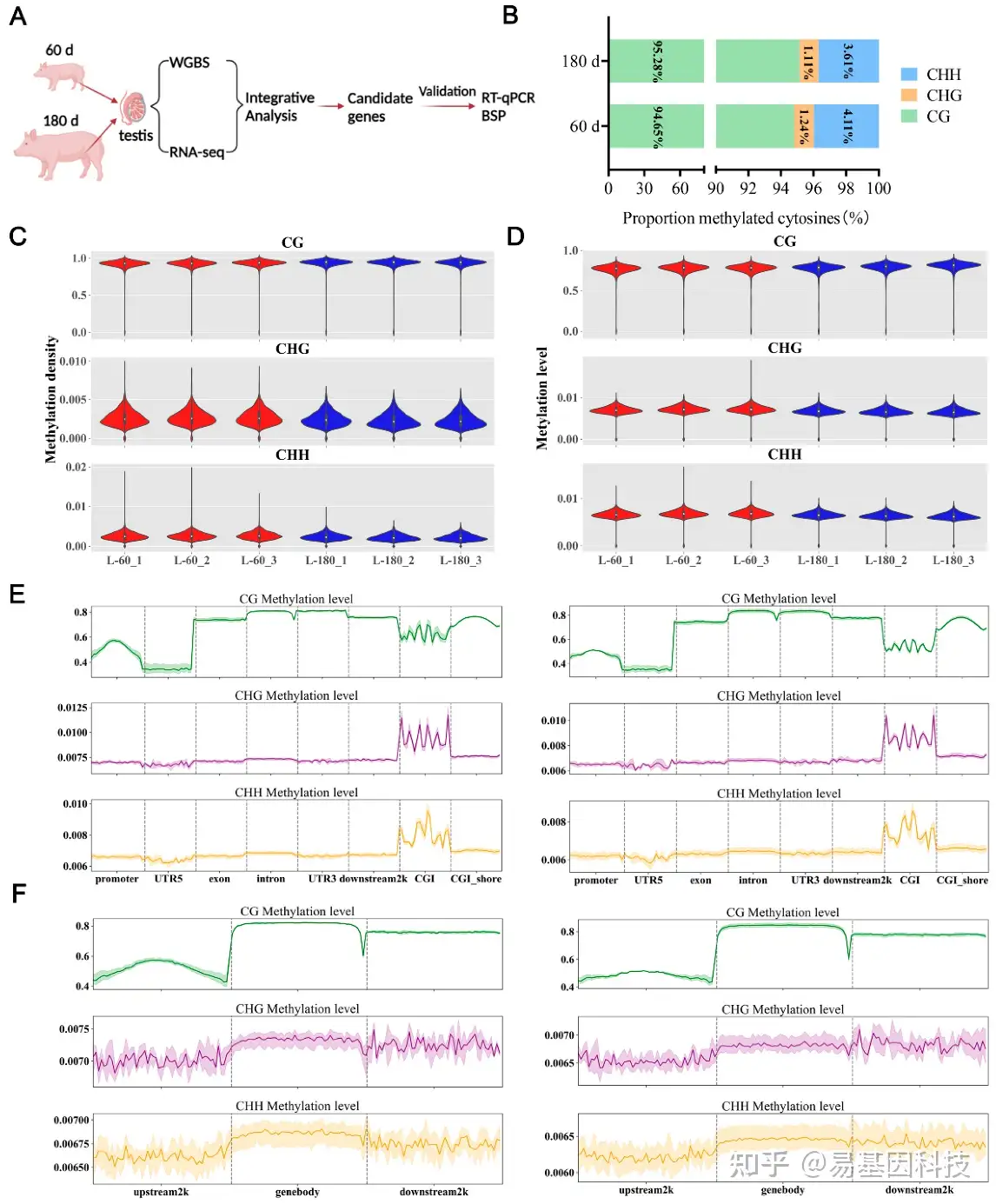
图1:猪睾丸组织中DNA甲基化。
A. 实验设计示意图。
B. 堆叠条形图显示60日龄和180日龄猪睾丸组织中三种不同甲基化类型(mCG、mCHG和mCHH;H=A、C或T)的平均比例。甲基化类型分别用绿色、橙色和蓝色表示。
C. 小提琴图描绘了三种不同序列环境(CG、CHG和CHH;H=A、C或T)中的甲基化密度。每一行表示一个不同的环境。Y轴表示甲基化密度,X轴代表不同的样本(60日龄和180日龄)。
D. 小提琴图展示三种不同序列环境(CG、CHG和CHH;H = A、C或T)中甲基化水平分布。
E. 60日龄与180日龄睾丸组织中不同基因组元件的甲基化水平。每一行表示一个不同的环境。X轴表示不同的基因组元件,Y轴表示甲基化水平。左侧和右侧面板分别显示60日龄和180日龄睾丸组织的甲基化水平分布。绿色、紫色和黄色线条分别代表CG、CHG和CHH甲基化水平。
F. 60日龄与180日龄睾丸组织中上下游±2K区域的甲基化水平分布。
(2)DMR鉴定
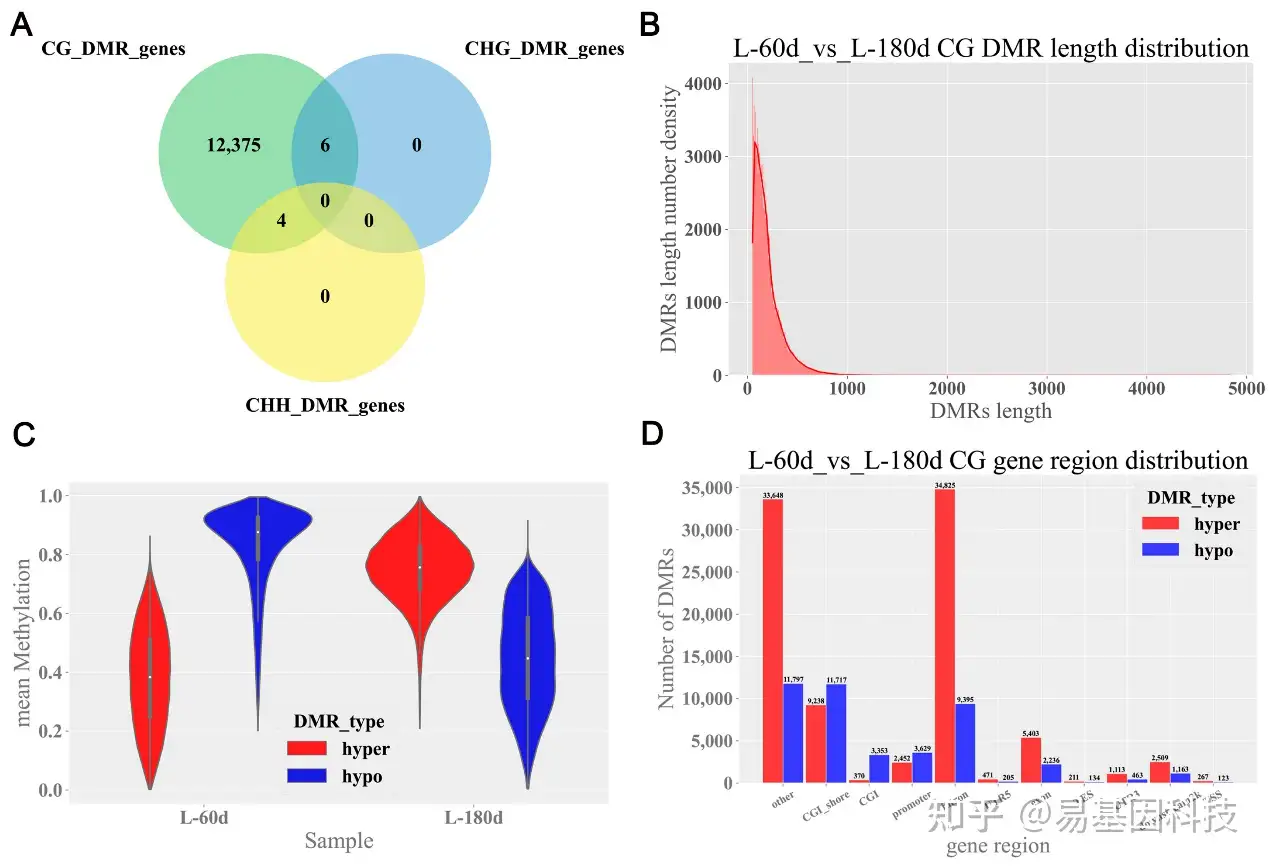
图2:猪睾丸组织中差异甲基化区域(DMRs)的鉴定。
A. CG、CHG和CHHDMR相关基因维恩图。
B. CG环境中DMRs长度分布。X轴表示DMRs长度;Y轴表示每个长度的密度,拟合曲线的DMRs长度分布用红色标出。
C. CG环境中甲基化水平分布小提琴图。X轴代表不同年龄组(60日龄和180日龄),Y轴代表平均甲基化水平的值。
D. CG环境中DMR锚定区域。X轴和Y轴分别表示不同基因区域和DMRs数量。
(3)DMG功能富集分析
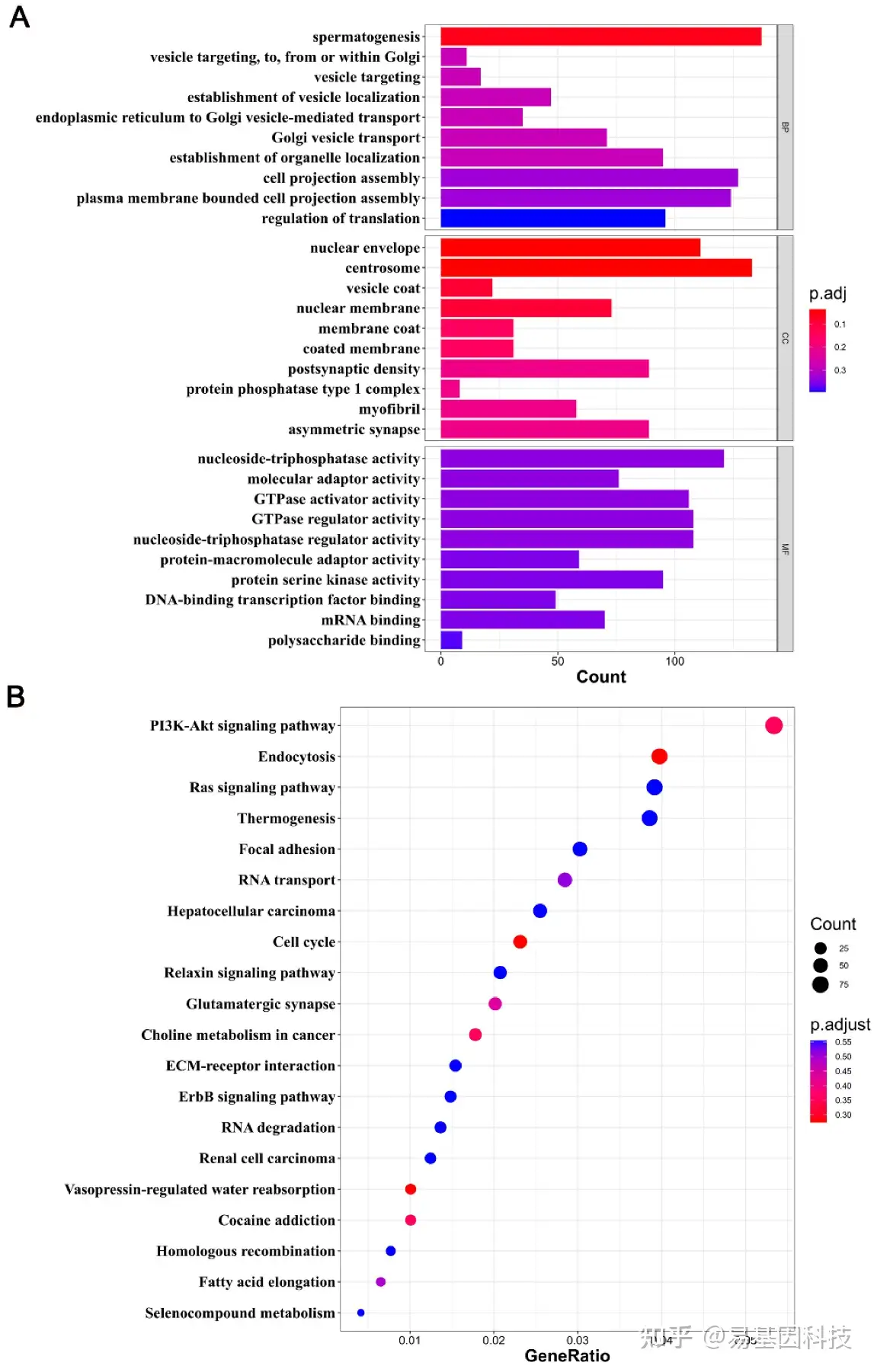
图3:差异甲基化基因(DMGs)的GO和KEGG分析。
A. 条形图显示CG中GO富集分析结果。Y轴描述不同的GO分析,X轴显示DMGs数量。BP、CC和MF分别代表生物过程、细胞组分和分子功能。
B. KEGG通路富集散点图示例。Y轴和X轴分别显示通路名称和基因比例。点大小表示DMGs数量,颜色对应于不同的q值。
(4)DNA甲基化与差异表达基因(DEG)的关联分析
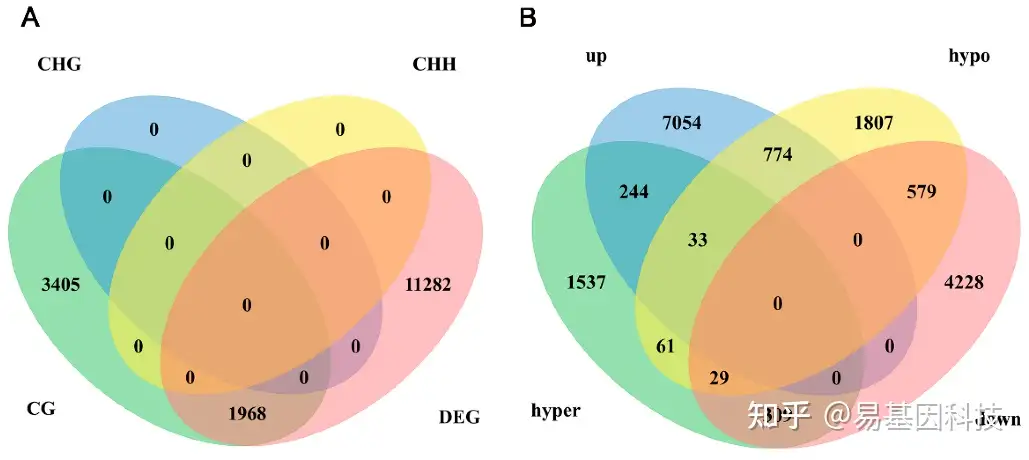
图4:DNA甲基化与基因表达之间的相关性分析。
A. DEGs和DMGs(CG、CHG和CHH环境)之间的重叠分析维恩图。
B. CG环境中的DMGs与DEGs维恩图,区分了与高甲基化(hyper)和低甲基化(hypo)相关基因,以及表达水平升高(up)和降低(down)的基因。
(5)与DEGs负相关DMG关键通路的富集
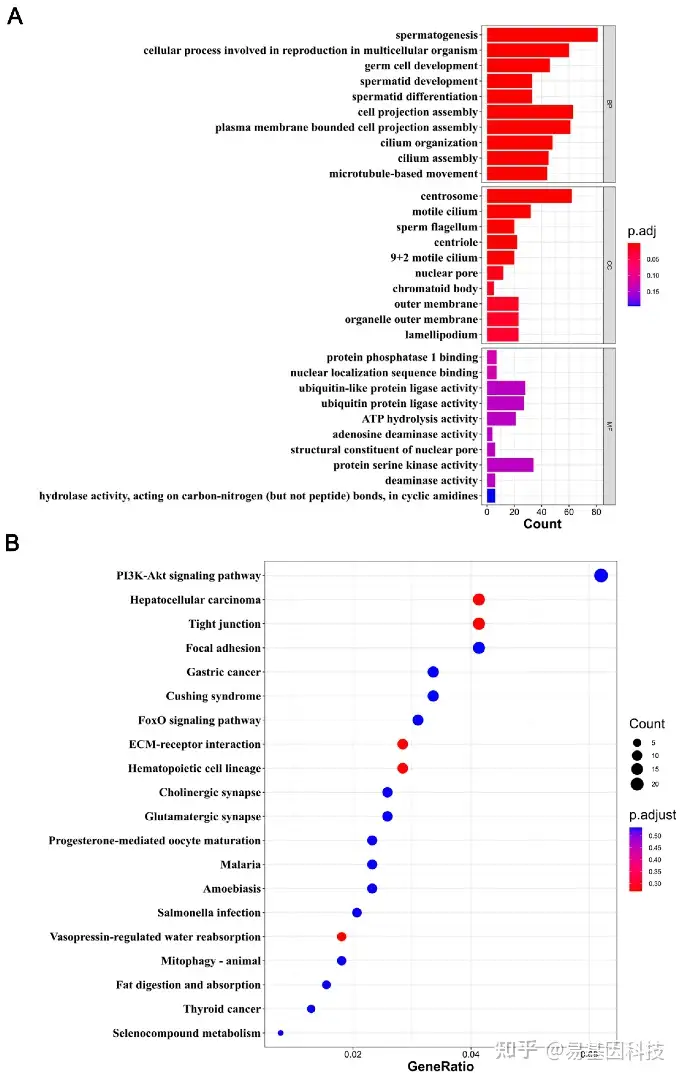
图5:与DEGs负相关的DMGs的功能富集分析。
A. 条形图显示了与DEGs负相关的DMGs的GO富集。
B. 散点图展示了与DEGs负相关的DMGs的KEGG通路。
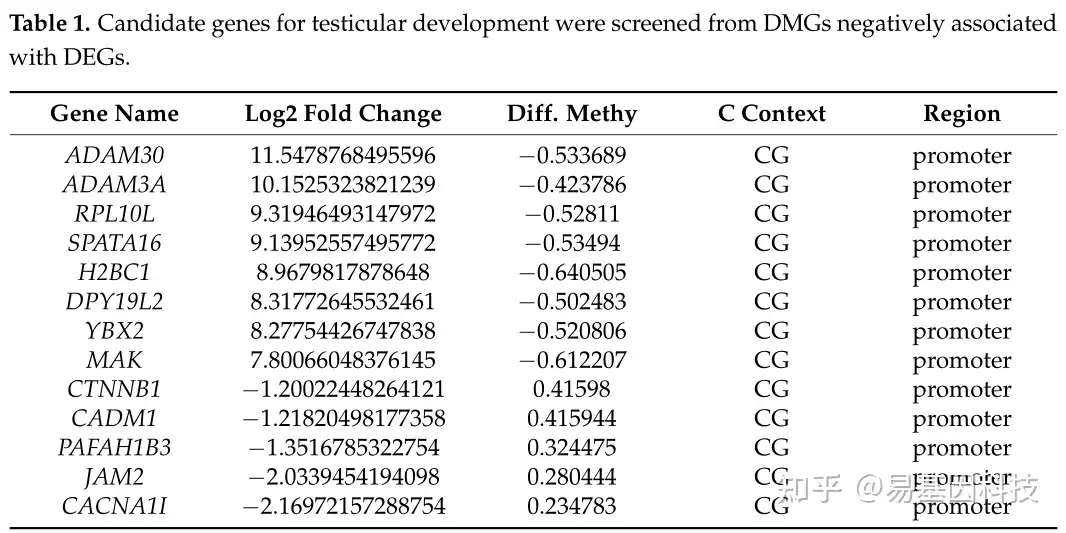
表1:从与DEGs负相关的DMGs中筛选睾丸发育候选基因。
(6)鉴定参与睾丸发育的基因
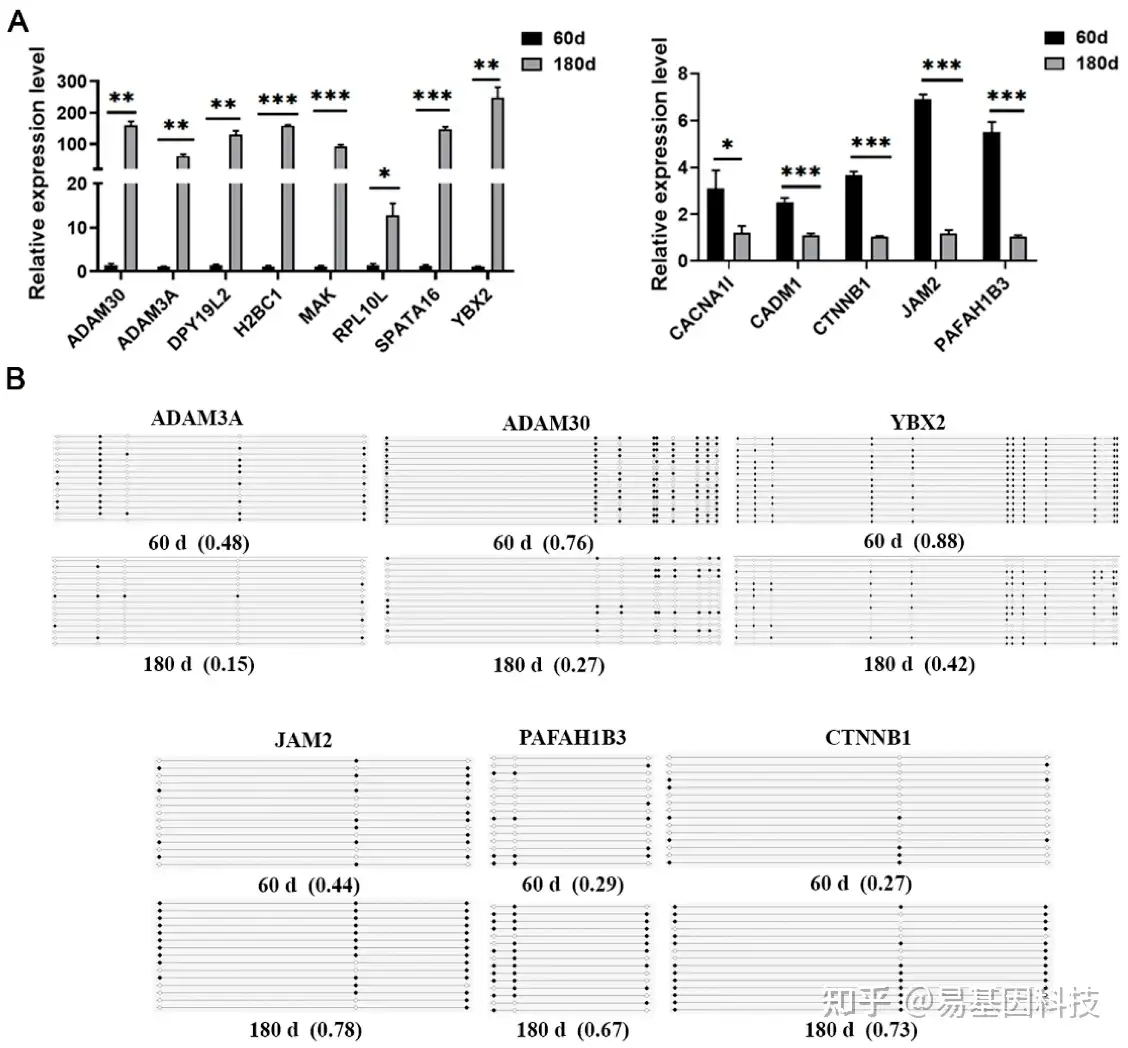
图6:DMGs及其表达模式的验证。
A. 基因的RT-qPCR表达水平与RNA-Seq数据一致;高甲基化抑制基因表达,而低甲基化刺激基因表达。
B. BSP检测到的DNA甲基化与WGBS数据一致。进行三次生物学重复实验,β-actin为对照基因。*p<0.05;**p<0.01;***p<0.001。
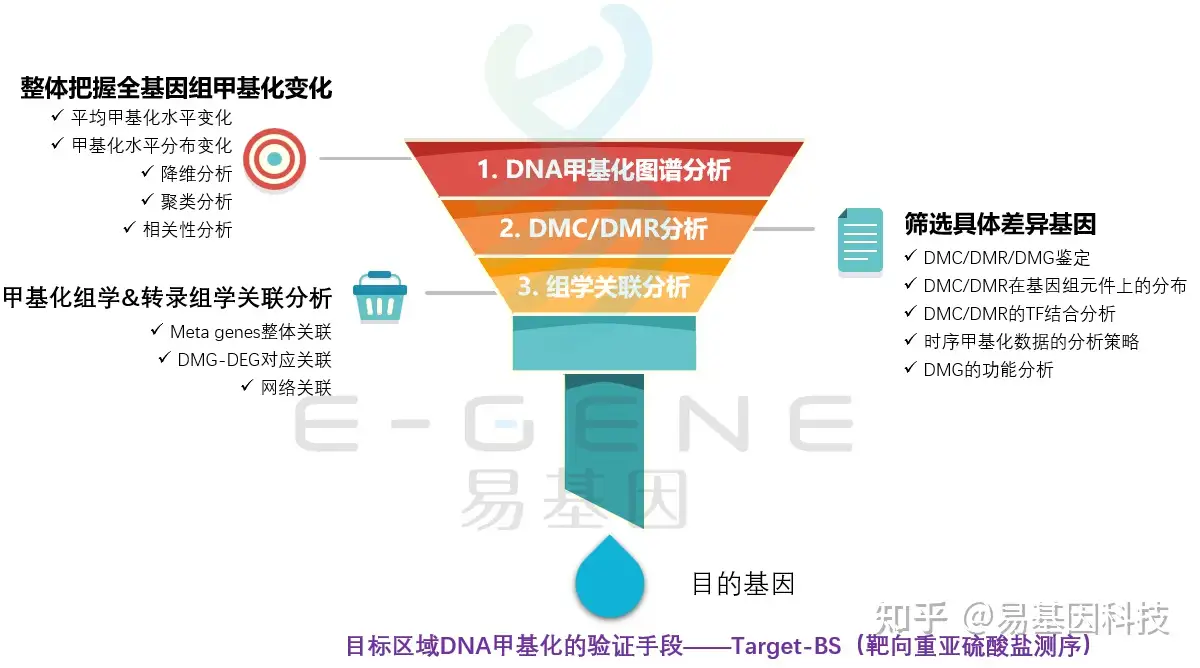
易基因:DNA甲基化研究基本思路
DNA甲基化一般遵循四个步骤:
首先,进行整体全基因组甲基化变化的分析,包括平均甲基化水平变化、甲基化水平分布变化、降维分析、聚类分析、相关性分析等。
其次,进行甲基化差异水平分析,筛选具体差异基因,包括DMC/DMR/DMG鉴定、DMC/DMR在基因组元件上的分布、DMC/DMR的TF结合分析、时序甲基化数据的分析策略、DMG的功能分析等。
再次,将甲基化组学&转录组学关联分析,包括Meta genes整体关联、DMG-DEG对应关联、网络关联等。
最后,对筛选出的目标区域DNA甲基化进行验证,通常采用靶基因重亚硫酸盐测序(Target-BS)。
关于易基因全基因组重亚硫酸盐测序(WGBS)
全基因组重亚硫酸盐甲基化测序(WGBS)可以在全基因组范围内精确的检测所有单个胞嘧啶碱基(C碱基)的甲基化水平,是DNA甲基化研究的金标准。WGBS能为基因组DNA甲基化时空特异性修饰的研究提供重要技术支持,能广泛应用在个体发育、衰老和疾病等生命过程的机制研究中,也是各物种甲基化图谱研究的首选方法。
易基因全基因组甲基化测序技术通过T4-DNA连接酶,在超声波打断基因组DNA片段的两端连接接头序列,连接产物通过重亚硫酸盐处理将未甲基化修饰的胞嘧啶C转变为尿嘧啶U,进而通过接头序列介导的 PCR 技术将尿嘧啶U转变为胸腺嘧啶T。
应用方向:
WGBS广泛用于各种物种,要求全基因组扫描(不错过关键位点)
- 全基因组甲基化图谱课题
- 标志物筛选课题
- 小规模研究课题
技术优势:
- 应用范围广:适用于所有参考基因组已知物种的甲基化研究;
- 全基因组覆盖:最大限度地获取完整的全基因组甲基化信息,精确绘制甲基化图谱;
- 单碱基分辨率:可精确分析每一个C碱基的甲基化状态。

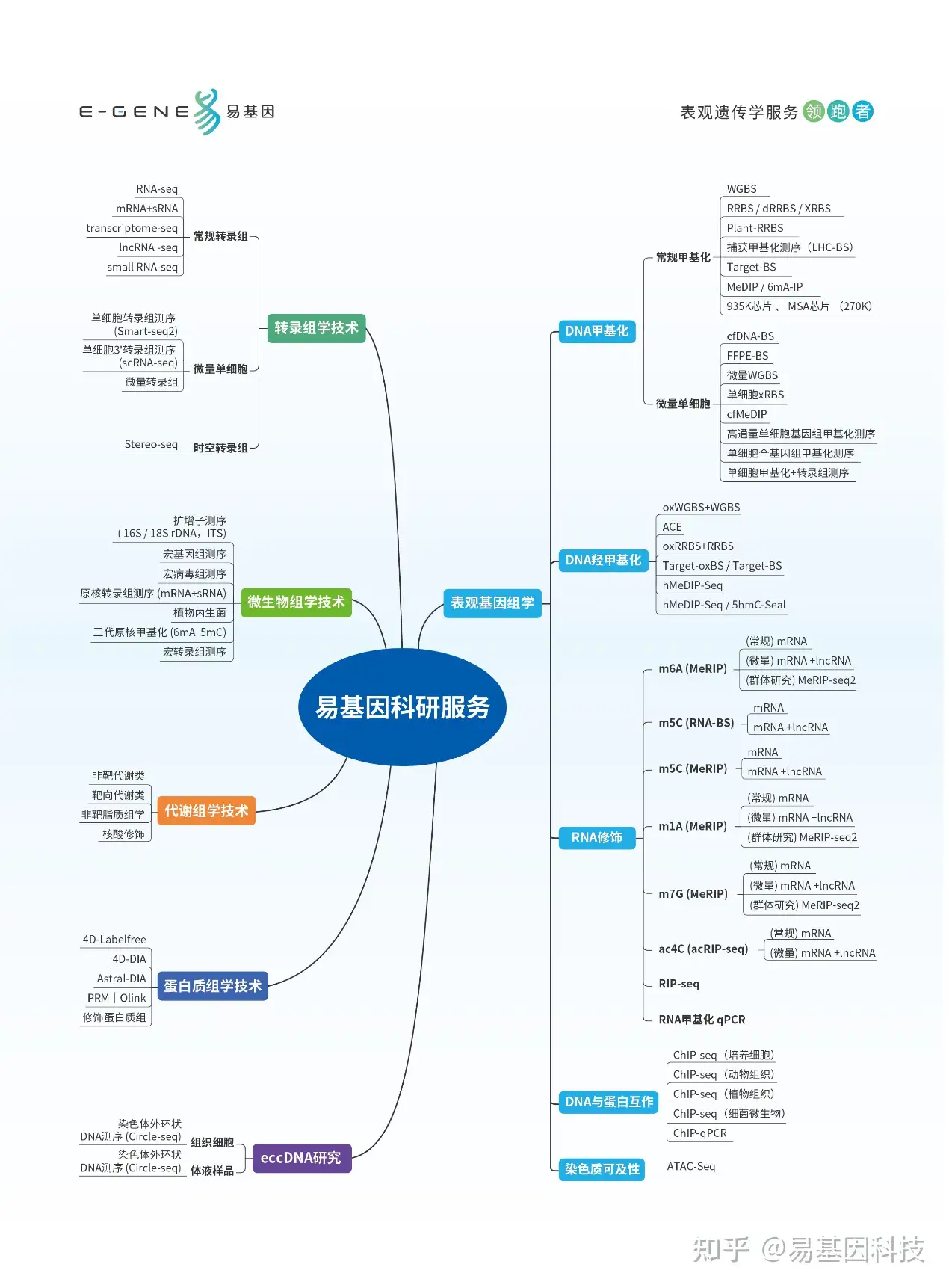
易基因提供全面的表观基因组学(DNA甲基化、DNA羟甲基化)和表观转录组学(m6A、m5C、m1A、m7G、ac4C)、染色质结构与功能组学技术方案(ChIP-seq、ATAC-seq),详询易基因:0755-28317900。
参考文献:
Feng Y, Zhang Y, Wu J, Qiao M, Zhou J, Xu Z, Li Z, Sun H, Peng X, Mei S. Comprehensive Analysis of Methylome and Transcriptome to Identify Potential Genes Regulating Porcine Testis Development. Int J Mol Sci. 2024 Aug 22;25(16) pii: ijms25169105. doi: 10.3390/ijms25169105. PubMed PMID: 39201790.
相关阅读:
项目文章 | WGBS+RNA-seq揭示松材线虫JIII阶段形成过程中的DNA甲基化差异
Cell|易基因微量DNA甲基化测序助力中国科学家成功构建胚胎干细胞嵌合体猴,登上《细胞》封面
Nature | 易基因DNA甲基化测序助力人多能干细胞向胚胎全能8细胞的人工诱导
标签:DNA,日龄,基因,基因组,甲基化,WGBS From: https://www.cnblogs.com/E-GENE/p/18471560