大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
前列腺癌(Prostate cancer,PCa)是男性中第二常见的恶性肿瘤,也是全球癌症相关死亡的第三大原因。虽然大多数原发性前列腺癌可以治愈,但转移性前列腺癌患者的5年生存率仍低至30%。大多数患者很快就会发展成转移性去势抵抗性前列腺癌(metastatic castration-resistant prostate cancer,mCRPC),最终导致死亡。因此转移性前列腺癌仍是一个主要的临床挑战,转移性病变高度异质性且难以活检,而液体活检提供了深入了解潜在生物学的机会。循环肿瘤DNA(ctDNA)在液体活检中的分析已显示出作为微创且准确的疾病监测工具潜力。在mCRPC中,ctDNA已被证明可以反映肿瘤或转移性病变的基因组特征、甲基化状态等表观遗传特征可以反映肿瘤负担和亚型。但在大规模临床队列中仍然缺乏mCRPC的全基因组cfDNA甲基化研究。最近,细胞游离甲基化DNA免疫沉淀与下一代测序(cfMeDIP-seq)的发展为分析cfDNA甲基化组提供了一种有效的方法。这种方法可以从微量cfDNA中敏感地检测ctDNA,并且与全基因组亚硫酸盐测序方法相比更具成本效益。
近日,厦门大学健康医疗大数据国家研究院吉国力教授团队和加拿大多伦多大学玛格丽特公主癌症中心何厚胜(Housheng Hansen He)教授团队等合作在Nature子刊《nature communications》发表题为“The cell-free DNA methylome captures distinctions between localized and metastatic prostate tumors”的科研成果,研究利用cfMeDIP-seq技术分析了60个原发和175个转移性前列腺癌样本的细胞游离DNA(cfDNA)甲基化组,全基因组甲基化组高效捕获了反映疾病异质性变异。进一步研究表明,cfDNA甲基化组可以以高准确率区分不同的疾病状态,突出其作为疾病监测和预后的微创策略潜力。

标题:The cell-free DNA methylome captures distinctions between localized and metastatic prostate tumors(cfDNA甲基化组捕获区分原发和转移性前列腺癌)
期刊:nature communications
影响因子: IF 14.7 / 1区
技术平台:cfMeDIP-seq(易基因优势技术)
作者利用高度敏感的富集型测序技术cfMeDIP-seq,分别对4个研究队列中60名原发前列腺癌患者和175名转移性前列腺癌患者的cfDNA甲基化组进行了分析。分析结果揭示cfDNA甲基化组可以捕获肿瘤变化。在转移性样本中观察到全基因组高甲基化,同时着丝粒周围区域出现低甲基化。糖皮质激素受体(glucocorticoid receptor,GR)基因NR3C1的启动子区域高甲基化与免疫信号减少有关。cfDNA甲基化组反映了临床结果,并且可以以98.9%高精度准确率预测区分不同的疾病类型(转移性和原发样本),表现出了强大的疾病监测和预测的微创策略潜力。最后,研究从数据中预测拷贝数变化的能力,为有限的生物样本提供了联合遗传和表观遗传分析的机会。
研究设计
样本收集:4个研究队列(原发性肿瘤患者来自CPC-Gene,转移性前列腺癌来自VPC、Barrier、WCDT)。分析方法:使用高灵敏度的基于富集测序技术cfMeDIP-seq,分析了60个局部前列腺癌和175个转移性前列腺癌患者的血浆DNA甲基化组,将所有血浆样本中的甲基化DNA片段沉淀并进行配对末端DNA测序。
结果图形
(1)对原发和转移性前列腺癌(PCa)的血浆游离DNA甲基化组进行全基因组范围分析。
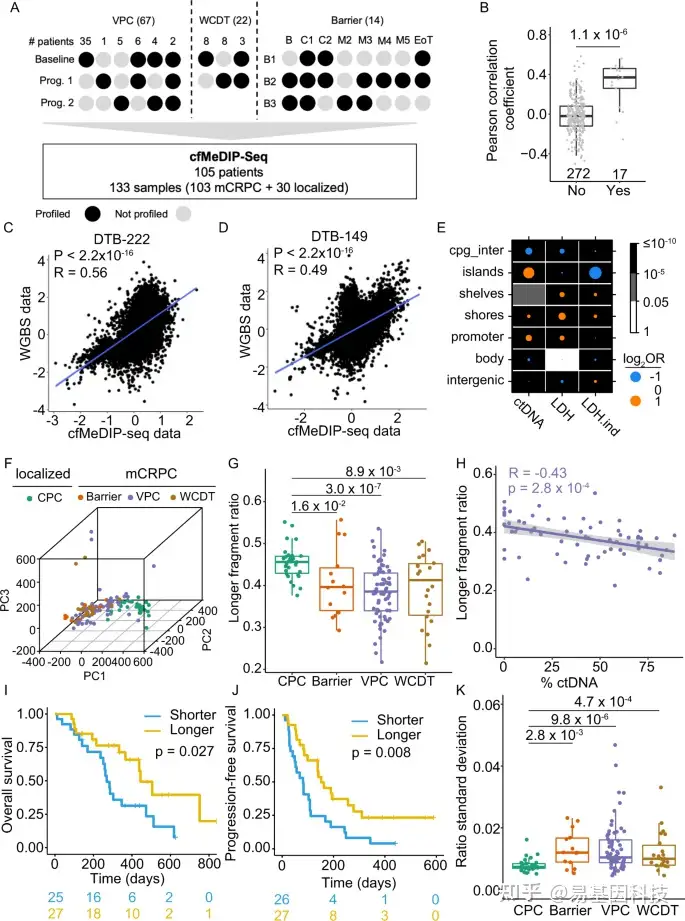
图1:cfMeDIP数据捕获原发与转移性去势抵抗性前列腺癌(mCRPC)患者血浆样本中cfDNA甲基化和片段化变化。
- 三个mCRPC队列的抽样示意图。括号中数字表示给定队列中的样本总数。
B-D. WCDT队列,组织WGBS和cfMeDIP数据的样本配对相关性,比较匹配样本与非匹配样本的差异。
E. %ctDNA、LDH以及与不依赖%ctDNA的LDH解释的不同基因组位点富集情况。
F. 前10,000个变异区间的主成分分析(PCA)对四个队列样本进行三维表示。
G. 各队列中较长cfDNA片段比例。
H. VPC队列中mCRPC样本的较长片段比例与ctDNA比例(%ctDNA)的皮尔逊相关性。
I-J. 片段大小与总生存期(OS)和无进展生存期(PFS)的相关性。
K. 样本内片段比例标准差分布。
(2)cfDNA甲基化分析显示转移性前列腺癌样本中广泛高甲基化和着丝粒周围区域低甲基化
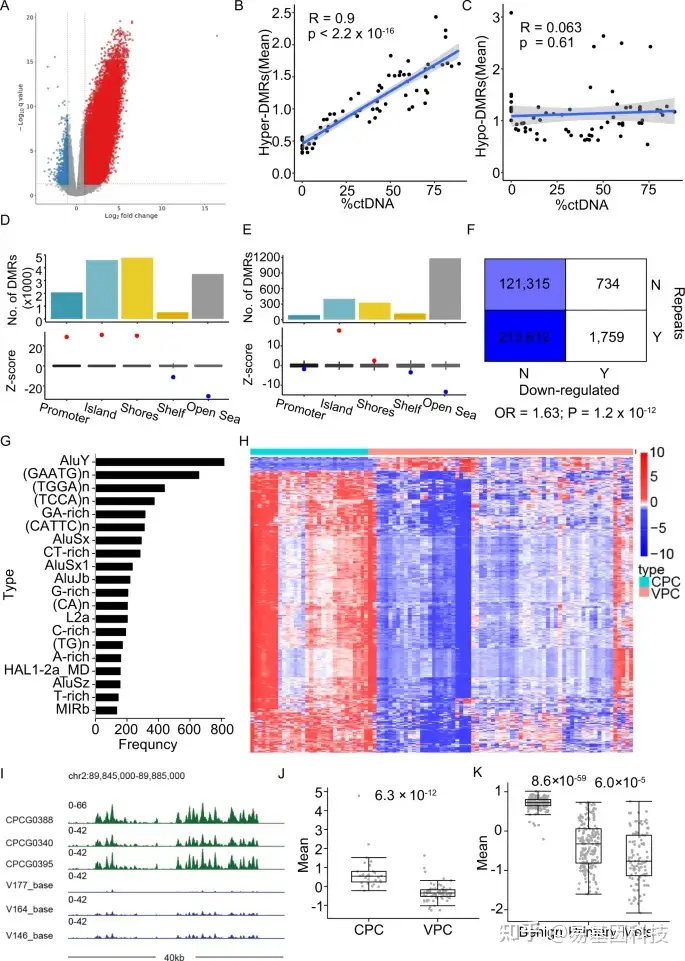
图2:cfMeDIP揭示转移样本中广泛的高甲基化和着丝粒区域低甲基化。
- 通过比较转移性和原发前列腺癌,鉴定差异性甲基化区域(DMRs)火山图。分别鉴定出2493个低甲基化和19048个高甲基化DMRs。
B-C. 高甲基化(Hyper)和低甲基化(Hypo)DMRs平均甲基化水平与%ctDNA之间的皮尔逊相关性。
D-E. 高甲基化和低甲基化DMRs的基因组分布。
F. 与低甲基化峰重叠的重复类型分布的列联表。
G. 与下调峰重叠的重复类型频率。
H. 位于着丝粒周围1 Mb区域内的差异性甲基化峰。
- 染色体2中着丝粒附近区域信号分布示例。
J-K. 分别展示了cfMeDIP-seq和WGBS数据中H中显示的差异峰的平均甲基化水平。
(3)NR3C1启动子甲基化水平与不同疾病结局相关
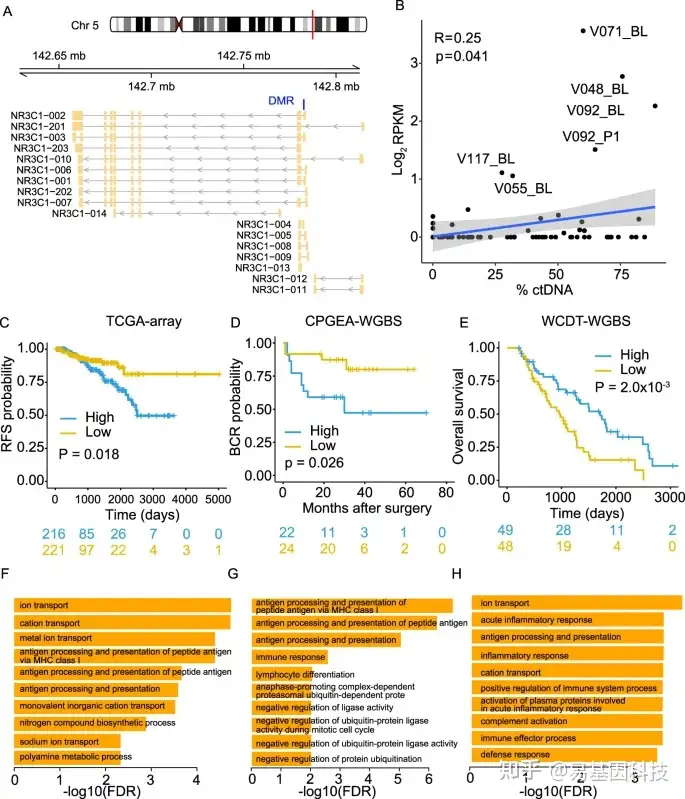
图3:GR基因甲基化差异与mCRPC改变相关。
- GR基因(NR2C1)及其不同异构体示意图。蓝色条表示已鉴定的异常DMR。
- VPC队列中GR位点甲基化水平与%ctDNA之间的皮尔逊相关性。
C-E. 癌症基因组图谱(TCGA)、中国前列腺癌基因组和表观基因组图谱(CPGEA)和WCDT队列中GR位点甲基化与疾病结果之间的相关性。
F-H. TCGA(F)、CPGEA(G)和WCDT(H)队列中高GR-DMR甲基化组中下调基因的生物学过程(BP)的基因本体(GO)富集分析。
(4)cfDNA甲基化组可高精度区分转移性和原发性样本
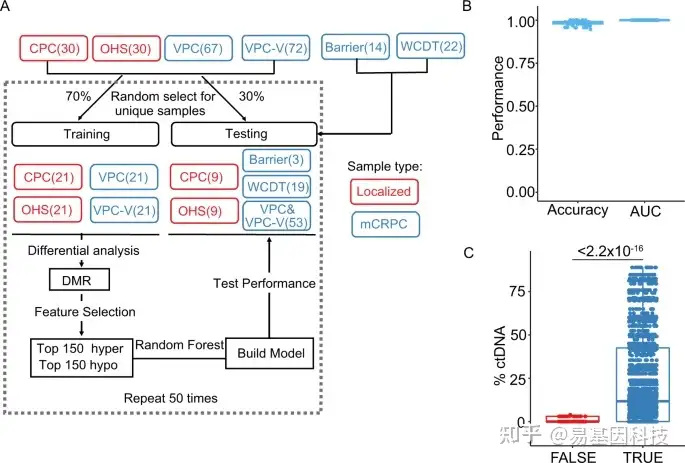
图4:甲基化谱以高准确率区分原发和转移性样本。
- 使用甲基化配置文件构建随机森林预测因子工作流程。
- 测试数据集上,基于甲基化谱预测因子的预测准确性和接收者操作特征曲线下面积(AUROC)。VPC-V验证样本来自VPC队列,OHS为安大略省健康研究队列,DMR为差异甲基化区域。
- 对于转移样本,正确或错误分类的样本中%ctDNA分布。
(5)游离DNA甲基化组可以预测大规模遗传变异
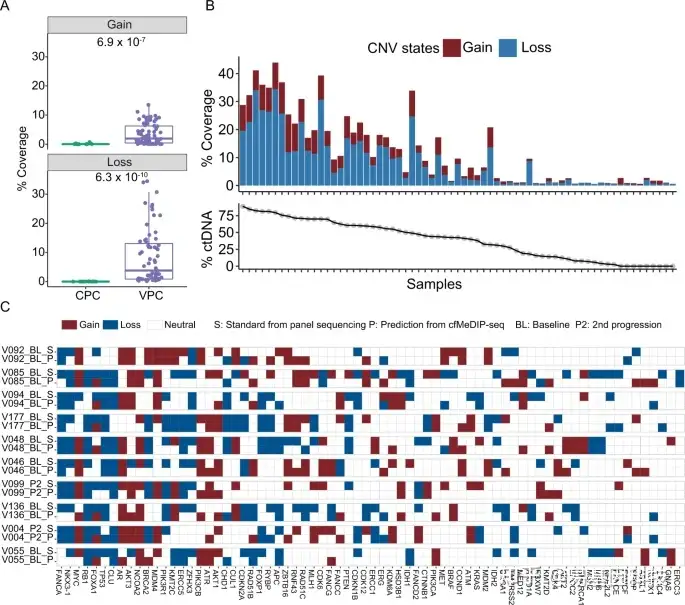
图5:使用cfDNA甲基化组预测样本拷贝数变异(CNA)。
- 预测来自CPC队列的原发样本和VPC队列的mCRPC样本的CNA覆盖度。
- VPC队列中mCRPC样本预测的CNA覆盖度与%ctDNA之间的相关性。
- 比较先前panel测序获得的标准状态和cfMeDIP-seq数据预测结果的基因CNA,针对CNA覆盖度最高的前10个样本。
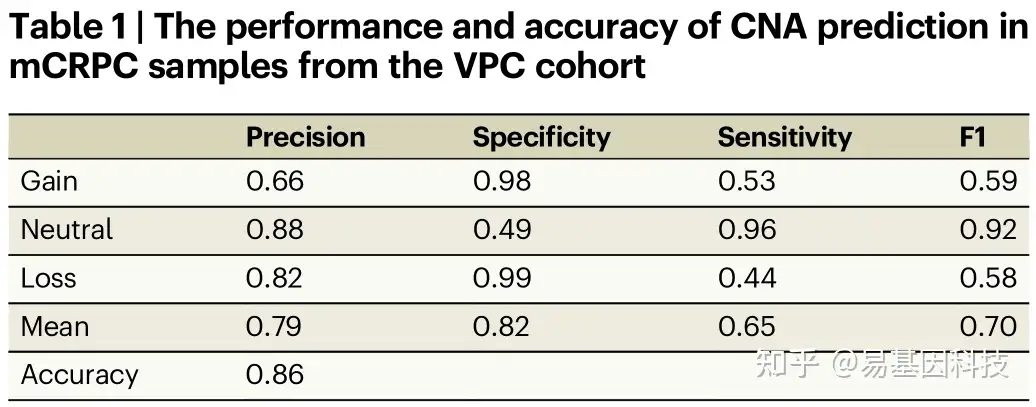
表1:VPC队列中mCRPC样本中CNA预测的性能和准确性
关于易基因甲基化游离DNA免疫共沉淀测序(cfMeDIP-seq)
由于血浆cfDNA的丰度低且碎片化程度高,片段大小常在150bp左右,限制了大部分血浆甲基化特异性PCR检测方法的应用,相关检测研究也存在较大的挑战。虽然有科学家尝试利用全基因组甲基化测序(WGBS)进行甲基化检测,但WGBS技术在亚硫酸盐转化过程中存在DNA容易降解、检测成本高等特点。
为突破现有检测技术限制,易基因推出灵敏、可分离、检测和分析低水平甲基化DNA的甲基化游离DNA免疫共沉淀测序(cfMeDIP-seq)方法。该方法将免疫共沉淀技术与高通量测序结合,通过对cfDNA中的甲基化DNA片段(富含CpG区域)进行特异性富集,提高检测效率。
技术优势:
- 检测范围广:全基因组范围鉴定甲基化修饰区域
- 起始量低:起始量低至5ng
- 富集倍数高:CpG富集倍数可达到上样前的2倍
- 针对性强:针对性检测基因组内具有甲基化修饰的区域
- 成本低:大大降低了测序数据量,测序成本低
应用场景:
- 癌前病变的癌变预警标志物检测
- 肿瘤早期筛查标志物检测
- 肿瘤预后标志物检测
- 药物疗效预测标志物检测
易基因提供全面的表观基因组学(DNA甲基化、DNA羟甲基化)和表观转录组学(m6A、m5C、m1A、m7G、ac4C)、染色质结构与功能组学技术方案(ChIP-seq、ATAC-seq),详询易基因:0755-28317900。
参考文献:
Chen S, et al. The cell-free DNA methylome captures distinctions between localized and metastatic prostate tumors. Nat Commun. 2022 Oct 29;13(1):6467. pii: 10.1038/s41467-022-34012-2. doi: 10.1038/s41467-022-34012-2. PubMed PMID: 36309516.
相关阅读:
技术推介 | 微量cfDNA简化基因组甲基化测序(cfDNA-RBS)
技术推介|羟甲基化DNA免疫共沉淀测序(hMeDIP-Seq)
标签:DNA,seq,Commun,样本,测序,cfMeDIP,甲基化,转移性,cfDNA From: https://www.cnblogs.com/E-GENE/p/18451463