大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
梨(Pyrus ssp.,蔷薇科杏仁核亚科)是世界上最重要的温带水果作物之一。与野生梨相比,栽培梨的果实在许多形态特征上表现出显著变化,包括果实大小、含糖量和核细胞含量。野生梨和栽培梨之间的比较分析可以深入了解关键表型变化的演变。DNA甲基化是一种重要的可遗传表观遗传学标记,可以改变基因组区域可及性,抑制或激活基因表达,最终导致表型变化。然而,表观等位基因在多年生果树驯化中的重要性尚待发现。
在植物中,胞嘧啶DNA甲基化发生在三种不同的背景中:CG、CHG和CHH(H=A、T或G)。DNA甲基化水平受到四种去甲基化酶的调节,包括抑制沉默1/Demeter样1(ROS1/DML1)、Demeter(DME)、Demeter样2(DML2)和Demeter样3(DML3)。
2024年4月5日,南京农业大学吴俊教授团队对41份亚洲梨(Pyruspyrifolia)样本(包括野生品种、地方品种和改良品种)进行了单碱基分辨率的甲基化分析。通过比较甲基化分析,在梨的驯化和改良过程中全基因组DNA甲基化水平增加,与编码DNA去甲基化酶的基因表达水平降低相关。研究还鉴定出梨的驯化和改良过程中的差异化甲基化区域(DMRs)。高甲基化DMRs基因与植物衰老和果实成熟显著相关。研究为揭示多年生果树驯化和改良过程中表观遗传调控重要性状的作用机制提供参考,为指导梨重要性状遗传改良提供了理论依据。相关研究成果以"Increased DNA methylation contributes to the early ripening of pear fruits during domestication and improvement"为题发表在《Genome Biology》(IF 12.3 / 1区)期刊上。

研究摘要:
本研究利用全基因组重亚硫酸盐测序技术研究了41份梨样本进行了梨驯化和改良过程中的DNA甲基化变化。与水稻驯化期间的显著减少相反,研究结果表明梨驯化和改良过程中整体DNA甲基化水平增加。梨的DNA甲基化特定增加与人类选择导致的Demeter-like1(DML1,编码DNA去甲基化酶)的下调显著相关。研究总共鉴定出5591个差异甲基化区域(DMRs)。在梨的驯化和改良过程中,CG和CHG中的甲基化经历共同进化。DMRs比选择性扫描区域具有更高的遗传多样性,特别是在内含子中。大约97%的DMRs与任何SNPs无关,这些DMRs与淀粉和蔗糖代谢以及苯丙烷生物合成相关。此外,研究还进行DNA甲基化与基因表达的相关性分析。分析结果表明,高甲基化DMRs基因与果实成熟显著相关,进一步验证与高甲基化DMRs相关基因CAMTA2的功能,并验证在番茄和梨愈伤组织中CAMTA2过表达抑制了果实成熟。
总之,本研究揭示了多年生梨树驯化和改良过程中DNA甲基化的特定模式,并表明DNA甲基化增加在梨果早熟中发挥着重要作用。
研究方法:

结果图形
(1)人对Demeter样1(DML1)选择可能与梨驯化和改良过程中DNA甲基化增加有关
为分析梨驯化和改良过程中发生的全基因组DNA甲基化变化,研究人员构建了全基因组重亚硫酸盐测序(WGBS)文库,包含41个代表性P. pyrifolia品种的两个生物学重复,其中包括14个野生品种、12个地方品种和15个改良梨品种。平均BS转化率为99.44%。以P. pyrifolia “Cuiguan”基因组为参考基因组。
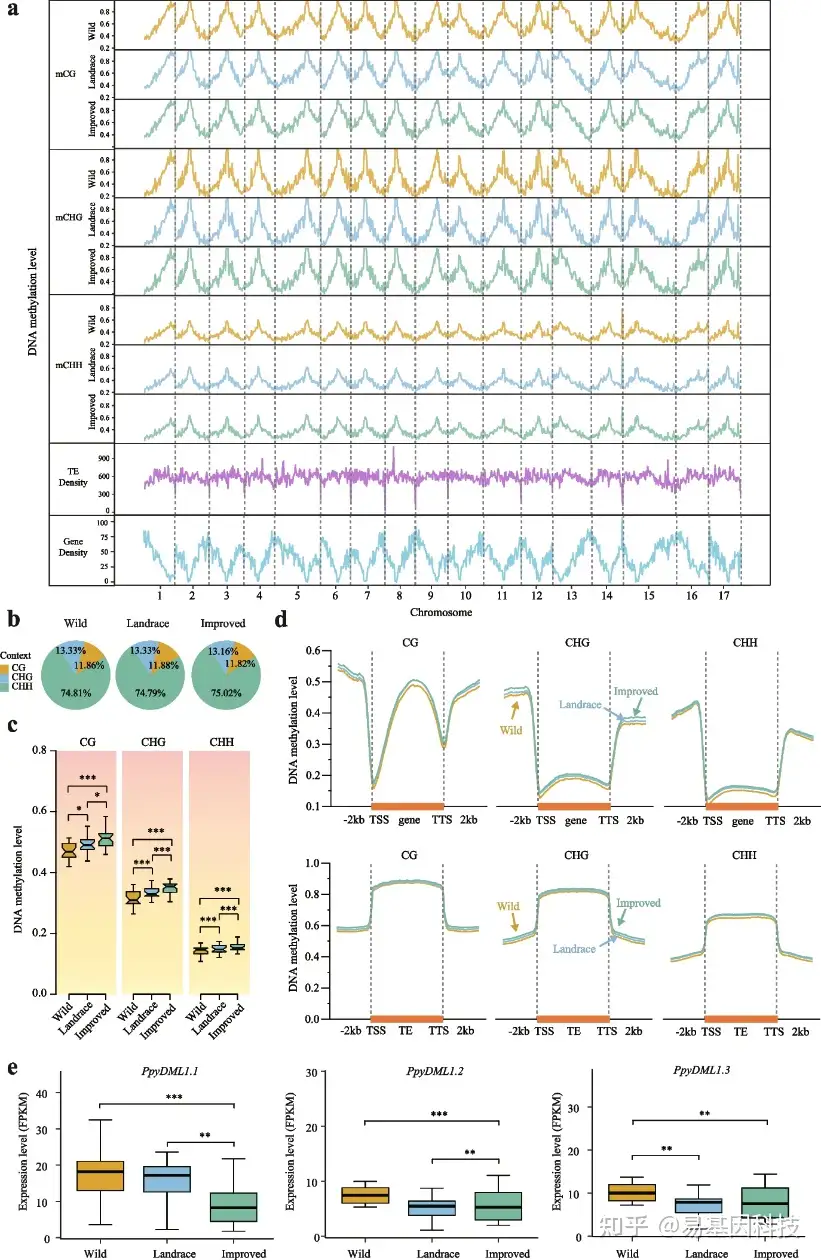
图1:梨基因组中DNA胞嘧啶甲基化水平的分布模式
- 17条梨染色体上的CG、CHG和CHH下DNA甲基化水平的密度分布、基因密度和TE密度。
- 三个梨品种(野生品种、地方品种和改良品种)中CG、CHG和CHH甲基化胞嘧啶(mC)平均比率。
- 野生品种、地方品种和改良品种中CG、CHG和CHH甲基化水平进行比较(*P < 0.05; **P < 0.01; ***P < 0.001,双尾配对学生t检验)。
- 基因和转座元件(TEs)的上下游±2kb区域、和genebody区域的DNA甲基化水平分布。
- 在野生品种、地方品种和改良品种中,PpyDML1.1、PpyDML1.2和PpyDML1.3的相对基因表达水平比较(FPKM)。PpyDML1.1、PpyDML1.2和PpyDML1.3在梨驯化和改良过程中表达水平持续下降(*P < 0.05; **P < 0.01; ***P < 0.001,使用cuffdiff进行差异表达分析)。
(2)梨驯化和改良过程中CG和CHG甲基化的共同进化
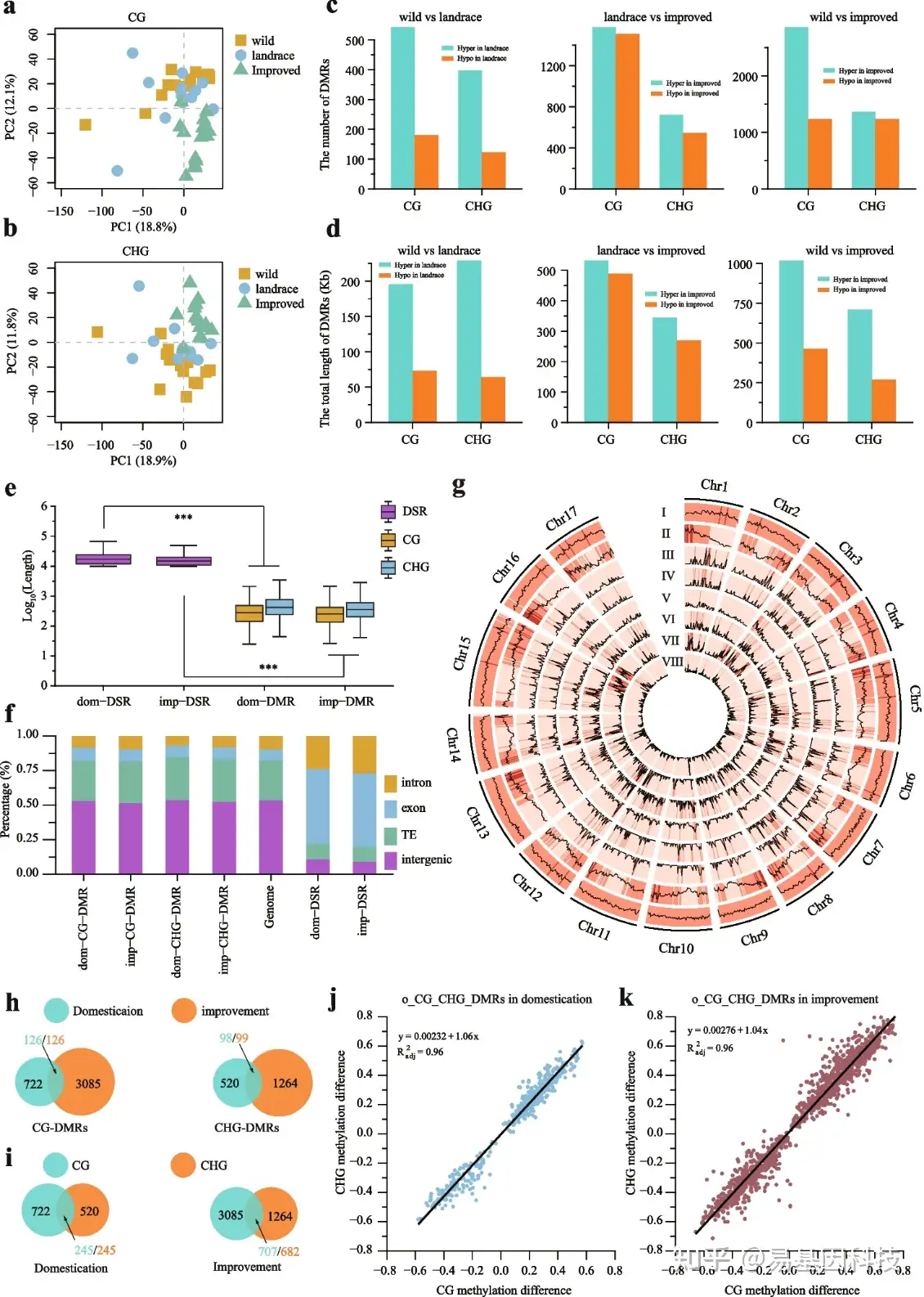
图2:野生品种梨 vs地方品种梨以及地方品种梨vs改良品种梨的差异甲基化区域(DMRs)比较
- CG中的DNA甲基化水平主成分分析(PCA)图。黄色方块:野生梨品种,蓝色点:地方品种,绿色三角形:改良品种。
- CHG中的DNA甲基化水平主成分分析(PCA)图。
- 比较野生与地方品种、地方品种与改良品种以及野生与改良品种的高甲基化/低甲基化DMRs(hyper/hypo-DMRs)数量。
- c中三种比较中高甲基化/低甲基化DMRs的总长度。
- 比较野生与地方品种和地方品种与改良品种比较中,DNA序列选择区域(DSRs)长度与CG和CHG背景DMRs的长度(*P < 0.05; **P < 0.01; ***P < 0.001,双尾配对学生t检验)。
- 显示DSRs和DMRs的基因组组成,包括转座元件(TEs)、内含子、外显子和基因间区域。
- 对17条梨染色体上野生与地方品种群间DMRs分布图。从外圈到内圈的数据分别代表TE密度(I)、基因密度(II)、驯化过程中CG-DMR密度(III)、驯化过程中CHG-DMR密度(IV)、改良过程中CG-DMR密度(V)、改良过程中CHG-DMR密度(VI)、驯化过程中的DSR(VII)和改良过程中的DSR(VIII)。
- 对野生与地方品种(CG和CHG背景)以及地方种与改良品种之间的DMRs重叠分析。
- 对野生与地方品种(驯化过程)和地方品种与改良品种(改良过程)比较中两种甲基化背景的DMRs重叠。
- 在梨驯化过程中,o_CG_CHG_DMRs(CG-DMRs与CHG-DMRs重叠区域)的CG和CHG甲基化水平相关性分析。
- 在梨改良过程中,对o_CG_CHG_DMRs的CG和CHG甲基化水平进行相关性分析。
(3)遗传多样性变化与梨驯化和改良过程中的DNA甲基化变化无关
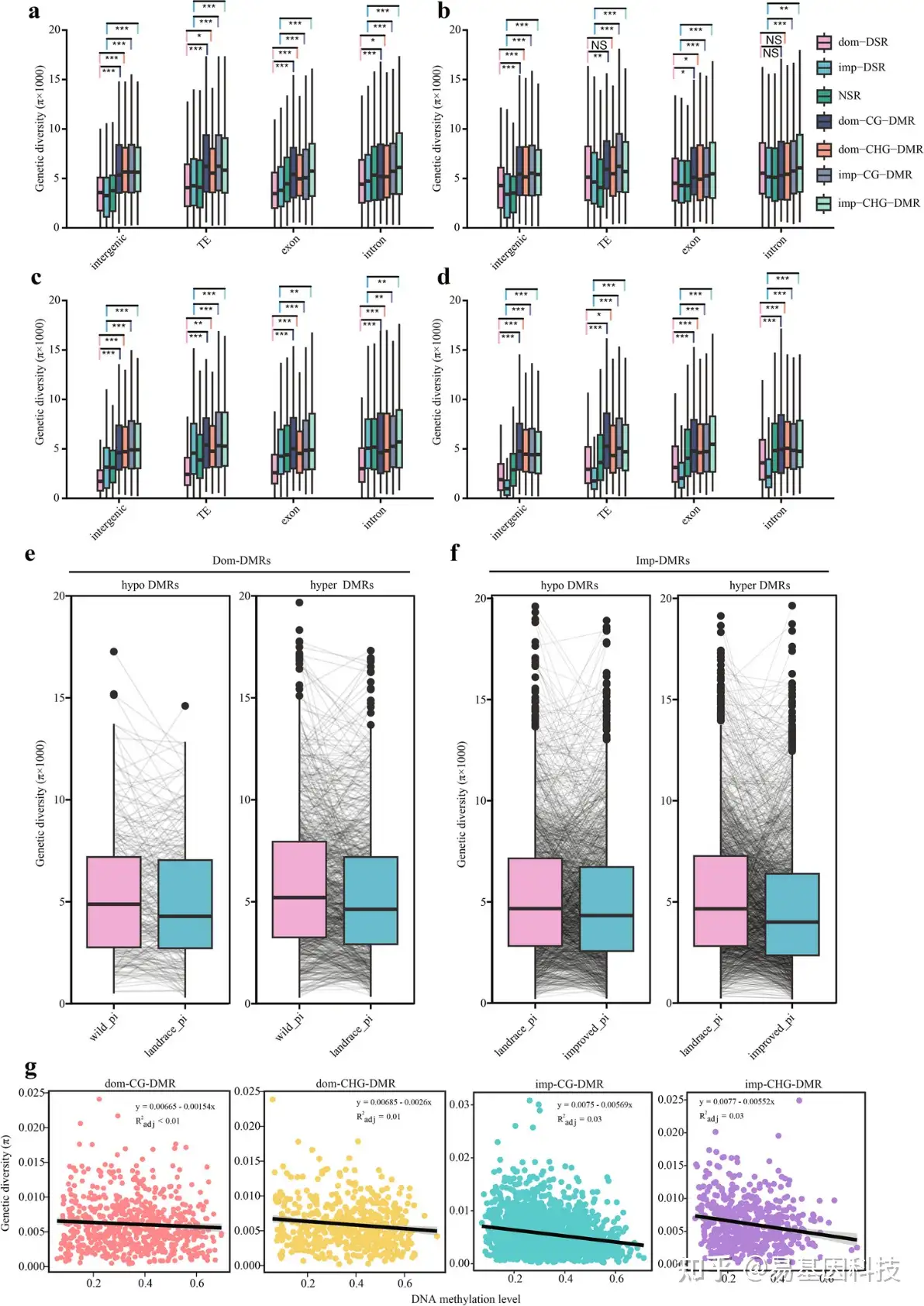
图3:梨的驯化过程中差异甲基化区域(DMRs)的遗传多样性变化:
a-d. 所有梨(a)和野生品种梨(b)、地方品种梨(c)和改良品种梨(d)不同基因组组成中DMR、DSRs和NSRs之间的遗传多样性比较(*P<0.05;**P<0.01;***P<0.001;NS,不显著;双尾配对学生t检验)。不同基因组组成包括基因间区域、TE、外显子和内含子。
e-f. 在驯化(Dom-DMRs)(e)和改良(Imp-DMRs)(f)过程中,高甲基化DMRs和低甲基化DMRs的遗传多样性变化。每一条黑线代表一个DMR。
g. dom CG DMR、dom CHG DMR、imp CG DMR和imp CHG DMRs中DNA甲基化水平与遗传多样性之间的关系。
(4)DMRs遗传基础的meQTL分析
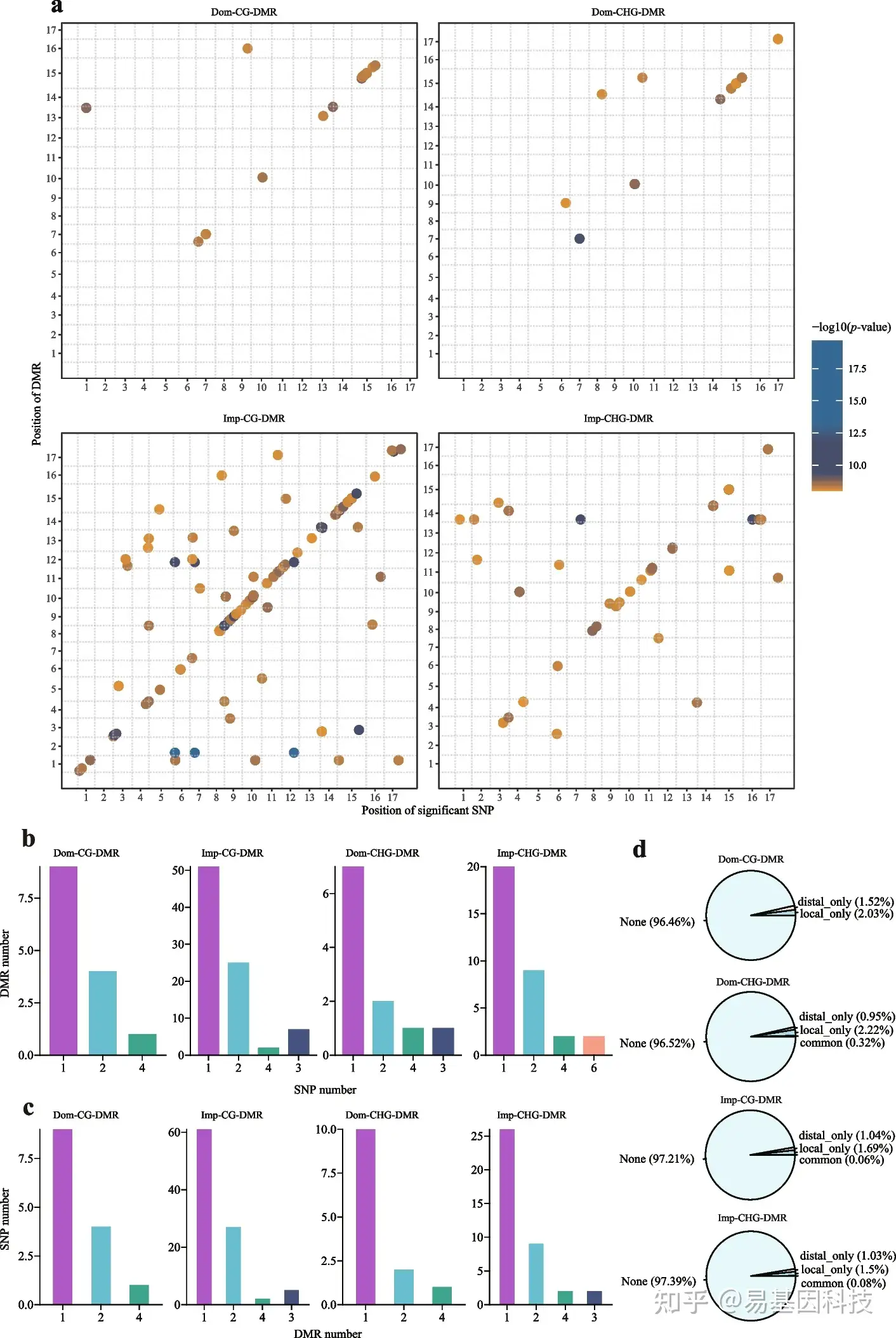
图4:梨驯化和改良过程中差异甲基化区域(DMRs)的遗传基础。
- 在梨驯化和改良过程中鉴定的meQTL的染色体位置分布。x轴表示显著SNPs的基因组位置,y轴表示SNPs相应DMR的基因组位置。点的颜色表示meQTL分析中的P值。meQTL显著阈值设定为1.78×10−9(0.01/N,N=5618948),仅绘制显著meQTL。Dom-CG-DMR代表野生和地方品种之间的CG DMR;Dom-CHG-DMR代表野生和地方品种之间的CHG DMRs;Imp-CG-DMR代表地方品种和改良品种之间的CG-DMR;Imp CHG DMR代表地方品种和改良梨品种之间的CHG DMRs。
- 每个DMR的显著SNP数量分布。
- 每个SNP的显著相关DMR的数量分布。
- 梨驯化和改良过程中CG和CHG DMRs的遗传基础。
(5)DNA甲基化对基因表达的作用
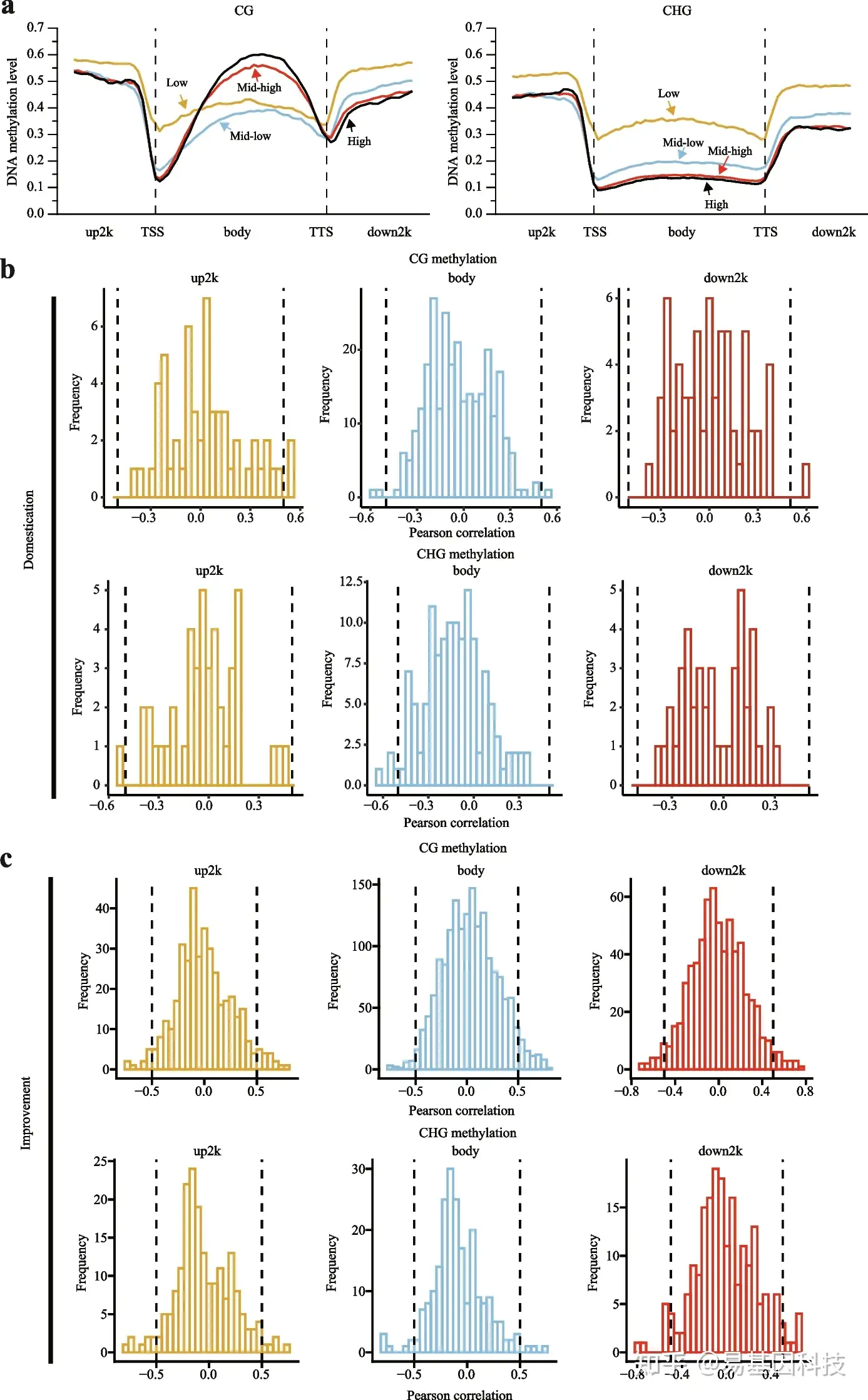
图5:梨驯化改良过程中DNA甲基化与基因表达水平的相关性研究。
- 在2kb上游、基因体和2kb下游区域的所有基因的CG和CHG甲基化水平与表达水平之间的关系。根据表达水平将基因分为四组(低、中低、中高和高)。
b-c. 梨驯化(b)和改良(c)过程中,2kb上游、基因体和2kb下游区域的基因表达水平与CG和CHG环境中DMR甲基化水平之间的Pearson相关系数分布。
(6)梨驯化和改良过程中DNA高甲基化与果实早熟有关
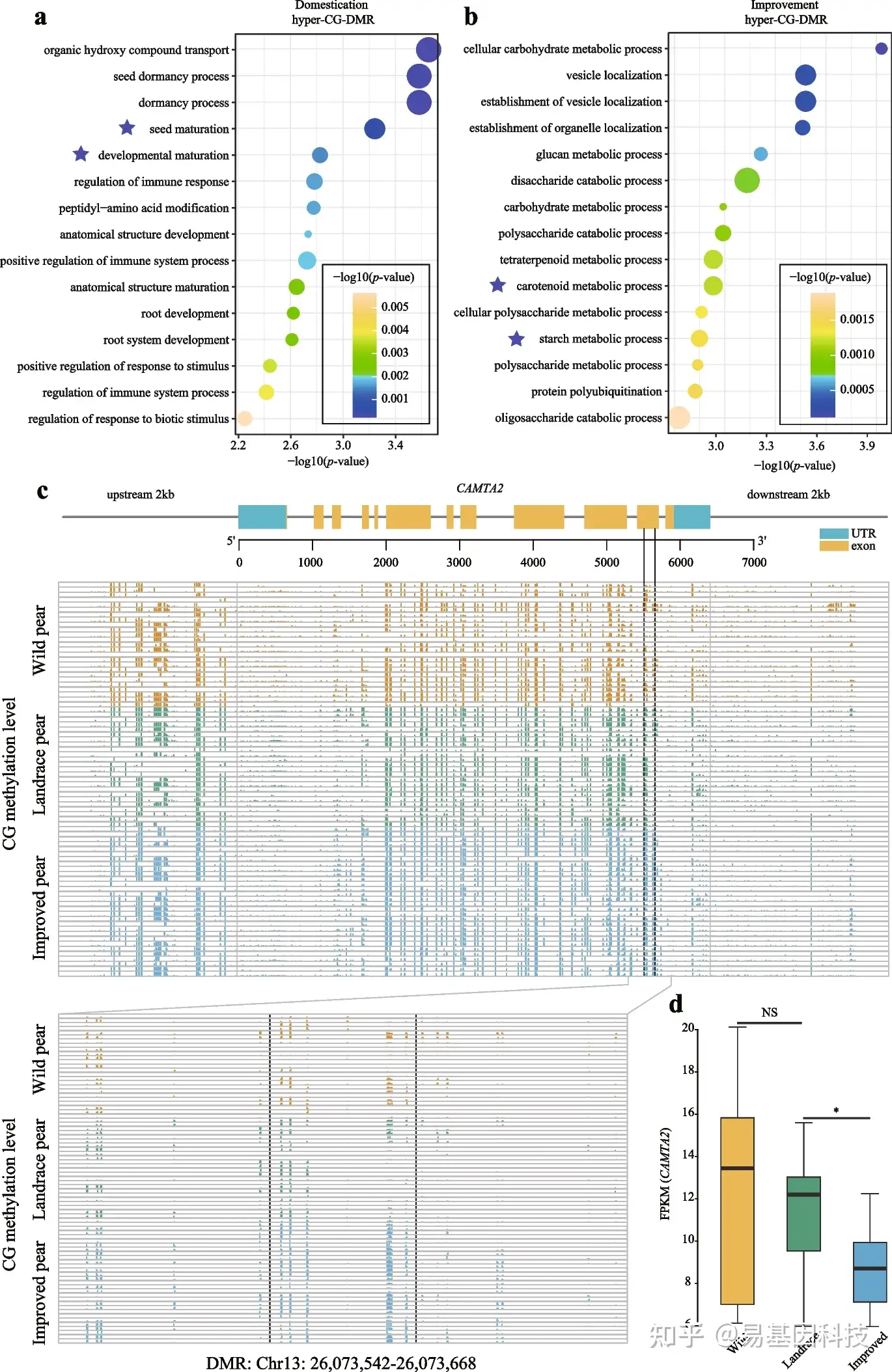
图6:梨CAMTA2基因中的一个高甲基化DMR以及它在梨驯化和改良过程中的作用。
- 梨驯化过程中高CG DMR相关基因的GO富集分析(前15个显著项)。
- 梨改良过程中高CG DMR相关基因的GO富集分析(前15个显著项)。蓝色星号表示与衰老相关的GO术语。
- CAMTA2基因结构如图顶部所示。外显子用黄色阴影框表示,内含子用黑线表示,蓝色阴影框表示5′和3′UTR。下图显示了野生、地方品种和改良梨品种中位于CAMTA2基因中的DMR(Chr13:26073542–26073668)的CG甲基化水平。上图表示整个基因(2kb上游、基因体和2kb下游区域),下图扩大显示第11外显子中的DMR。
- 野生品种(黄框)、地方品种(绿框)和改良品种(蓝框)梨中CAMTA2表达水平(FPKM)的比较(*P<0.05;**P<0.01;***P<0.001;NS,不显著;双尾配对Student t检验)
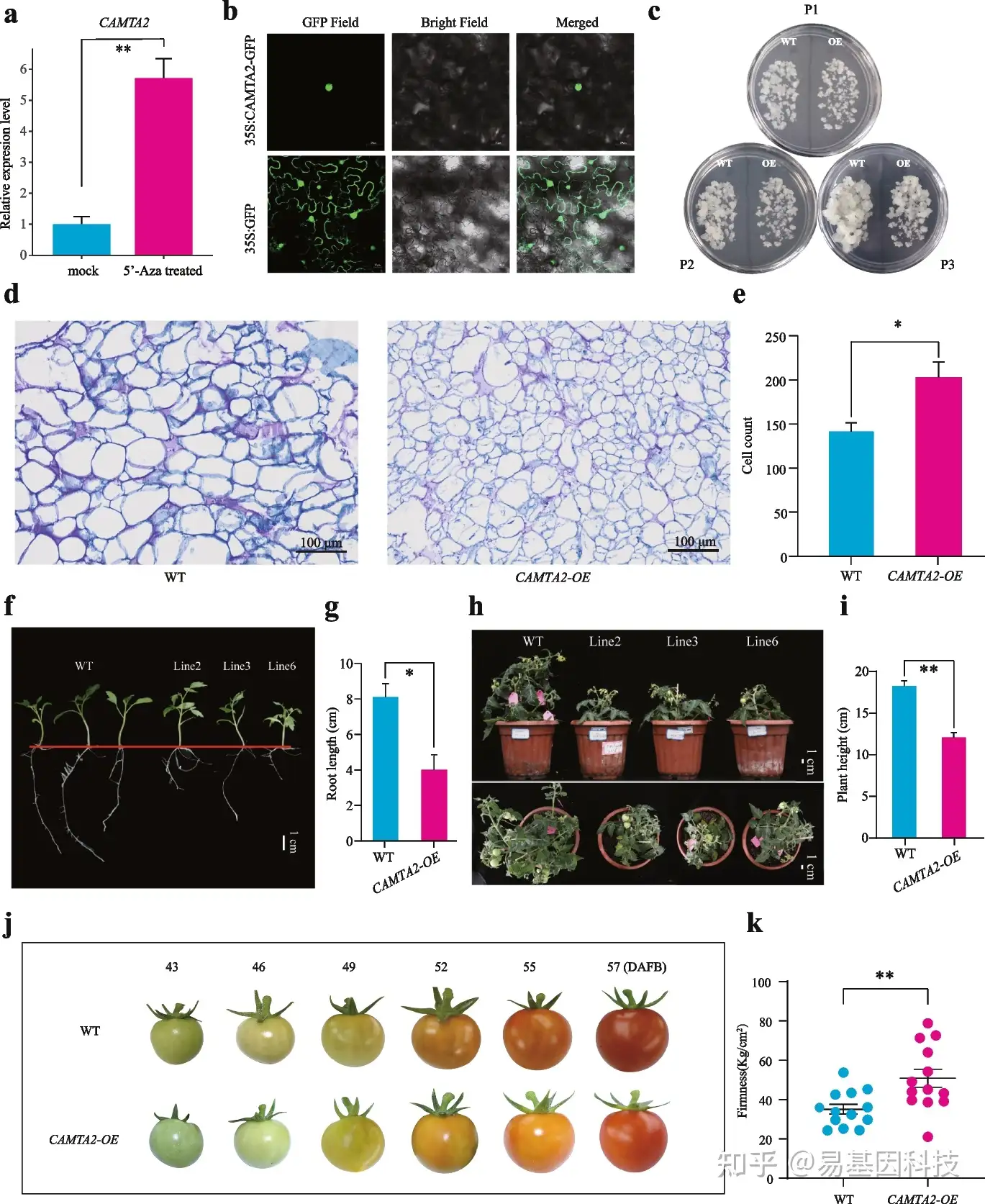
图7:CAMTA2在转基因梨愈伤组织和转基因番茄植株中的作用。
- CAMTA2基因在对照和5′-氮杂胞苷(5′-Aza)处理的梨愈伤组织中的相对表达。
- CAMTA2-GFP融合蛋白定位于农业过滤的本氏烟草(Nicotiana benthamiana)叶片细胞的细胞核。
- WT和CAMTA2过表达(OE)梨愈伤组织的生长。P1=继代培养后立即,P2=继代培养后14天,P3=继代培养后24天。
d-e. 用甲苯胺蓝染色的梨愈伤组织的横截面。图像显示了过表达CAMTA2(e)的WT(d)和转基因梨愈伤组织的横截面的相同视野中的细胞数量。比例尺=100μm。
f. T1代转基因幼苗在移植前的生长状况。比例尺=1cm。
g. WT和T1代CAMTA2-OE幼苗根长的统计分析。
h. WT和CAMTA2-OE转基因番茄植株的表型。比例尺=1cm。
- 野生型和转基因番茄植株株高的统计分析。
j. WT CAMTA2-OE转基因番茄果实在盛开后43、46、49、52、55和57天(DAFB)的代表性表型。
k.在红期收获的WT和CAMTA2-OE转基因番茄果实硬度统计分析。
研究结论
本研究结果表明,在梨的驯化和改良过程中DNA甲基化的整体增加。这种DNA甲基化增加与 DML1表达下调显著相关。在梨的驯化和改良过程中,分别鉴定出1242个和4349个DMR。高DMRs附近基因与植物衰老和果实成熟显著相关。研究还验证了高DMRs相关基因CAMTA2的功能,即CAMTA2过表达抑制果实成熟。简言之,本研究报告了在梨的驯化和改良过程中DNA甲基化的增加模式,并表明增加的DNA甲基化在调节梨果实成熟期中起着至关重要的作用。
关于易基因全基因组重亚硫酸盐测序(WGBS)
全基因组重亚硫酸盐甲基化测序(WGBS)可以在全基因组范围内精确的检测所有单个胞嘧啶碱基(C碱基)的甲基化水平,是DNA甲基化研究的金标准。WGBS能为基因组DNA甲基化时空特异性修饰的研究提供重要技术支持,能广泛应用在个体发育、衰老和疾病等生命过程的机制研究中,也是各物种甲基化图谱研究的首选方法。
易基因全基因组甲基化测序技术通过T4-DNA连接酶,在超声波打断基因组DNA片段的两端连接接头序列,连接产物通过重亚硫酸盐处理将未甲基化修饰的胞嘧啶C转变为尿嘧啶U,进而通过接头序列介导的 PCR 技术将尿嘧啶U转变为胸腺嘧啶T。
应用方向:
WGBS广泛用于各种物种,要求全基因组扫描(不错过关键位点)
- 全基因组甲基化图谱课题
- 标志物筛选课题
- 小规模研究课题
技术优势:
- 应用范围广:适用于所有参考基因组已知物种的甲基化研究;
- 全基因组覆盖:最大限度地获取完整的全基因组甲基化信息,精确绘制甲基化图谱;
- 单碱基分辨率:可精确分析每一个C碱基的甲基化状态。

易基因提供全面的表观基因组学(DNA甲基化、DNA羟甲基化)和表观转录组学(m6A、m5C、m1A、m7G)、染色质结构与功能组学技术方案(ChIP-seq、ATAC-seq),详询易基因:0755-28317900.

参考文献:
Song B, Yu J, Li X, Li J, Fan J, Liu H, Wei W, Zhang L, Gu K, Liu D, Zhao K, Wu J. Increased DNA methylation contributes to the early ripening of pear fruits during domestication and improvement. Genome Biol. 2024 Apr 5;25(1):87. pii: 10.1186/s13059-024-03220-y. doi: 10.1186/s13059-024-03220-y. PubMed PMID: 38581061.
相关阅读:
项目文章 | WGBS+RNA-seq揭示松材线虫JIII阶段形成过程中的DNA甲基化差异
项目文章 | WGBS+RNA-seq揭示黄瓜作物的“源-库”关系受DNA甲基化调控
项目文章 | WGBS等揭示SOX30甲基化在非梗阻性无精症中的表观遗传调控机制
项目文章|WGBS+RNA-seq揭示PM2.5引起男性生殖障碍的DNA甲基化调控机制
标签:DMRs,CHG,CG,甲基化,驯化,DNA,WGBS From: https://www.cnblogs.com/E-GENE/p/18193815