大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
DNA甲基化数据可以生成非常精确的年龄预测器,但关于这一关键表观遗传生物标志物在生命周期中的动态变化知之甚少。关于衰老不连续方面的研究仍处于起步阶段,关键的分子过程如表观基因组调控过程还有待研究。
莱布尼茨老龄化研究所-弗里茨·利普曼研究所(FLI)Hoffmann实验室的研究人员分析了雄性小鼠在衰老过程中五个不同时间点的结肠组织甲基化轨迹。研究结果表明,在生命的特定阶段存在突然的高甲基化事件:在早至中年(3-9月龄)和中至晚年(15-24月龄)转换期间的两个表观遗传开关,将啮齿动物的生命周期分为三个阶段,这些非线性的甲基化动态主要影响与神经系统相关的基因,并在双价标记的染色质区域富集。基于非线性改变的位点构建了一个类时钟分类器STageR(STage of aging estimatoR),能够准确预测小鼠的表观遗传阶段。并通过独立小鼠队列和公开可用的数据集中证明了所构建时钟的普适性。相关研究成果以“Nonlinear DNA methylation trajectories in aging male mice”为题发表在《nature communications》(IF16.6 / Q1)期刊上。

研究方法:
使用简化基因组亚硫酸盐测序(RRBS)对83只雄性C57BL/6J/Ukj小鼠在生命周期的五个时间点(3月龄、9月龄、15月龄、24月龄和28月龄)的结肠DNA样本进行甲基化模式分析。

研究结果:
(1)衰老过程中小鼠肠道的差异甲基化区域
利用MspI限制性内切酶,RRBS富集了富含GC的基因组区域,通常与关键的调控元件如启动子和增强子相关。原始甲基化数据的主成分分析(PCA)显示,第一主成分代表了数据总方差的25%以上,明显与样本的年龄相关。同一数据的无监督层次聚类分析表明,样本可以分为三个明显不同的主要生命阶段:早年(3月龄)、中年(9-15月龄)和晚年(24-28月龄)。为了关注与年龄相关的最大甲基化变化,将分析限制在衰老过程中位于差异甲基化区域的胞嘧啶(Differentially methylated regions,aDMR)。并计算了所有时间点组合之间的成对aDMR。
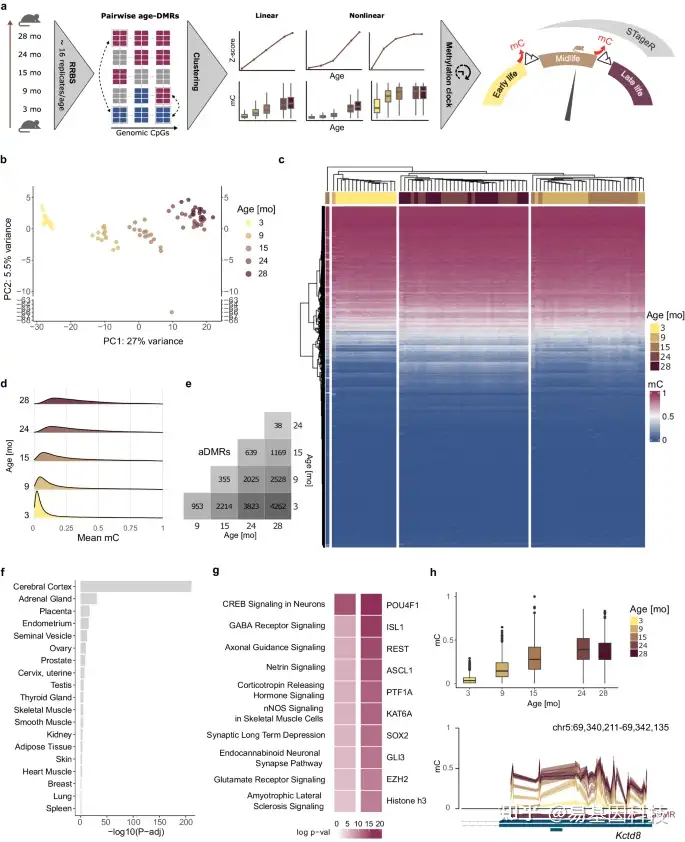
图1:衰老过程中雄性小鼠结肠的DNA甲基化动态变化。
- 实验策略。对83只不同年龄的成年雄性小鼠结肠DNA样本进行了RRBS测序。基于成对差异甲基化区域(aDMRs)中CpG位点的甲基化水平Z评分进行聚类,揭示了衰老过程中的差异甲基化轨迹。最富集的非线性聚类将生命分为三个阶段,并构建表观遗传时钟分类器——STageR。图中将分隔表观遗传阶段的高甲基化事件呈现为山峰状。
- 基于随机选取的10000个CpG位点进行主成分分析(PCA)。一个异常样本剔除。
- 在随机选取的10000个CpG位点中,通过所有样本的分层聚类揭示了三个主要的表观遗传生命阶段(早年、中年和晚年)。
- 不同年龄组中aDMR CpG位点的甲基化分布;n=16(3月龄、9月龄、15月龄、24月龄),n=18(28月龄)。
- 所有成对年龄组组合中衰老差异甲基化区域(aDMRs)数量。
- 与aDMRs相关基因中组织特异性基因的富集。显示了FDR校正后的超几何单侧P值。
- 与aDMRs相关基因的前十个富集的Ingenuity通路(左侧)和潜在的上游调控因子(右侧)。显示了Fisher精确检验的FDR校正后的单侧P值。
- Kctd8基因第一外显子中aDMR的甲基化模式。DNA甲基化水平以箱线图(上)中的平均水平(线)和基因组浏览器图(下)的置信区间(阴影区域)(下)显示;n=874(3月龄),n=834(9月龄),n=823(15月龄),n=841(24月龄),n=945(28个月),根据相应年龄组中55个CpG位点的可用测量值。箱线图中心线表示中位数,箱线限制显示第一和第三四分位数,上下须从铰链延伸到最大值或最小值,不超过铰链1.5×四分位距(IQR)。
(2)衰老中的非线性DNA甲基化轨迹
为了鉴定胞嘧啶甲基化的重复轨迹,对与差异甲基化区域(aDMRs)相关的CpG位点进行无监督聚类分析。
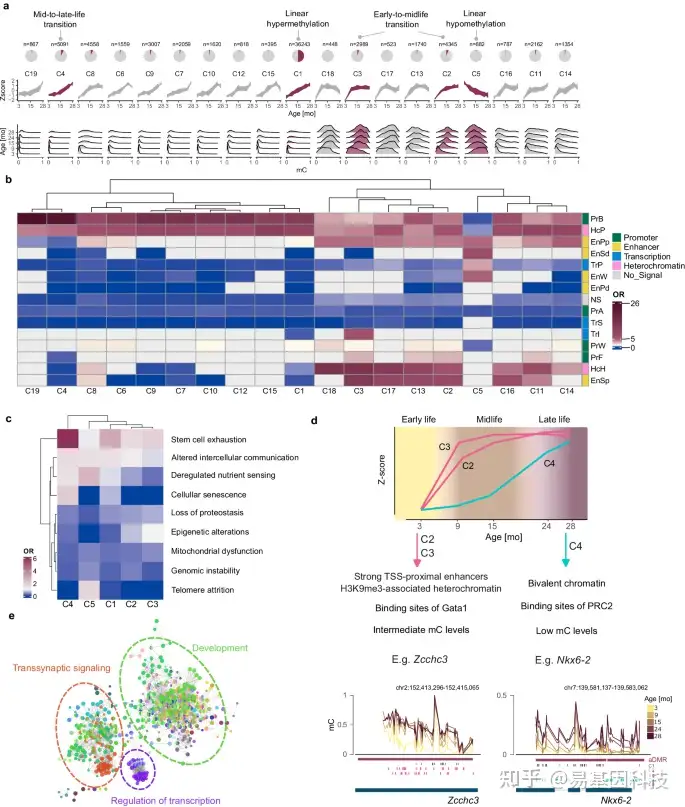
图2:研究中鉴定的所有19个甲基化轨迹。
- 从上到下:饼图显示特定簇中胞嘧啶比例在所有aDMR胞嘧啶中的占比;以Z分数表示的衰老过程中的DNA甲基化轨迹和每个年龄组的甲基化分布。红色阴影标记了进一步分析所选择的簇。
- 在每个簇中从ChromHMM获得的染色质片段的富集情况。PrA - Promoter, Active;PrW - Promoter, Weak;PrB - Promoter, Bivalent;PrF – 启动子侧翼区;EnSd – 强TSS远端Enhancer区;EnSp - 强TSS近端Enhancer区;EnW - Enhancer, Weak;EnPd - Enhancer, Poised TSS-distal;EnPp - Enhancer, Poised TSS-proximal;TrS - Transcription, Strong;TrP - Transcription, Permissive;TrI - Transcription, Initiation;HcP - Polycomb相关异染色质;HcH - H3K9me3相关异染色质;Ns – 无显著信号.
- 与五个代表性簇相关联的基因中衰老标志物基因集的富集情况。
- 提出将生命分为三个阶段的非线性DNA甲基化轨迹及其主要特征方案。
- 与C2、C3和C4簇相关基因的GO分析(按功能分组)。

图3:公开的亚硫酸盐测序测序数据集中,遵循非线性轨迹CpG位点的DNA甲基化。
上图:本研究中最富集非线性聚类C2、C3和C4中CpG位点的DNA甲基化。
中间图:27周龄小鼠结肠样本的RRBS数据,包括感染和未感染幽门螺杆菌(Helicobacter hepaticus)的小鼠。红色箭头突出显示了两种实验条件下中位数变化。
下图:从40日龄小鼠的结肠隐窝培养的类器官RRBS数据,并在标签指示的时间内继续培养。
(3)衰老过程中的非线性基因表达轨迹
为了评估在非线性DNA甲基化轨迹下的基因表达模式,研究人员对所有样本进行了bulk RNA-Seq测序分析。
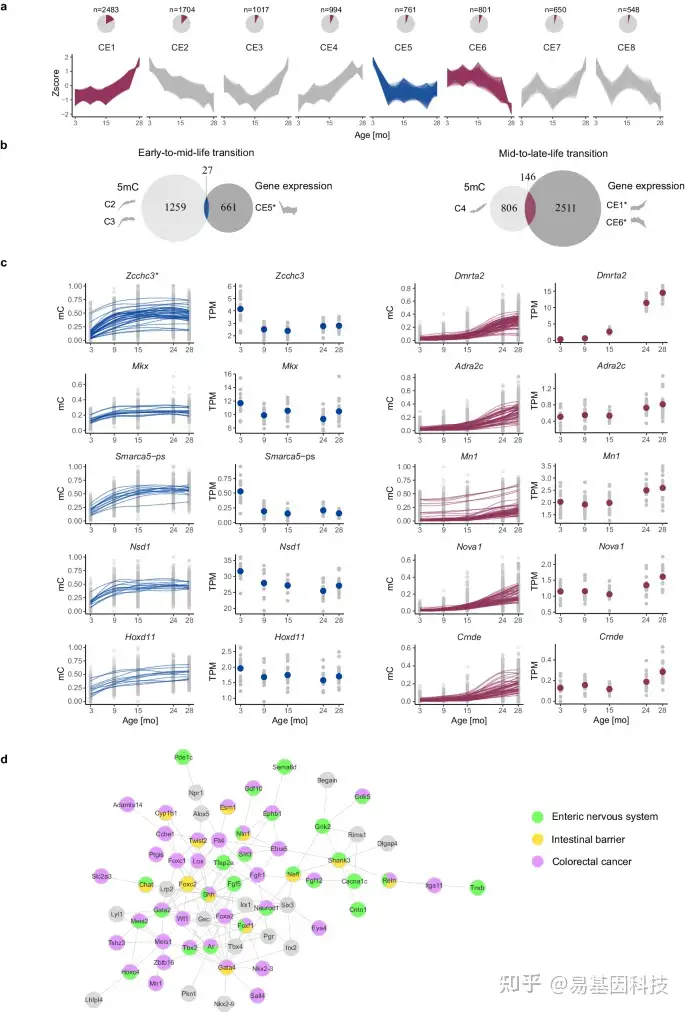
图4:衰老过程中小鼠结肠的非线性基因表达轨迹。
- 饼图显示特定簇中基因比例相对于所有差异表达基因的比例(上)。以Z评分表示衰老过程中的基因表达轨迹(下)。蓝色阴影对应于早至中年转换的簇,红色阴影对应于中至晚年转换的簇。
- 与至少一个早至中年或中至晚年甲基化轨迹CpG重叠的基因和在基因表达水平相同类型转换基因的交集分析。*仅包括RRBS数据集中覆盖的基因进行分析。
- 从b图中所示的交叉集中选择在早至中年转换(左侧)或中至晚年转换(右侧)中CpG数量最多的前五个基因。
- 从同时在DNA甲基化和基因表达水平上遵循中至晚年轨迹的基因创建的STRING蛋白-蛋白互作网络。在肠神经系统、肠屏障和/或结直肠癌中具有已知功能的基因用特定颜色标记。
(4)验证数据集
为了验证衰老过程中非线性甲基化轨迹的存在,研究人员分析了来自另一个小鼠实验装置的四个年龄的20只雄性C57BL6/J小鼠:3月龄(n=4)、7月龄(n=5)、12月龄(n=5)和27月龄(n=6)。
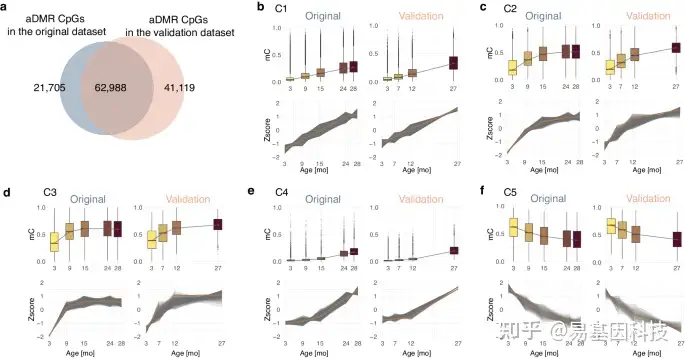
图5:验证数据集。
- 在原始数据集和验证数据集中,衰老差异甲基化区域(aDMR)CpG位点的重叠情况。
b–f. 分别展示了原始数据集(左侧)和验证数据集(右侧)中按年龄组划分的平均甲基化水平分布,以及C1-C5簇在衰老过程中的甲基化Z评分轨迹。黄线表示原始数据集中Z分数的三次平滑样条曲线。验证数据集绘制了每个聚类中与原始轨迹最接近的70%胞嘧啶。CpG位点数量:n=36243(C1原始)n=24920(C1验证);n=4345(C2原始)n=2956(C2验证);n=2989(C3原始)n=2025(C3验证);n=5091(C4原始)n=3502(C4验证);n=882(C5原始)n=602(C5验证)。
(5)基于聚类的表观遗传时钟STageR
表观遗传时钟通过5mC(5-甲基胞嘧啶)水平模拟年龄(chronological年龄),通常使用监督机器学习方法构建,如弹性网络回归。这些模型的应用通常依赖于一组固定的CpG位点,即模型CpG位点。这种时钟之一缺点是缺乏可传递性,尤其是在使用RRBS时,捕获和充分覆盖的位点数量在不同数据集之间高度变化。为了弥补这个缺点,作者提出基于高度相关且冗余的CpG甲基化信息来构建衰老时钟。例如,使用上述聚类的甲基化中心而不是单个CpG值。
本研究提出了一个表观遗传时钟分类器STageR(STage of aging estimatoR),它基于有限的甲基化信息预测潜在的衰老阶段。具体来说,基于三个非线性簇(C2、C3和C4)中CpG的中位甲基化水平,执行多项logistic弹性网络回归(multinomial logistic elastic net regression),以预测每个样本的衰老阶段(早年、中年或晚年)。因此在这种情况下,特征空间的维度从80000多个与年龄相关的CpG位点大幅降低到仅有的三个甲基化聚类。选择早至中年(3-9月龄)和中至晚年(15-24月龄)聚类是基于一些重要观察的结果:
①原始甲基化数据的分层聚类已经将样本分为三个主要组,即3月龄、9和15月龄、24和28月龄;
②第一轮聚类中检测到早至中年(C2和C3)以及中至晚年(C4)的甲基化聚类,连同C1和C6,表明这些聚类中的CpG位点表现出相对较强的信号;
③基因表达轨迹支持在DNA甲基化水平上识别的转换时机。具体来说,最大的聚类CE1和CE6支持中至晚年转换,CE5支持早至中年转换。
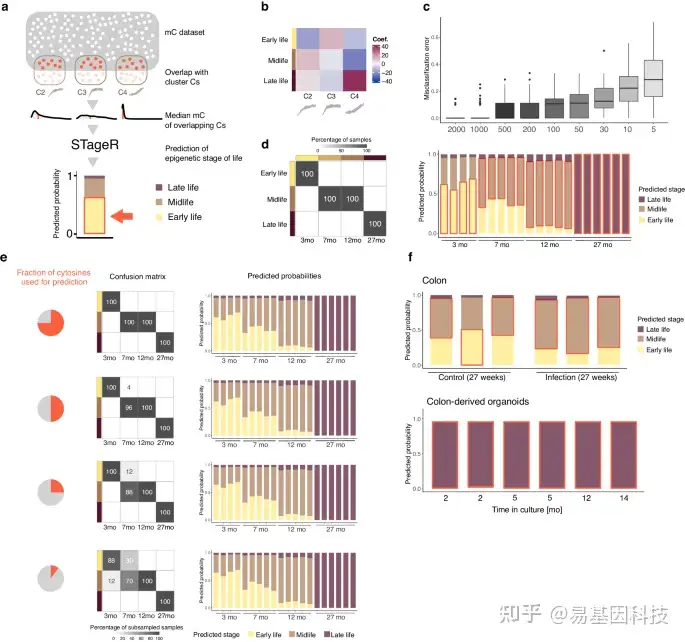
图6:STageR-基于聚类的表观遗传阶段分类器。
- 使用STageR预测表观遗传生命阶段的工作流程。首先,确定查询数据集与非线性CpG聚类的重叠部分。计算重叠CpG的DNA甲基化水平中位数,将特征空间维度显著降低到三个。STageR执行多项logistic弹性网络回归,为表观遗传生命的三个阶段分配概率。最高概率阶段在条形图中以红色突出显示。
- STageR模型中每个生命阶段的聚类在多项逻辑回归中的平均β系数。底部面板显示相关的Z分数轨迹。
- 在每个聚类中随机抽取一定数量胞嘧啶的十倍交叉验证模型中的误分类误差(x轴),每个箱线图中n = 100。
- 验证数据集的STageR预测。左侧:来自四个不同年龄组(x轴)验证样本的预测生命阶段(y轴)的混淆矩阵。右侧:所有验证样本的生命阶段(y轴)的预测概率(x轴)。红框表示生命阶段最大概率。
- 随机抽取75%、50%、25%和10%(从上到下)的每个聚类胞嘧啶进行STageR预测混淆矩阵和预测概率。
- 公开可用数据集的STageR预测。
总的来说,作者构建了一个基于聚类的表观遗传阶段时钟STageR,它能够捕捉到即使在使用少量数据时也与衰老相关的甲基化动态。稳健的衰老阶段分类器STageR可以轻松应用于其他数据集,即使只有少量聚类覆盖的胞嘧啶。
关于易基因简化基因组甲基化测序(RRBS)研究解决方案
简化甲基化测序(Reduced Representation Bisulfite Sequencing,RRBS)是利用限制性内切酶对基因组进行酶切,富集启动子及CpG岛等重要的表观调控区域并进行重亚硫酸盐测序。该技术显著提高了高CpG区域的测序深度,在CpG岛、启动子区域和增强子元件区域可以获得高精度的分辨率,是一种准确、高效、经济的DNA甲基化研究方法,在大规模临床样本的研究中具有广泛的应用前景。
为适应科研技术的需要,易基因进一步开发了可在更大区域内捕获CpG位点的双酶切RRBS(dRRBS),可研究更广泛区域的甲基化,包括CGI shore等区域。
为助力适用低起始量DNA样本(5ng)量多维度甲基化分析,易基因开发了富集覆盖CpG岛、启动子、增强子、CTCF结合位点的甲基化靶向基因组测序方法:extended-representation bisulfite sequencing(XRBS),实现了高灵敏度和微量样本复用检测,使其具有高度可扩展性,并适用于有限的样本和单个细胞基因组CG位点覆盖高达15M以上。
技术优势:
- 起始量:100ng gDNA;
- 单碱基分辨率;
- 多样本的覆盖区域重复性可达到85%-95%、测序区域针对高CpG调控区域,数据利用率更高;
- 针对性强,成本较低;
- 基因组CG位点覆盖高达10-15M,显著优于850K芯片。
应用方向:
RRBS/dRRBS/XRBS广泛应用于动物,要求全基因组扫描(覆盖关键调控位点)的:
- 队列研究、疾病分子分型、临床样本的甲基化 Biomarker 筛选
- 复杂疾病及肿瘤发病机制等甲基化研究
- 模式动物发育和疾病甲基化研究
易基因提供全面的表观基因组学(DNA甲基化、DNA羟甲基化)和表观转录组学(m6A、m5C、m1A、m7G)、染色质结构与功能组学技术方案(ChIP-seq、ATAC-seq),详询易基因:0755-28317900.

参考文献:
Olecka M, van Bömmel A, Best L, Haase M, Foerste S, Riege K, Dost T, Flor S, Witte OW, Franzenburg S, Groth M, von Eyss B, Kaleta C, Frahm C, Hoffmann S. Nonlinear DNA methylation trajectories in aging male mice. Nat Commun. 2024 Apr 9;15(1):3074.
相关阅读:
研究速递 | 肝脏DNA甲基化分析揭示表观遗传衰老对异生素代谢和转运基因的影响
全基因组DNA甲基化测序(WGBS)揭示衰老对肝再生的表观基因组调控机制
标签:DNA,Commun,基因,测序,CpG,甲基化,聚类,月龄 From: https://www.cnblogs.com/E-GENE/p/18163229