大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
癌症是全球主要的公共卫生威胁,虽然癌症死亡率自 1991 年达到顶峰以来持续下降,但仅在 2021 年,美国就有超过 60万人死于癌症。2020年,全球有近1000万人死于癌症,近年来一些低收入和中等收入国家的死亡率有所上升。因此,抗击癌症的需求仍然紧迫且未得到满足。研究表明,早期肿瘤检测对于改善癌症患者的预后至关重要。例如,肝细胞癌 (HCC) 的早期诊断时的五年生存率为 34%,但晚期诊断(远端转移)时,其生存率则降至3%。因此,开发用于早期癌症检测的检测方法至关重要。血浆中的细胞游离细胞DNA(cell-free DNA,cfDNA)是肿瘤检测的潜在生物标志物,但其中存在于约10%CpG二核苷酸中的半甲基化(hemi-methylation)模式尚未得到充分研究。
2024年7月20日,美国哥伦比亚大学Zhiguo Zhang(张志国)教授团队在Nature子刊《Nature Communications》杂志发表题为“Tumor detection by analysis of both symmetric- and hemi-methylation of plasma cell-free DNA”的研究论文,研究通过cfMeDIP-seq结合机器学习方法分析了肝脏肿瘤和血浆游离DNA(cfDNA)中的差异半甲基化区域(DHMRs),揭示了大多数DHMRs与相同样本中的差异甲基化区域(DMRs)不重叠,表明DHMRs可以作为独立的生物标志物。同时,通过分析患有肝癌或脑癌的个体样本以及无癌症个体样本(对照组)共计215例样本的cfDNA甲基化组,并利用DMRs、DHMRs或DMRs+DHMRs两者训练机器学习模型。结合DMRs+DHMRs的模型,比只使用DMRs或DHMRs训练的模型表现出更优越的性能,在验证队列中,区分对照组、肝癌和脑癌的AUROC值分别为0.978、0.990和0.983。这项研究支持了同时利用DMRs和DHMRs进行多癌种检测的潜力。

标题:Tumor detection by analysis of both symmetric- and hemi-methylation of plasma cell-free DNA(通过分析血浆cfDNA的对称甲基化和半甲基化来检测肿瘤)
期刊:Nature Communications
影响因子:IF 14.7 / 1区
技术平台:cfMeDIP-seq等
研究思路:
- 利用改进版甲基化DNA免疫沉淀测序(MeDIP-Seq)方法,分析来自肝脏肿瘤和脑肿瘤患者以及健康对照组的血浆cfDNA样本。
- 研究对称甲基化(symmetric methylation)和半甲基化(hemi-methylation)在肿瘤检测中的独立作用。
方案设计:
本研究通过分析肝癌患者、脑癌患者和健康对照者共计215例样本的cfDNA甲基化组图谱,并利用DMRs、DHMRs及两者结合训练机器学习模型。
开发两种甲基化DNA免疫沉淀和链特异性(strand-specific,ss)测序方法(MeDIP-Seq):基于基因组DNA的ssg-MeDIP-Seq和基于血浆cfDNA的sscf-MeDIP-Seq。使用pA-Tn5转座酶进行DNA片段化和链特异性标记。
使用机器学习模型:训练并分析基于差异甲基化区域(DMRs)和差异半甲基化区域(DHMRs)的数据集。使用GLMnet、随机森林和深度神经网络(DNN)进行模型训练和验证。
研究亮点:
本研究结果揭示大多数DHMRs与相同样本中的DMRs不重叠,表明DHMRs可以作为独立的生物标志物。训练的机器学习模型结合DMRs和DHMRs显示出比单独使用DMRs或DHMRs更好的性能,尤其是在区分对照组、肝癌和脑癌方面。
表明利用DMRs和DHMRs作为生物标志物,通过sscf-MeDIP-Seq方法分析血浆cfDNA,可以提高多癌种检测的准确性。研究支持将cfDNA甲基化和半甲基化分析作为癌症早期检测和分类的潜在手段。
本研究创新性地开发了sscf-MeDIP-Seq方法,能够同时分析cfDNA的对称甲基化和半甲基化。证明了DHMRs作为独立生物标志物在肿瘤检测中的潜力。机器学习模型的结合使用DMRs和DHMRs提高了肿瘤检测的准确性(尤其是在独立验证队列中)。研究提供了一种新的策略,通过分析血浆cfDNA的甲基化模式来检测和分类肿瘤,具有潜在的临床应用价值。
结果图形:
(1)开发基因组甲基化DNA免疫沉淀与链特异性测序方法(ssg-MeDIP-Seq)
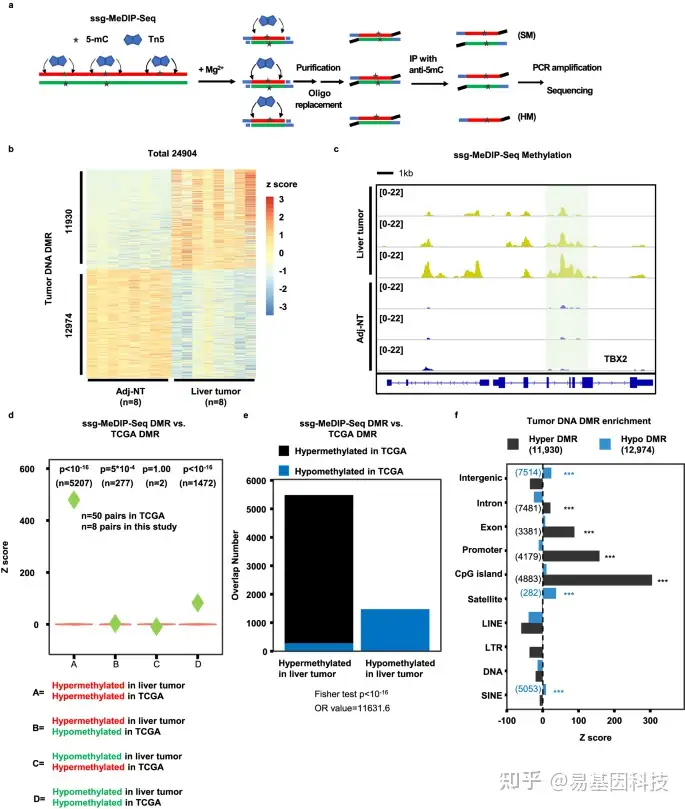
图 1:一种基于 pA-Tn5转座酶的MeDIP-Seq方法,用于以链特异性方式分析基因组DNA的甲基化组。
- ssg-MeDIP-Seq程序,用于以链特异性方式分析基因组DNA的DNA甲基化。SM对称性甲基化,HM半甲基化。
- 8个肝脏肿瘤样本与相应邻近非肿瘤组织(Adj-NT)之间的差异性甲基化区域(DMRs)热图。
- 3个肝癌症样本及相应邻近非肿瘤组织(Adj-NT)样本中TBX2基因位点的肝脏肿瘤 DNA DMR。
d-e. 通过ssg-MeDIP-Seq鉴定的肝癌DMRs与TCGA肿瘤样本中通过450K甲基化芯片鉴定的DMRs进行重叠分析,通过小提琴图(d)和条形图(e)展示。
f. 肝癌DNA DMRs的序列元件富集分析。首先将 DMRs 与每个注释位点重叠,并与随机分布中的重叠数量进行比较,以计算Z分数。P值是通过单侧随机分布计算得出,没有进行多重比较校正。显著富集的序列元件用星号标记,黑色(高甲基化 DMR)和蓝色(低甲基化DMR),每个类别中的DMR数量在括号中显示。(p<0.05; **p<0.01; ***p<0.001)。
(2)肝脏肿瘤DNA的差异半甲基化区域(DHMRs)和差异甲基化区域(DMRs)可能是独立生物标志物
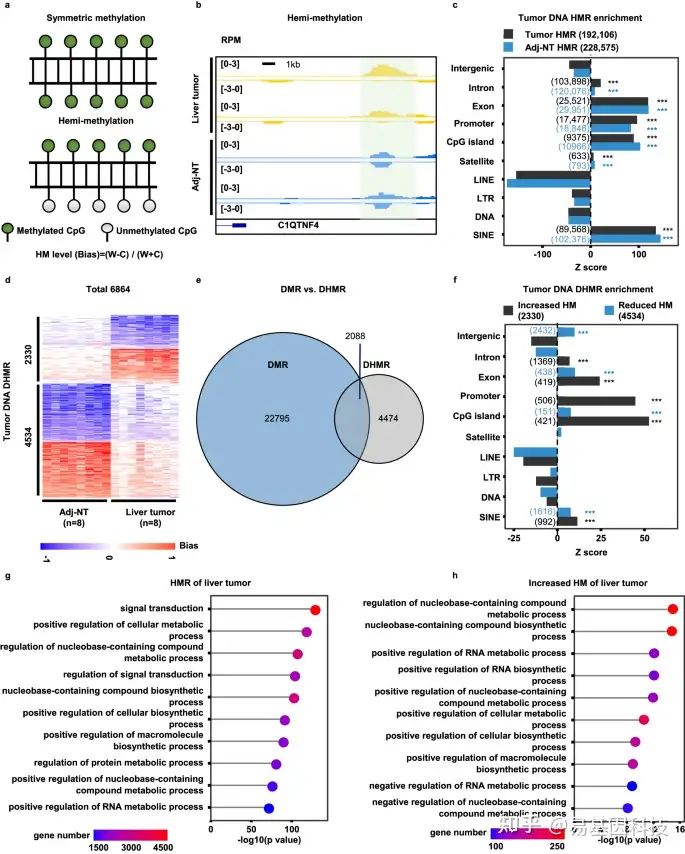
图2:通过ssg-MeDIP-Seq分析肝癌样本的DNA半甲基化。
- 对称性甲基化(SM)和半甲基化(HM)示意图。SM指在Watson和Crick两条链上的CpG二核苷酸位点上等量DNA甲基化,而HM区域(HMR)指在一条链上的CpG位点相较于另一条链偏好性甲基化。
- 两个肝脏肿瘤样本在C1QTNF4基因位点上的肿瘤DNA差异半甲基化区域(DHMR)快照,与其相应的邻近非肿瘤组织(Adj-NT)进行比较,阴影区域表示DHMR。
- 肝脏肿瘤DNA HMRs的序列富集分析。
- 8个肝脏肿瘤样本与其相应的Adj-NT相比的6864个DHMRs热图。HM水平以颜色从-1~1显示,其中有2330个肝脏肿瘤DNA DHMRs在Watson或Crick链上显示增加HM,而4534个DHMRs与对照组相比显示减少HM。
- 8个肝脏肿瘤样本的DMRs和DHMRs与其相应的Adj-NT的重叠比较分析。
- 与对照样本相比,增加(黑色)和减少(蓝色)的肝脏肿瘤DNA DHMRs的富集分析。
- 与肝脏肿瘤DNA HMRs邻近基因的GO功能富集分析。
- 与增加的HM相比,与Adj-NT对照样本中邻近肝脏肿瘤DNA DHMRs的GO功能富集分析。
以上结果表明肝脏肿瘤DHMRs和DMRs很可能是独立的生物标志物。
(3)开发用于分析cfDNA甲基化和半甲基化的sscf-MeDIP-Seq方法
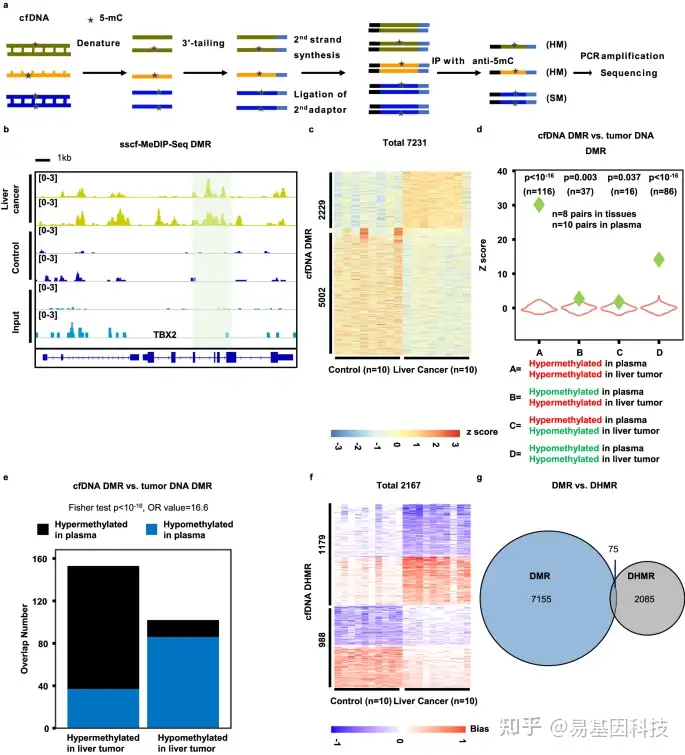
图3:一种用于分析血浆cfDNA甲基化的单链cfDNA甲基化DNA免疫沉淀测序(sscf-MeDIP-Seq)方法
- 用于分析cfDNA甲基化的sscf-MeDIP-Seq方法概述。SM对称性DNA甲基化,HM半甲基化。单链(ss)DNA上的DNA甲基化为半甲基化区域(HMR)。基于8个input样本,由ssDNA产生的HMR的数量可能很小。
- TBX2基因位点的cfDNA DMR快照。阴影区域突出了10个肝脏肿瘤患者的cfDNA样本与10个对照组cfDNA样本的DMR,每组仅显示两个样本。还显示了两个未进行甲基化DNA免疫沉淀的input样本的序列reads片段。
- 10个肝癌血浆样本和10个非肿瘤对照血浆样本的cfDNA DMR热图。
d-e. 小提琴图(d)和条形图(e)显示通过sscf-MeDIP-Seq鉴定肝脏肿瘤cfDNA DMRs与本研究中使用ssg-MeDIP-seq鉴定的肝脏肿瘤DNA DMRs之间的重叠。
f. 10个肝脏肿瘤样本和10个对照的血浆cfDNA DHMRs热图。
g. 与10个对照相比,10个肝脏肿瘤样本的血浆cfDNA DMRs和cfDNA DHMRs的重叠。
(4)大多数血浆cfDNA中的DHMRs与cfDNA中的DMRs不重叠,使用 DMR、DHMR 和 DMRs+DHMR 作为input训练的机器学习模型鉴定癌症类型
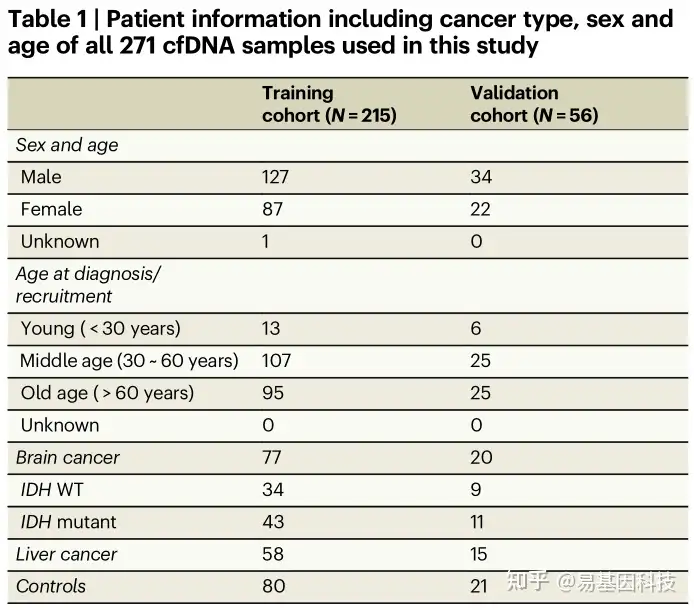
表1:本研究中使用的所有271个cfDNA样本的患者信息,包括癌症类型、性别和年龄
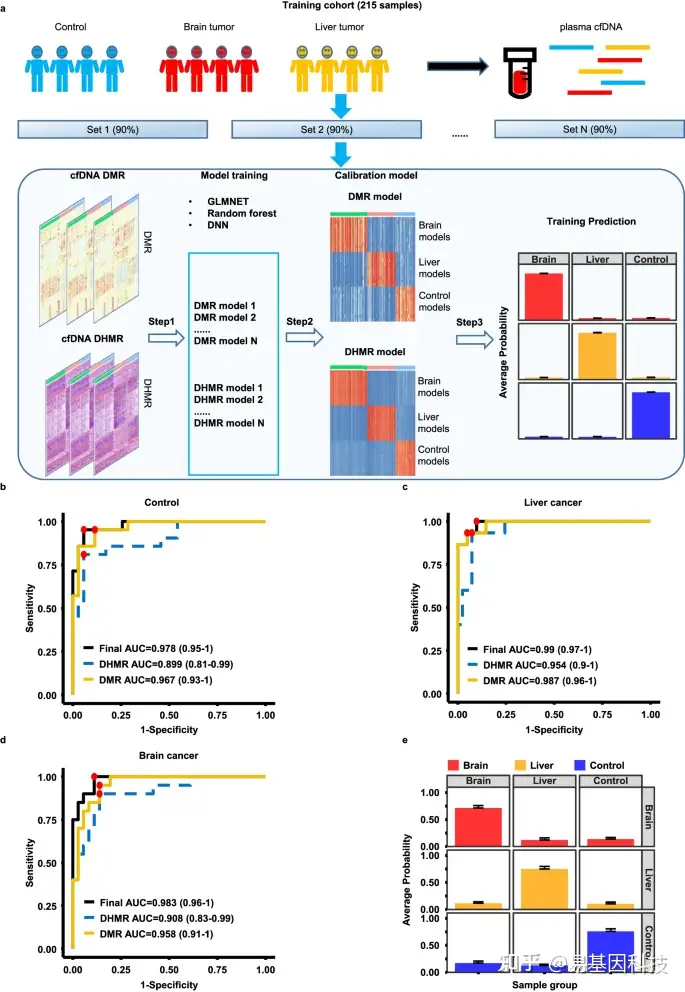
图4:使用DMRs和DHMRs以及机器学习模型进行多癌种检测。
- 机器学习模型训练的流程图。使用单链cfDNA甲基化DNA免疫沉淀测序(sscf-MeDIP-Seq)分析了来自三组(对照组、脑癌和肝癌患者)的271个cfDNA样本甲基化组。215个sscf-MeDIP-seq数据集(占80%)被用作训练队列,剩余的56个(占20%)样本作为独立的验证队列。训练队列用于选择DMR和DHMR并训练机器学习模型,每个样本组使用DMR或DHMR作为训练input,结果产生了10个模型。基于DMR和DHMR的模型然后进一步统一构建最终的校准模型。然后使用训练有DMRs、DHMRs和DMRs+DHMRs作为input的模型评估验证队列。
b-d. 评估模型性能,预测验证队列中的对照组(b)、肝脏肿瘤(c)和脑肿瘤(d)cfDNA样本,使用训练有DMRs、DHMRs或DMRs+DHMRs的模型。每种预测的最高灵敏度和特异性点用红点标记。每个模型的AUC的95%置信区间在括号中标记。
e. 使用训练有DMRs+DHMRs的模型,每组样本的平均预测概率。每一列代表验证样本组,每一行代表模型预测。条形图显示为平均值+标准误差。红色、黄色和蓝色条分别代表20个脑癌、15个肝癌和21个健康对照样本概率。
(5)通过cfDNA甲基化组区分神经胶质瘤亚型
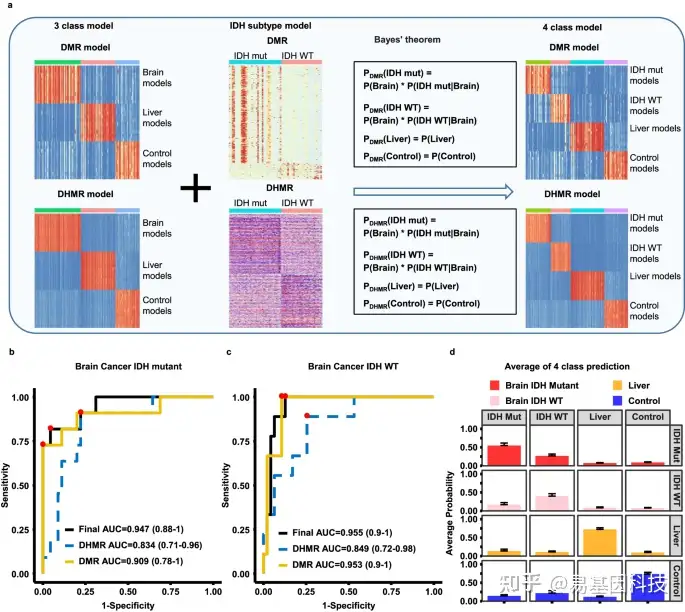
图5:使用sscf-MeDIP-Seq数据集预测脑肿瘤亚型。
- 构建脑肿瘤亚型模型流程图。首先使用训练队列样本鉴定的DMR和DHMR训练IDH WT和IDH突变体神经胶质瘤模型,然后将这些模型与基于贝叶斯定理的三类模型(对照组、肝癌和脑肿瘤)结合,得出用于预测四个样本组的模型:IDH WT脑肿瘤和IDH突变体脑肿瘤、肝脏肿瘤和对照样本。
- 训练DMR、DHMR、DMRs+DHMRs模型在验证队列中预测IDH突变体脑癌样本的评估。
- 训练DMR、DHMR、DMRs+DHMRs模型在验证队列中预测IDH WT(野生型)脑癌样本的评估。
- 使用训练DMRs+DHMRs模型,每组样本的平均预测概率。每一列代表验证队列中的样本组,每一行代表模型预测。条形图显示为平均值+标准误差。红色、粉色、黄色和蓝色条分别代表来自11个IDH突变体脑癌、9个IDH WT脑癌、15个肝癌和21个健康对照样本的概率。
(6)血浆cfDNA DMRs与肿瘤组织样本基因表达相关,这些基因表达能够预测患者生存率
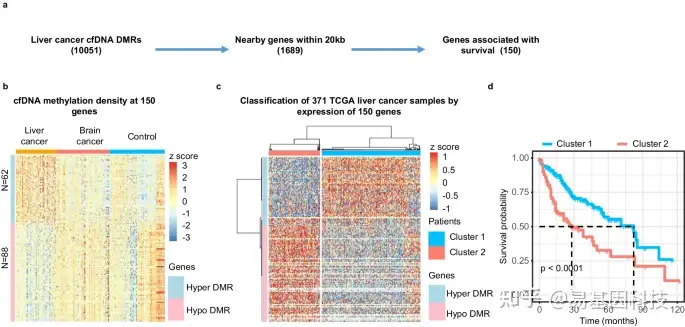
图6:基于TCGA肝脏肿瘤组织中具有肝脏肿瘤特异性血浆cfDNA DMR邻近的基因表达对肝癌样本进行分类及患者生存预测。
- 鉴定在启动子周围至少有一个肝癌cfDNA DMR且其在TCGA肝脏肿瘤组织样本中的表达与患者生存相关基因的流程。
- 与至少一个cfDNA DMR邻近的150个基因周围的sscf-MeDIP-Seq信号密度。以颜色表示的z分数,是sscf-MeDIP-Seq信号的log2(RPKM)。"HyperDMR"至少有一个高甲基化cfDNA DMR邻近的基因,"HypoDMR"有一个低甲基化cfDNA DMR邻近的基因。
- 基于上述鉴定的150个标记基因的表达,对TCGA-LIHC队列中的371个肝脏肿瘤样本进行分类。患者被分为两个聚类。颜色代表371个肝癌样本中150个基因的RNA-seq信号的log2(RPKM)的z分数。
- 对(c)中分为两个聚类的371个肝癌患者进行Kaplan-Meier生存分析。P值通过logrank检验计算。
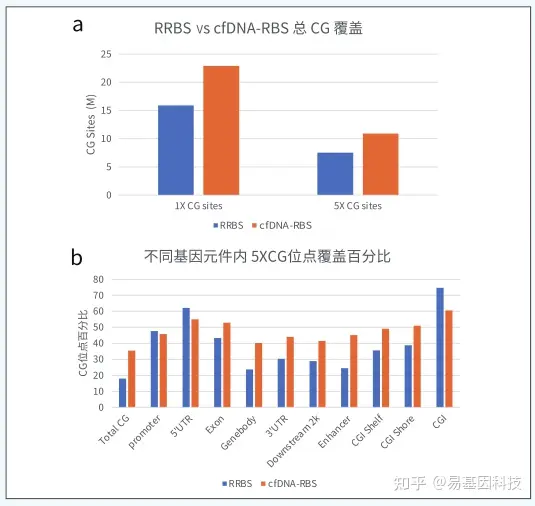
关于微量cfDNA甲基化测序(cfDNA-BS)技术
cfDNA片段化严重,片段大小常在150bp左右,现有甲基化检测技术包括cfMeDIP和微量WGBS等。无法做到碱基分辨、具有抗体特异性和非特异性捕获、覆盖深度低、检测成本高等特点。常规RRBS富集约70-350bp范围酶切片段,如对于CG含量高的片段将被切割的更碎而无法检测,保留下来的片段反而是CG含量低,无甲基化信息的基因片段。
技术优势:
- 超低起始量:100-500ul血浆或1ng cfDNA;
- 测序覆盖度高:20G测序数据,可达10M的CG位点覆盖,涵盖CpG岛、启动子、增强子、CTCF结合位点等多种核心调控区域
- 单碱基分辨率:在其覆盖范围内可精确分析每一个C碱基的甲基化状态;
- 性价比高:成本相对于现有技术大幅降低。
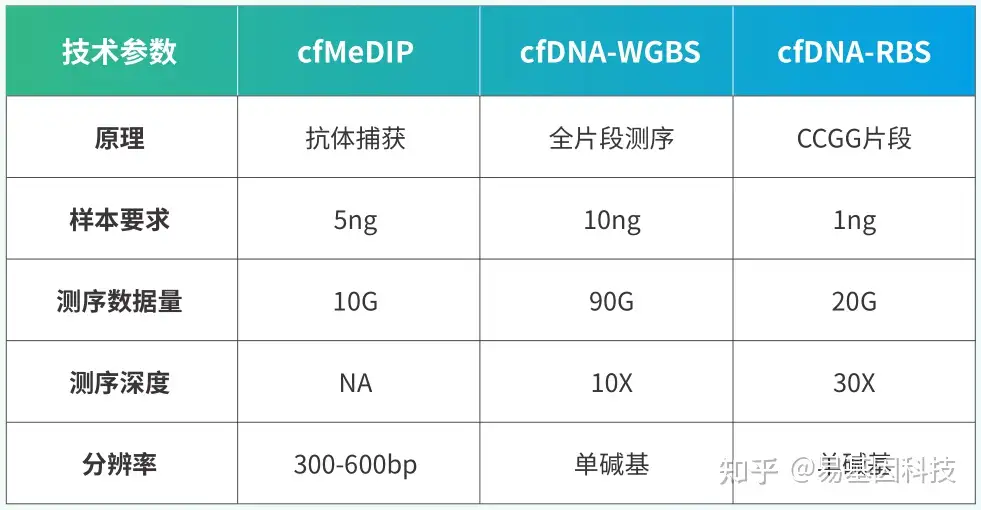
技术指标
应用场景:
- 癌前病变的癌变预警标志物检测
- 肿瘤早期筛查标志物检测
- 肿瘤预后标志物检测
- 药物疗效预测标志物检测
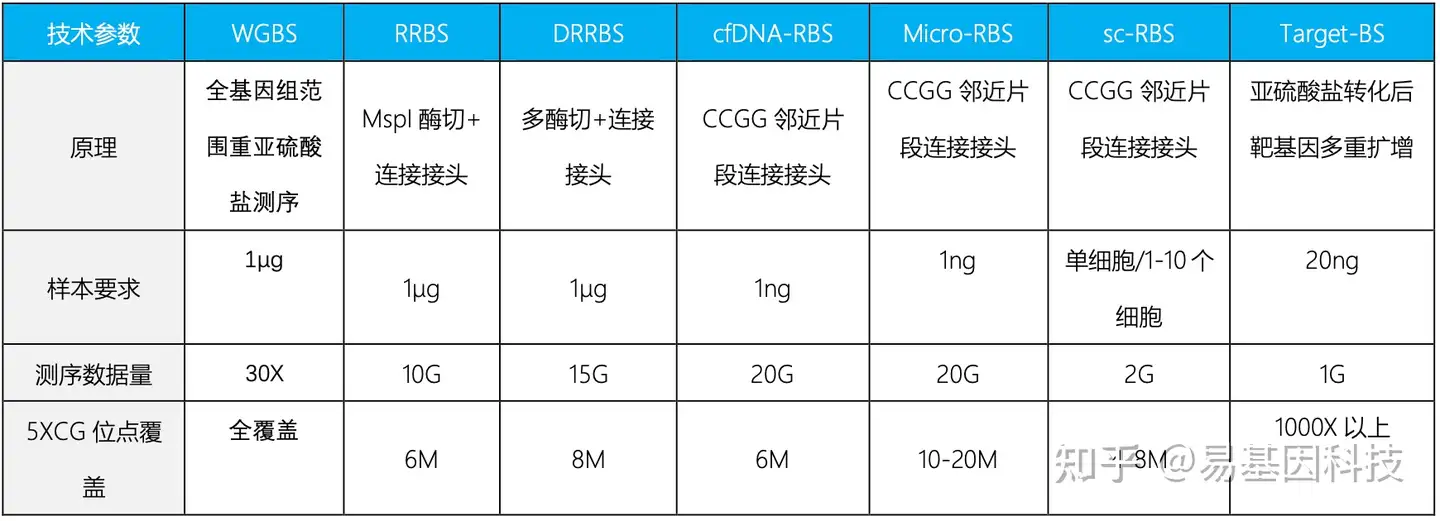
参考文献:
Hua X, Zhou H, Wu HC, Furnari J, Kotidis CP, Rabadan R, Genkinger JM, Bruce JN, Canoll P, Santella RM, Zhang Z. Tumor detection by analysis of both symmetric- and hemi-methylation of plasma cell-free DNA. Nat Commun. 2024 Jul 20;15(1):6113. pii: 10.1038/s41467-024-50471-1. doi: 10.1038/s41467-024-50471-1. PubMed PMID: 39030196.
相关阅读:
2. 3文一览:基于cfDNA甲基化的液体活检在胰腺疾病中的研究进展
4. 深度综述 | cfDNA甲基化诊断和监测肿瘤的研究进展与展望:胰腺癌
5. 揭秘:cfDNA甲基化在器官和组织损伤检测中的强大力量
6. Nature子刊:cfDNA甲基化组多模式分析早期检测食管鳞状细胞癌和癌前病变
7. 全基因组cfDNA甲基化分析提高了早期乳腺癌无创诊断成像的准确性
8. 精准医学:新发现!全基因组cfDNA甲基化分析或可用于胰腺癌早期诊断应用
9. 精准医学:结直肠癌诊断和监测的cfDNA甲基化标志物MYO1-G
10. 前沿技术:cfDNA甲基化分析揭示造血细胞移植的所有主要并发症
11. 新品:新型肿瘤标志物检测利器——cfDNA甲基化测序(cfDNA-RBS)
12. 技术推介 | 微量cfDNA简化基因组甲基化测序(cfDNA-RBS)
13. 课程回顾|基于微量cfDNA甲基化的液体活检如何开展?
14. 课程回顾|表观遗传学--cfDNA甲基化疾病早筛研究思路分享
标签:DMRs,DHMRs,甲基化,半甲基化,样本,多癌种,DNA,cfDNA From: https://www.cnblogs.com/E-GENE/p/18324825