大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
丹参(Salvia miltiorrhiza,S. miltiorrhiza)是一种具有重要经济价值和药用价值的模式药用植物,丹参的根会合成一组称为丹参酮(tanshinone)的二萜类亲脂性生物活性成分。丹参酮的生物合成和调控引起广泛关注。DNA甲基化变化在调控植物种子发育、茎和叶生长、春化、果实成熟和次级代谢等方面发挥着重要作用。然而丹参的甲基化组尚未得到分析,DNA甲基化在丹参酮合成过程中的调节机制仍然未知。
2023年05月31日,中国医学科学院药用植物研究所研究员卢善发团队在《Hortic. Res.》杂志在线发表了题为“Characteristics of Salvia miltiorrhiza methylome and the regulatory mechanism of DNA methylation in tanshinone biosynthesis”的研究论文,该研究利用全基因组重亚硫酸盐测序(WGBS)等分析揭示了丹参酮积累与关键酶基因甲基化水平之间的相关性,并提示CHH甲基化水平在调控丹参酮生物合成中的意义。

标题:Characteristics of Salvia miltiorrhiza methylome and the regulatory mechanism of DNA methylation in tanshinone biosynthesis(丹参的甲基化表征及DNA甲基化在丹参酮生物合成中的调控机制)
发表期刊:Horticulture Research
发表日期:2023年05月31日
影响因子:IF 8.7/ 1区
技术:全基因组重亚硫酸盐测序(WGBS)、RNA-seq等
样品及方法:
- S. miltiorrhiza 系 99-3丹参在试验田自然条件下生长,三月底从两年生的植物中采集了March_root样品,7月下旬采集了July_root和July_leaf样本,收获的植物样本立即冷冻在液氮中直到使用。
- BS-seq文库构建与全基因组重亚硫酸盐测序;亚硫酸盐测序数据比对和甲基化水平计算;差异甲基化分析
- 转录组(RNA-seq)分析和qRT-PCR检测
- sRNA测序和分析
- 5-氮杂胞苷(5-Azacytidine)处理和丹参酮分析
摘要
本研究应用无偏好性的全基因组重亚硫酸盐测序(WGBS)分析了丹参根和叶的单碱基分辨率DNA甲基化组。比较分析揭示了CG、CHG和CHH序列的差异甲基化模式,以及DNA甲基化与基因和小RNA(sRNA)表达之间的关联。分析结果表明,低甲基化基因的表达水平较高,24nt sRNA(24-nucleotide sRNA)可能关键性参与丹参RdDM(RNA-directed DNA methylation)通路。DNA甲基化变异分析表明,CHH甲基化是造成差异的主要原因。Go富集分析表明,与March_root相比,hypoCHHDMR下游重叠基因在July_root的二萜生物合成过程显著富集。丹参酮生物合成相关酶基因如DXS2、CMK、IDI1、HMGR2、DXR、MDS、CYP76AH1、2OGD25和CYP71D373,在July_root基因启动子或下游区域中的CHH甲基化水平低于March_root。与March_root相比,July_root基因表达上调,DNA甲基化抑制剂5-氮杂胞苷的处理显著促进了丹参酮合成。研究结果揭示了DNA甲基化通过改变丹参酮关键酶基因启动子或下游CHH甲基化水平,在丹参酮生物合成中起重要调控作用。
研究结果:
(1)丹参DNA甲基化通路相关基因及整体DNA甲基化模式
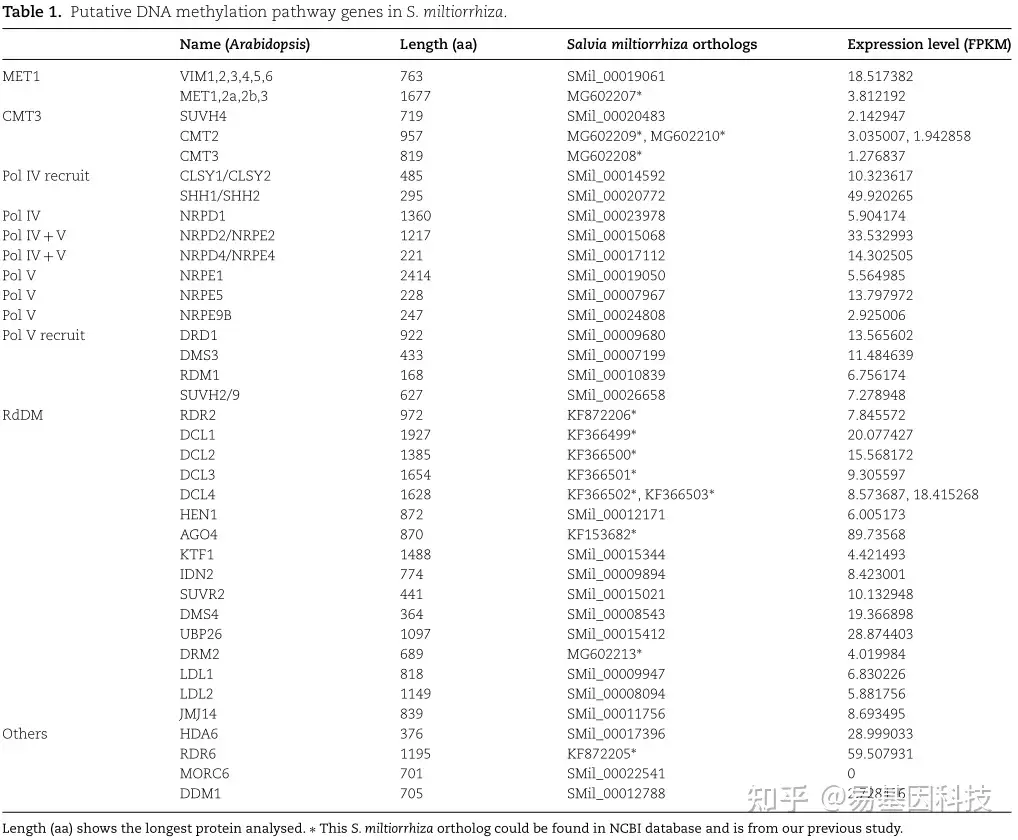
表1:丹参中推定的DNA甲基化通路基因
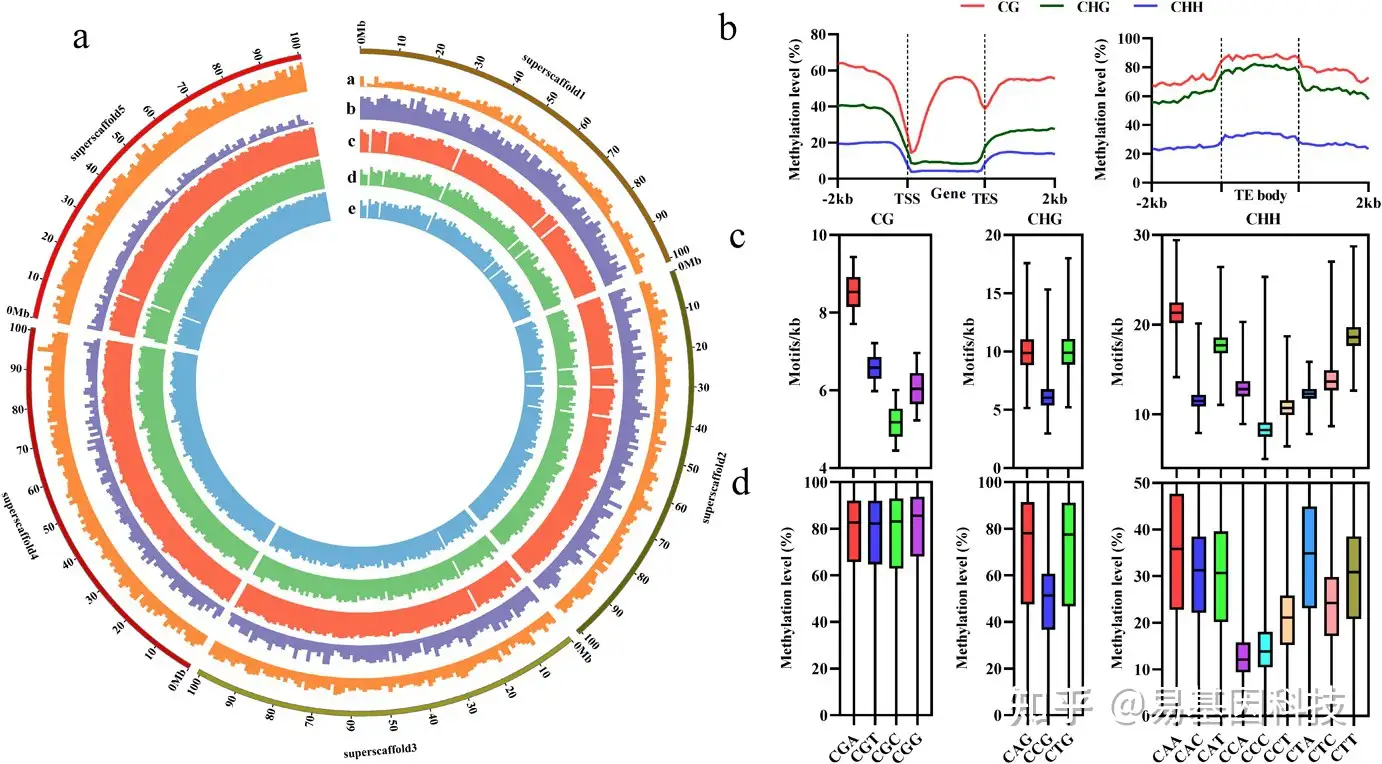
图2:丹参(S. miltiorrhiza )DNA甲基化图谱。
- 在March_root中1-Mb bins范围内绘制DNA甲基化图谱。圆周上的单位显示兆值(Mb)。Track a,TE覆盖率(5%-18%/Mb)。Track b,基因密度(4-89/Mb)。Track c,CG甲基化水平(1.6%-87%/Mb)。Track d,CHG甲基化水平(1.2%-77.5%/Mb)。Track e,CHH甲基化水平(0.7%–32.6%/Mb)。
- 基因和TE的CG、CHG和CHH的DNA甲基化水平。
- 区域内子序列的密度。
- 区域内子序列的DNA甲基化水平。
(2)DNA甲基化在基因表达中的作用
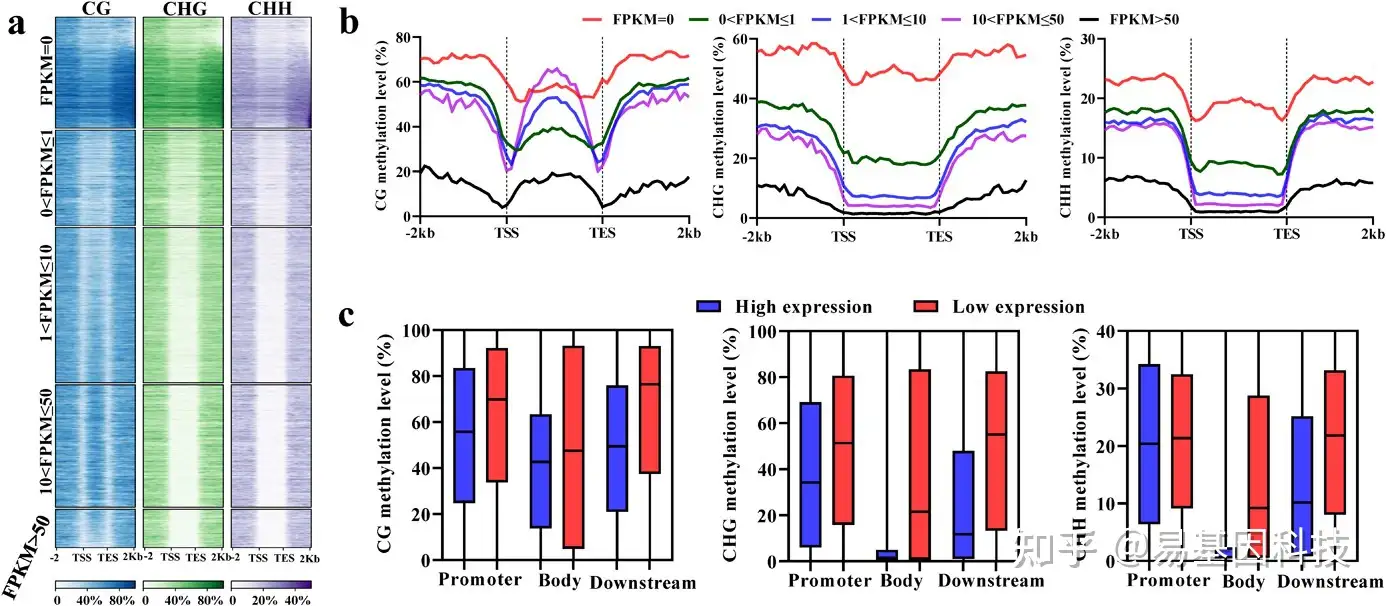
图2:DNA甲基化在基因表达中的作用。
- 不同基因体(gene body)和2kb周围区域的DNA甲基化水平。根据表达水平将基因分为五组。将基因体和周围区域分别等分为20个bin进行甲基化分析。
- CG、CHG和CHH序列中五个基因组的平均DNA甲基化。
- 每个基因区域的低表达基因和高表达基因之间的DNA甲基化比较。1/3表达水平最高的基因中定义为高表达,1/3表达水平最低的基因被定义为低表达。Promoter,上游2kb。Downstream,下游2kb。
(3)sRNA与DNA甲基化的相关性
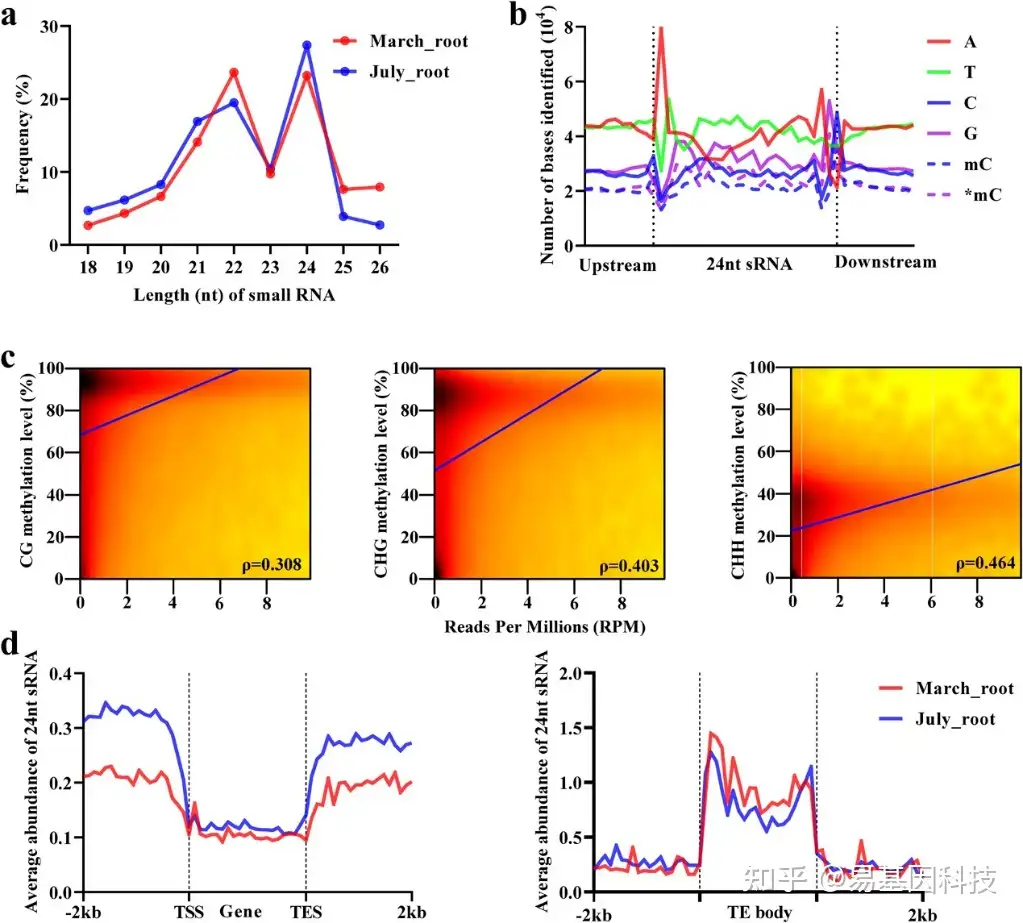
图3:DNA甲基化与24nt sRNA的相关性。
- March_root和July_root中sRNA的长度分布。
- 24nt sRNA图谱和10 nt周围区域的核苷酸频率分布。mC为有义链上的甲基化胞嘧啶;mC*为反义链上的甲基化胞嘧啶。
- DNA甲基化与24nt sRNA丰度之间的相关性。ρ:Spearman秩相关系数(P<0.001)。
- 24nt sRNA在March_root和July_root的基因/TE体和2kb周围区域的平均丰度(RPM)分布。
(4)不同丹参(S. miltiorrhiza)样品DNA甲基化变化特征
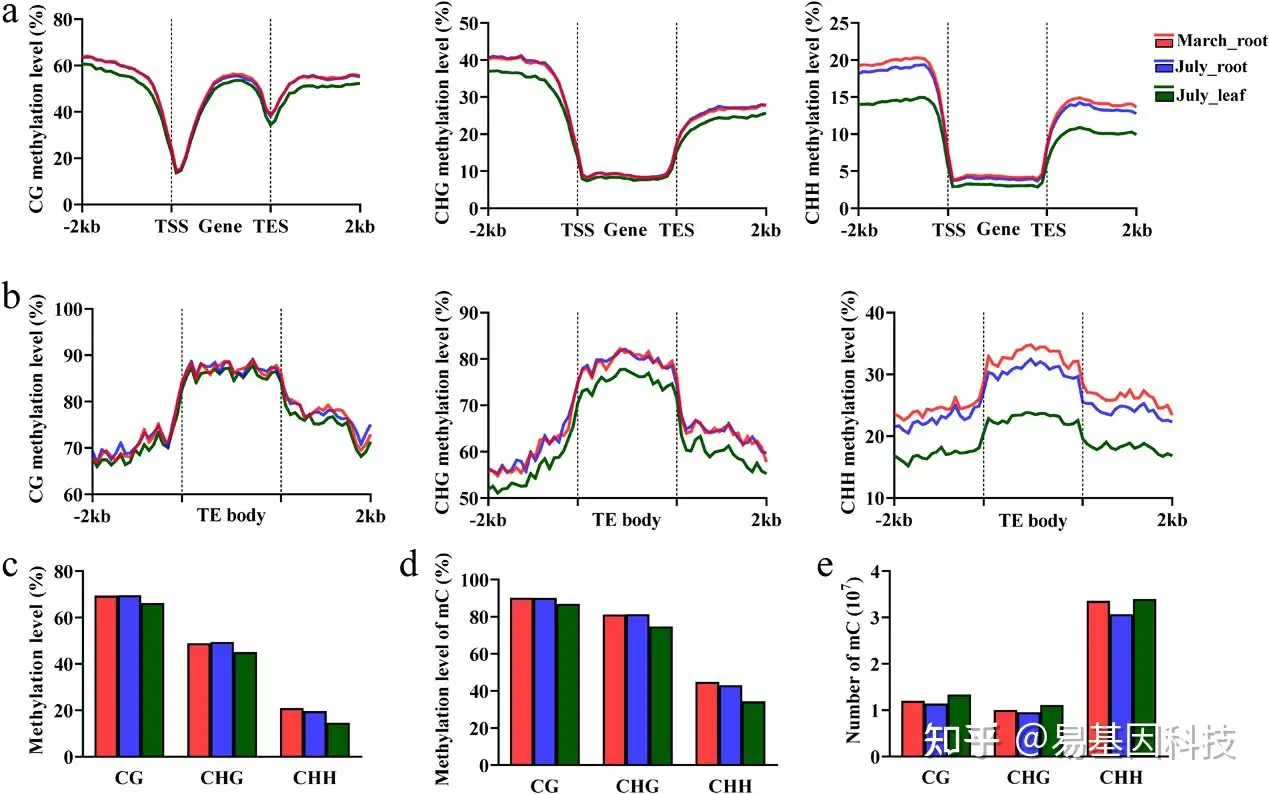
图4:丹参全基因组DNA甲基化比较分析。
- 三个样本中基因体和周围区域的CG、CHG和CHH序列中的DNA甲基化水平。
- 三个样本中TE体和周围区域的CG、CHG和CHH序列中的DNA甲基化水平。
- 三个样本中所有序列的整体甲基化水平。
- 三个样本中所有序列mC的整体甲基化水平。
- 柱状图显示三个样本中所有三个序列的mC数量。
(5)DNA甲基化参与萜烯(terpenes)的生物合成和代谢
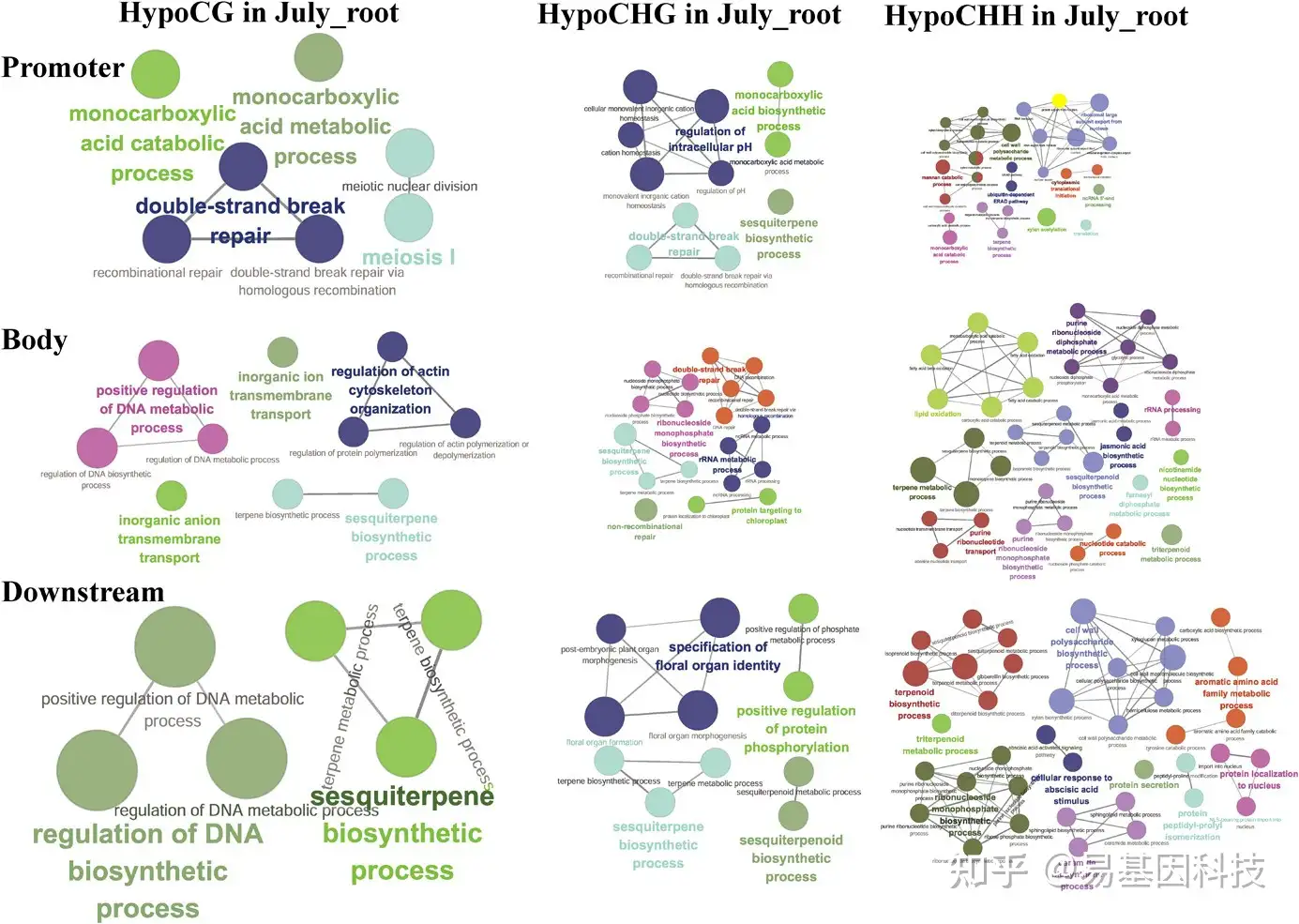
图5:与March_root相比,July_root的 hypoDMR相关基因在生物学过程显著富集。启动子表示启动子区域与hypoDMR重叠的基因。Body表示gene body与hypoDMR重叠的基因。Downstream表示下游基因与hypoDMR重叠的基因。
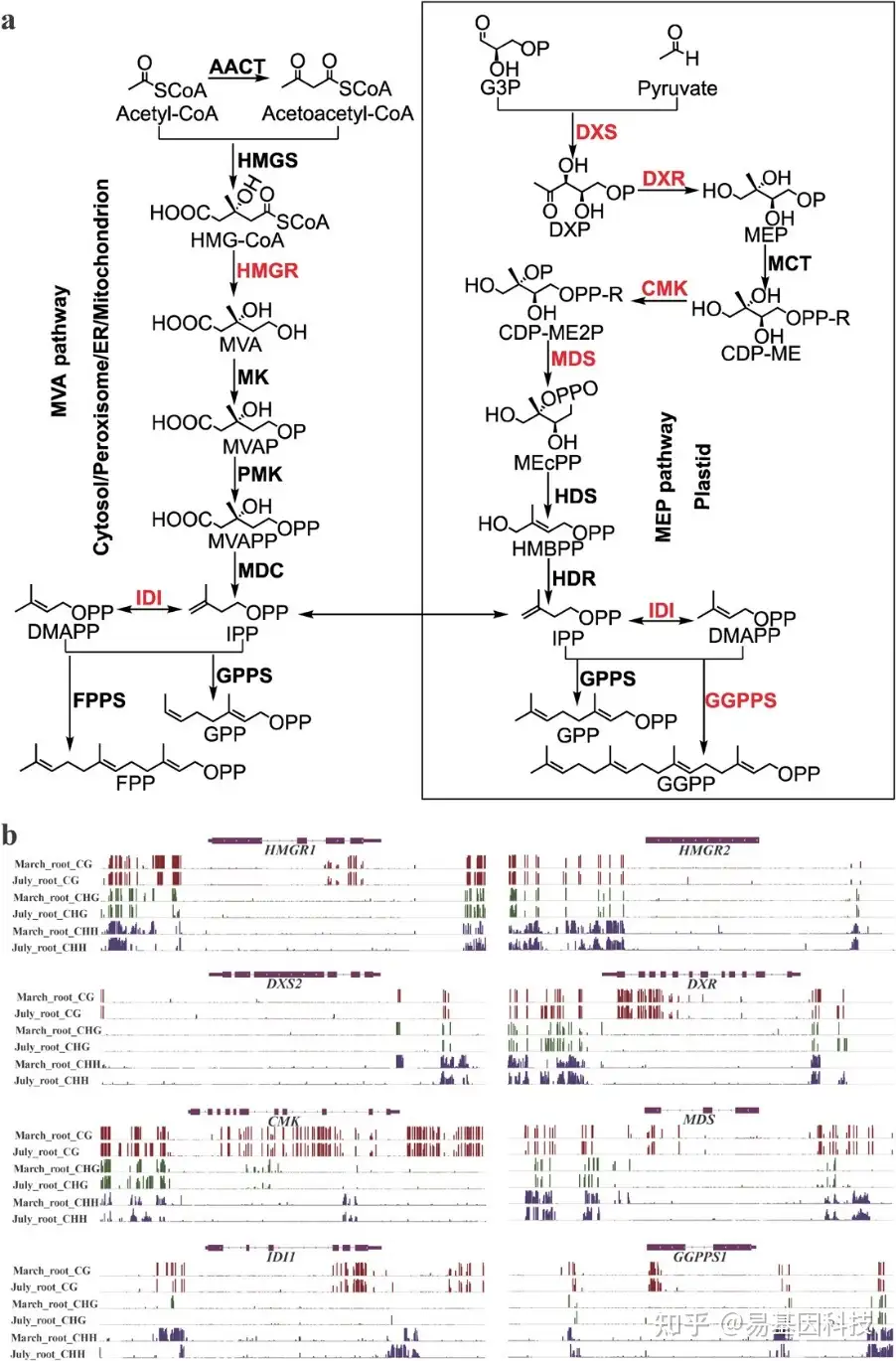
图6:丹参酮生物合成上游通路中的DMR相关酶基因。
- 丹参酮生物合成的上游通路。包括质体中的MEP通路,细胞质、内质网(endoplasmic reticulum,ER)、过氧化物酶体和线粒体中的MVA通路,以及中间体二磷酸前体的生物合成。其中,MEP通路在1-脱氧-D-木酮糖5-磷酸合成酶(DXS)的催化下合成中间体1-脱氧-D -木酮糖5-磷酸(DXP)、2- C-甲基-D -赤藓糖醇4-磷酸(MEP)、4-二磷酸胞苷基-2-C-甲基-D-赤藓糖醇2-磷酸(CDP-ME)、4-二磷酸胞苷基-2- C-甲基-D-赤藓糖醇2-磷酸(CDP-ME2P)、2-C-甲基-D-环二磷酸(MEcPP)、1-羟基-2-甲基-2-丁烯基4-二磷酸(HMBPP)和异戊烯基二磷酸(IPP)。分别生成1-脱氧-D-木酮糖5-磷酸还原异构酶(DXR)、2- C-甲基-D-赤藓糖醇4-磷酸胞苷基转移酶(MCT)、4-二磷酸胞苷基-2- C -甲基-D-赤藓糖醇激酶(CMK)、2-C -甲基-赤藓糖醇2,4-环二磷酸合酶(MDS)、1-羟基-2-甲基-2-丁烯基4-二磷酸合酶(HDS)和1-羟基-2-甲基-2-(E)-丁烯基4-二磷酸还原酶(HDR)。MVA通路在乙酰辅酶A C-乙酰转移酶(AACT)、3-羟基-3-甲基戊二酰-CoA合成酶(HMGS)、3-羟-3-甲基戊四酰-CoA还原酶(HMGR)、甲戊酸激酶(MK)的催化下分别生成中间体3-羟基-3-甲戊二酰辅酶A(HMG-CoA),5-磷酸甲戊酸激酶(PMK)和甲戊酸焦磷酸脱羧酶(MDC)。IPP可以在异戊烯基二磷酸异构酶(IDI)的催化下转化为二甲基烯丙基二磷酸(DMAPP)。二磷酸前体香叶基香叶基二磷酸(GGPP)的形成在香叶基香叶基二磷酸盐合酶(GGPPS)的催化下进行。红色代表DMR相关基因。
- 整合基因组Viewer显示DMR相关酶基因的DNA甲基化水平
(6)mCHH在关键酶基因启动子和下游区域对丹参酮生物合成的调控
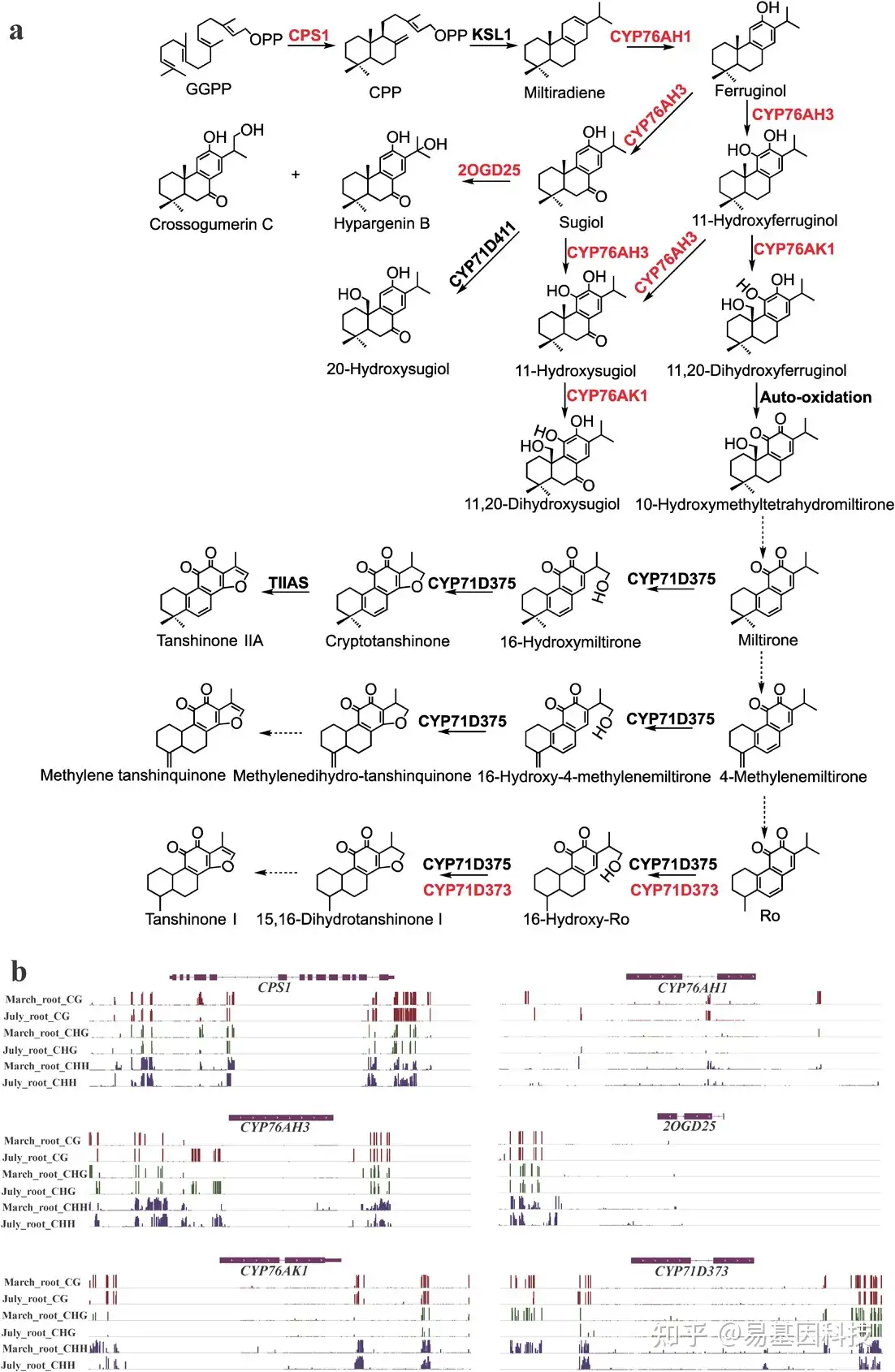
图7:丹参酮生物合成下游通路中的DMR相关酶基因。
- 丹参酮相关的二萜生物合成下游通路。红色代表DMR相关酶基因。实线箭头和虚线箭头分别表示已建立的关系和假设的关系。
- 综合基因组学Viewer显示DMR相关酶基因的DNA甲基化水平。
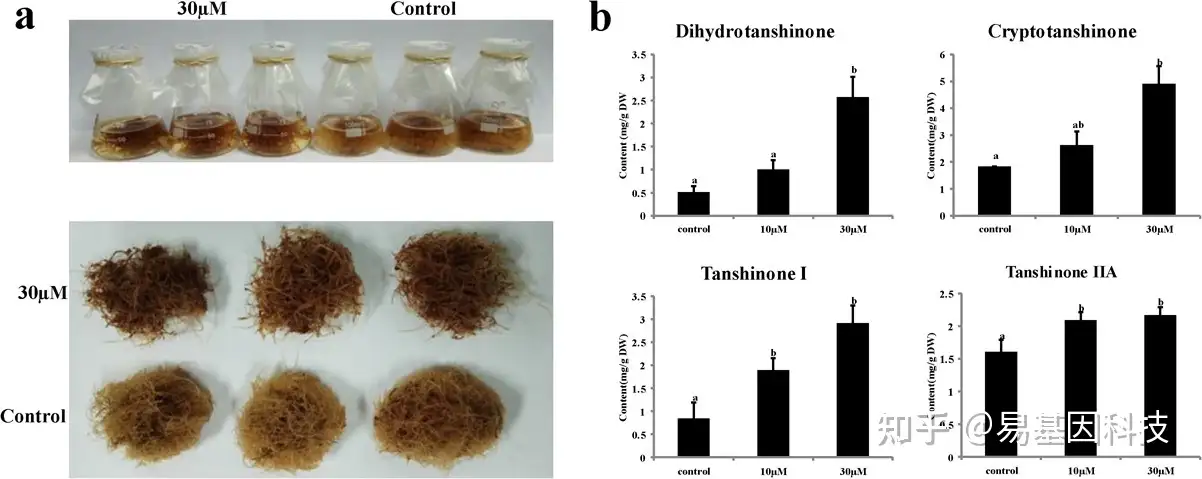
图8:5-氮杂胞苷在丹参毛状根丹参酮生物合成中的作用。
- 有或没有5-氮杂胞苷处理的丹参毛状根表型。
- 用不同浓度的5-氮杂胞苷处理30d后的丹参毛状根中二氢丹参酮I(dihydrotanshinone I)、隐丹参酮(cryptotanshinone)、丹参酮I(tanshinone I)和丹参酮IIA(tanshinone IIA)的含量。显示三次生物学重复的平均值。误差条代表SE。通过单因素方差分析计算5%水平的显著差异以不同的字母表示。
研究结论
本研究首次在丹参中绘制了单碱基分辨率的全基因组DNA甲基化图谱。结果表明, DNA低甲基化可以上调丹参的基因表达,24nt sRNA可能是RdDM通路的主要参与者。此外,DMC/DMR分析表明,差异甲基化主要发生在CHH序列中,与March_root相比,July_root中与hypoCHHDMR相关的基因在萜烯生物合成过程中富集。最重要的是,在July_root和March_root之间的14个DMR相关丹参酮生物合成酶基因中,包括DXS2、CMK、IDI1、HMGR2、DXR、MDS、CYP76AH1、2OGD25和CYP71D373在内的9个基因在July_root基因启动子区或下游区域表现出CHH低甲基化。进一步的DNA甲基化抑制剂处理促进了丹参毛状根中丹参酮的生物合成。总之,DNA甲基化可以通过丹参酮生物合成酶基因启动子和下游的CHH去甲基化来促进丹参酮的生物合成。本研究为丹参酮生物合成的表观遗传学调控机制提供了新的见解,将有助于进一步提高丹参活性化合物的产量。
关于易基因全基因组重亚硫酸盐测序(WGBS)
全基因组重亚硫酸盐甲基化测序(WGBS)可以在全基因组范围内精确的检测所有单个胞嘧啶碱基(C碱基)的甲基化水平,是DNA甲基化研究的金标准。WGBS能为基因组DNA甲基化时空特异性修饰的研究提供重要技术支持,能广泛应用在个体发育、衰老和疾病等生命过程的机制研究中,也是各物种甲基化图谱研究的首选方法。
易基因全基因组甲基化测序技术通过T4-DNA连接酶,在超声波打断基因组DNA片段的两端连接接头序列,连接产物通过重亚硫酸盐处理将未甲基化修饰的胞嘧啶C转变为尿嘧啶U,进而通过接头序列介导的 PCR 技术将尿嘧啶U转变为胸腺嘧啶T。
应用方向:
WGBS广泛用于各种物种,要求全基因组扫描(不错过关键位点)
- 全基因组甲基化图谱课题
- 标志物筛选课题
- 小规模研究课题
技术优势:
- 应用范围广:适用于所有参考基因组已知物种的甲基化研究;
- 全基因组覆盖:最大限度地获取完整的全基因组甲基化信息,精确绘制甲基化图谱;
- 单碱基分辨率:可精确分析每一个C碱基的甲基化状态。

易基因科技提供全面的DNA甲基化研究整体解决方案,详询易基因:0755-28317900。
参考文献:
Li J, Li C, Deng Y, Wei H, Lu S. Characteristics of Salvia miltiorrhiza methylome and the regulatory mechanism of DNA methylation in tanshinone biosynthesis. Hortic Res. 2023 Jul;10(7):uhad114.
相关阅读:
科研速递 | 全基因组DNA甲基化测序(WGBS)揭示儿童哮喘增强子区域的整体低甲基化
项目文章 | WGBS等揭示SOX30甲基化在非梗阻性无精症中的表观遗传调控机制
项目文章|WGBS+RNA-seq揭示PM2.5引起男性生殖障碍的DNA甲基化调控机制
14种全基因组DNA甲基化测序(WGBS)标准分析比对软件的比较 | 生信专区
标签:DNA,基因,基因组,甲基化,丹参酮,WGBS,root From: https://www.cnblogs.com/E-GENE/p/17653782.html