大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
口腔鳞状细胞癌(Oral squamous cell carcinoma, OSCC)是世界范围内最常见的癌症类型之一。尽管已有研究表明5-甲基胞嘧啶(5-methylcytosine, 5mC)和5-羟甲基胞嘧啶(5-hydroxymethylcytosine, 5hmC)在口腔鳞状细胞癌中的整体缺失,但对OSCC中5mC和5hmC进行全基因组、单碱基分辨和同时定位仍未完成。同样,5mC和5hmC如何共同导致OSCC基因异常表达的机制尚未完全阐明。
2023年10月12日,南华大学王丹玲教授和万正卿为共同通讯作者在《Front Genet》杂志发表题为“Unidirectional alteration of methylation and hydroxymethylation at the promoters and differential gene expression in oral squamous cell carcinoma”的研究论文,该研究利用平行全基因组亚硫酸盐测序(WGBS)和全基因组氧化亚硫酸氢测序(oxWGBS),在成对原发口腔鳞状细胞癌样本及其正常癌旁组织(NATs)中以单核苷酸分辨率表征5mC和5hmC图谱,并采用多组学方法分析5mC和5hmc修饰对OSCC差异基因表达的影响。易基因科技为本研究提供oxWGBS+WGBS测序分析服务。

研究摘要:
本研究通过oxWGBS+WGBS在OSCC样品中观察到不同基因组区域的5mC和5hmC整体水平降低。在启动子区域,OSCC中共鉴定出6921个差异甲基化区域和1024个差异羟甲基化区域。与5mC和5hmC共修饰相比,在启动子区域5mC和5hmC单修饰与基因表达更大变化相关。此外,启动子区域5mC和5hmC单修饰基因在细胞增殖、细胞分化和受体酪氨酸激酶通路等信号通路中富集,这些通路对肿瘤发生至关重要。最后,具有5mC和5hmC启动子单修饰的前20个基因分组表达特征往往与某些亚型头颈部鳞状细胞癌的临床结局相关。
研究结果表明使用平行WGBS和oxWGBS分析揭示了OSCC不同基因组区域的5mC和5hmC修饰整体减少。启动子区域5mC和5hmC的单修饰与OSCC组织中基因表达的增强变化有关。此外这种在启动子区域的5mC和5hmC单修饰差异表达基因可能与OSCC的临床结局具有相关性。
结果图形:
(1)5mC和5hmC在全基因组中的分布
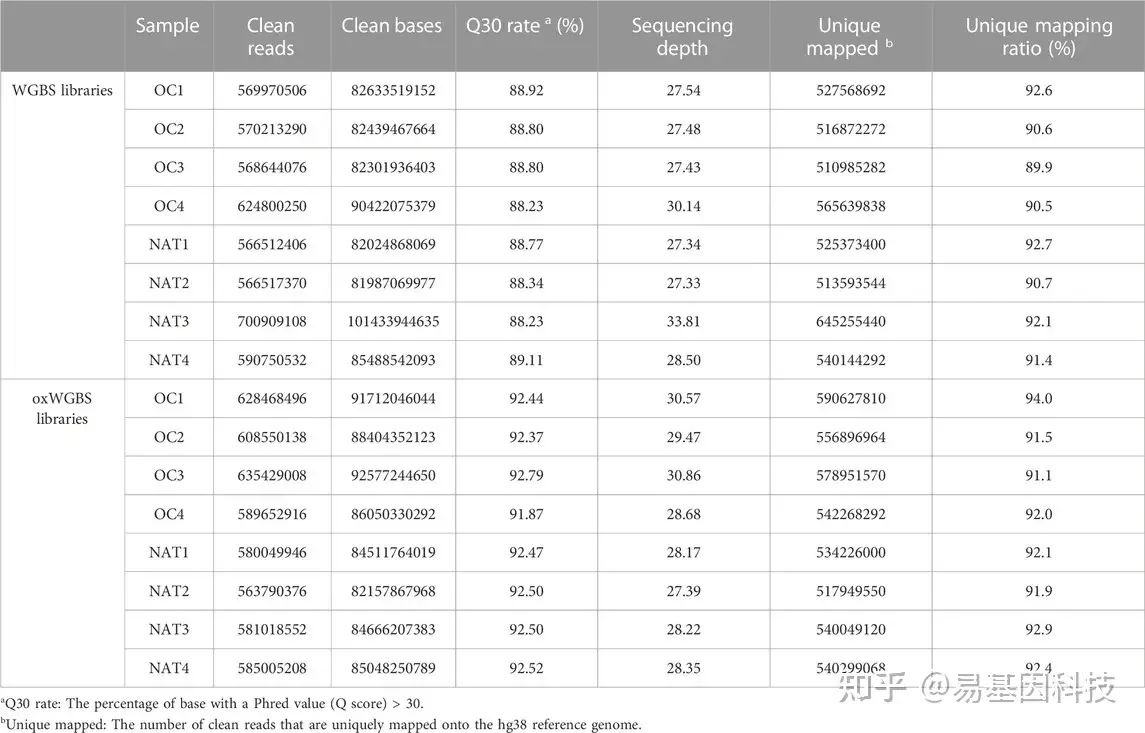
表1:WGBS和oxWGBS文库测序概况
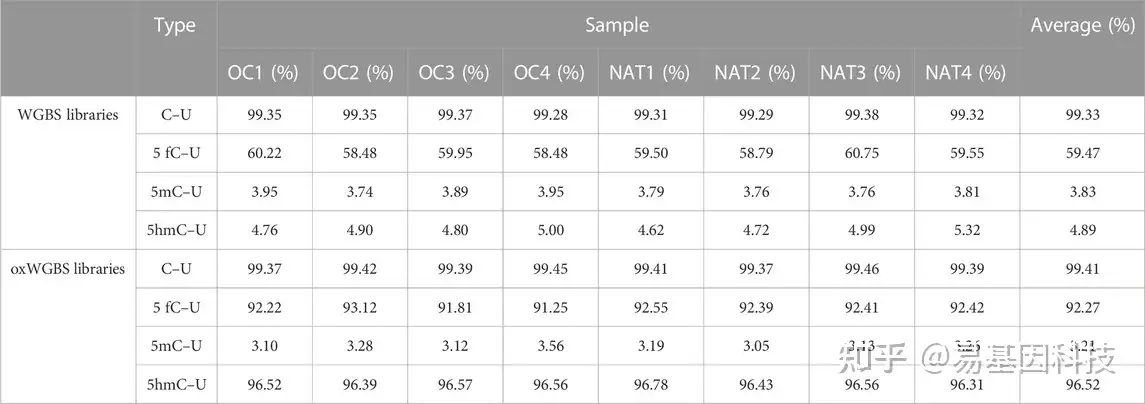
表2:WGBS和oxWGBS文库中的亚硫酸盐和氧化亚硫酸盐转化率
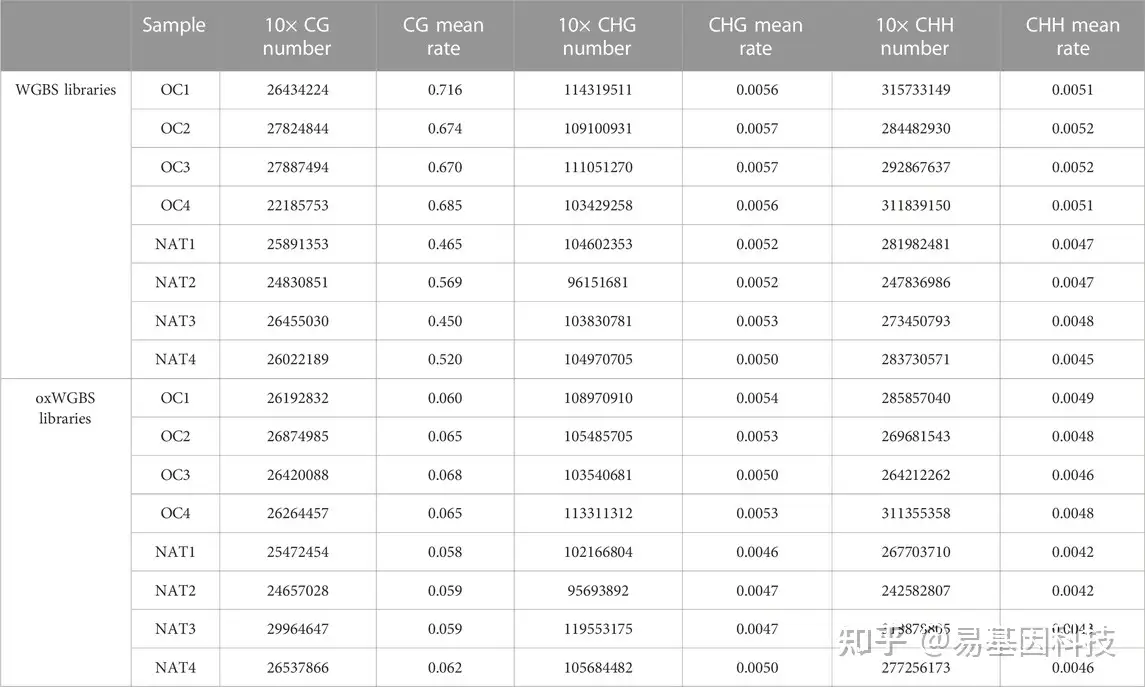
表3:WGBS和oxWGBS文库中不同胞嘧啶序列数量和平均甲基化率。
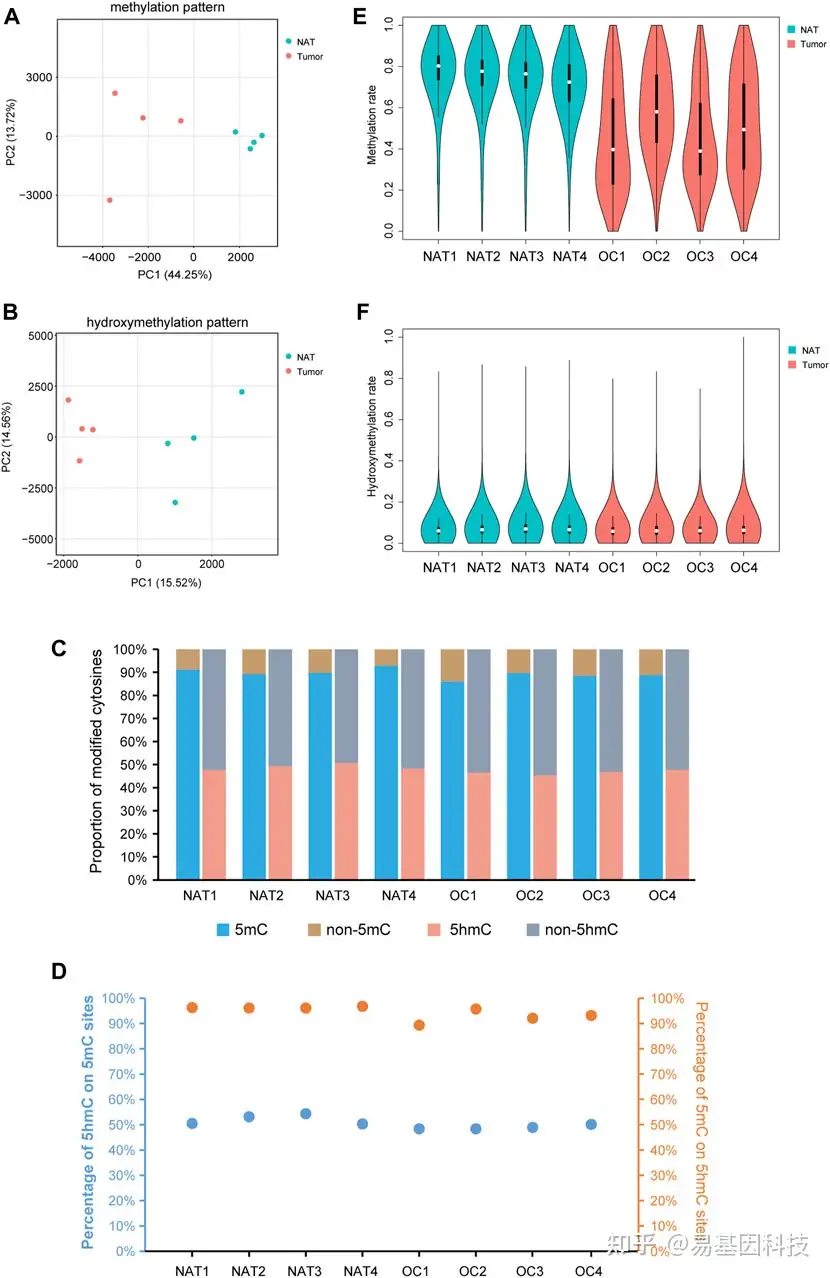
图1:OSCC和NAT样本中DNA甲基化和羟甲基化图谱
- PCA图显示OSCC组织和癌旁组织NAT的DNA甲基化模式的不同特征。每个点表示一个样本。
- PCA图显示OSCC组织和NAT的DNA羟甲基化模式的不同特征。每个点表示一个样本。
- 柱状图显示不同样本中甲基化或羟甲基化胞嘧啶比例。
- 5hmC修饰位点中5hmC百分比和5hmC修饰位点中5mC百分比。
- 小提琴图显示OSCC和NAT样本的整体甲基化水平。
- 小提琴图显示OSCC和NAT样本的整体羟甲基化水平。
OSCC:口腔鳞状细胞癌。NAT:正常癌旁组织。5mc: 5-methylcytosine。5 hmc: 5-hydroxymethylcytosine。
(2)5mC和5hmC在基因组区域中的分布
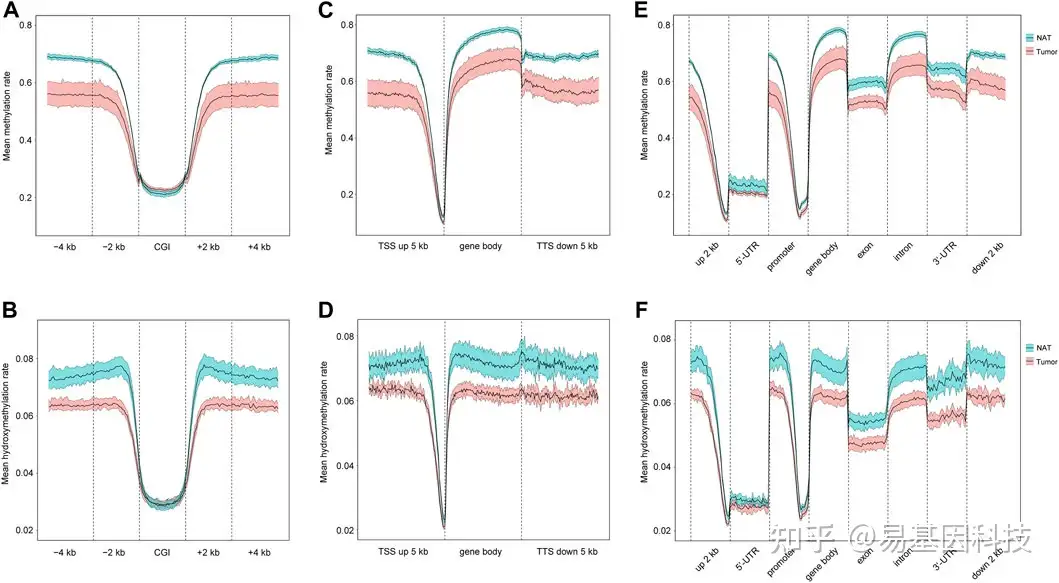
图2:5mC和5hmC在基因调控元件中的分布。
- CGI、CGI±2 kb区域和CGI±4 kb区域的平均甲基化率。
- CGI、CGI±2 kb区域和CGI±4 kb区域的平均羟甲基化率。
- Genebody、上下游±5 kb TSS的平均甲基化率。
- Genebody、上下游±5 kb TSS的平均羟甲基化率。
- 5'-UTR上游2 kb、5'-UTR、启动子、基因体、外显子、内含子、3'-UTR、3'-UTR下游2 kb区域的5mC平均甲基化率。
- 5'-UTR上游2 kb、5'-UTR、启动子、基因体、外显子、内含子、3'-UTR、3'-UTR下游2 kb的平均羟甲基化率。
蓝色和红色的均数线分别代表NAT和肿瘤样本的95%置信区间。CGIs:CpG岛。TSSs:转录起始位点。TTS:转录终止位点。5′-UTR:5′-非翻译区。3′-UTR:3′-非翻译区。
(3)启动子DMRs和DhMR鉴定
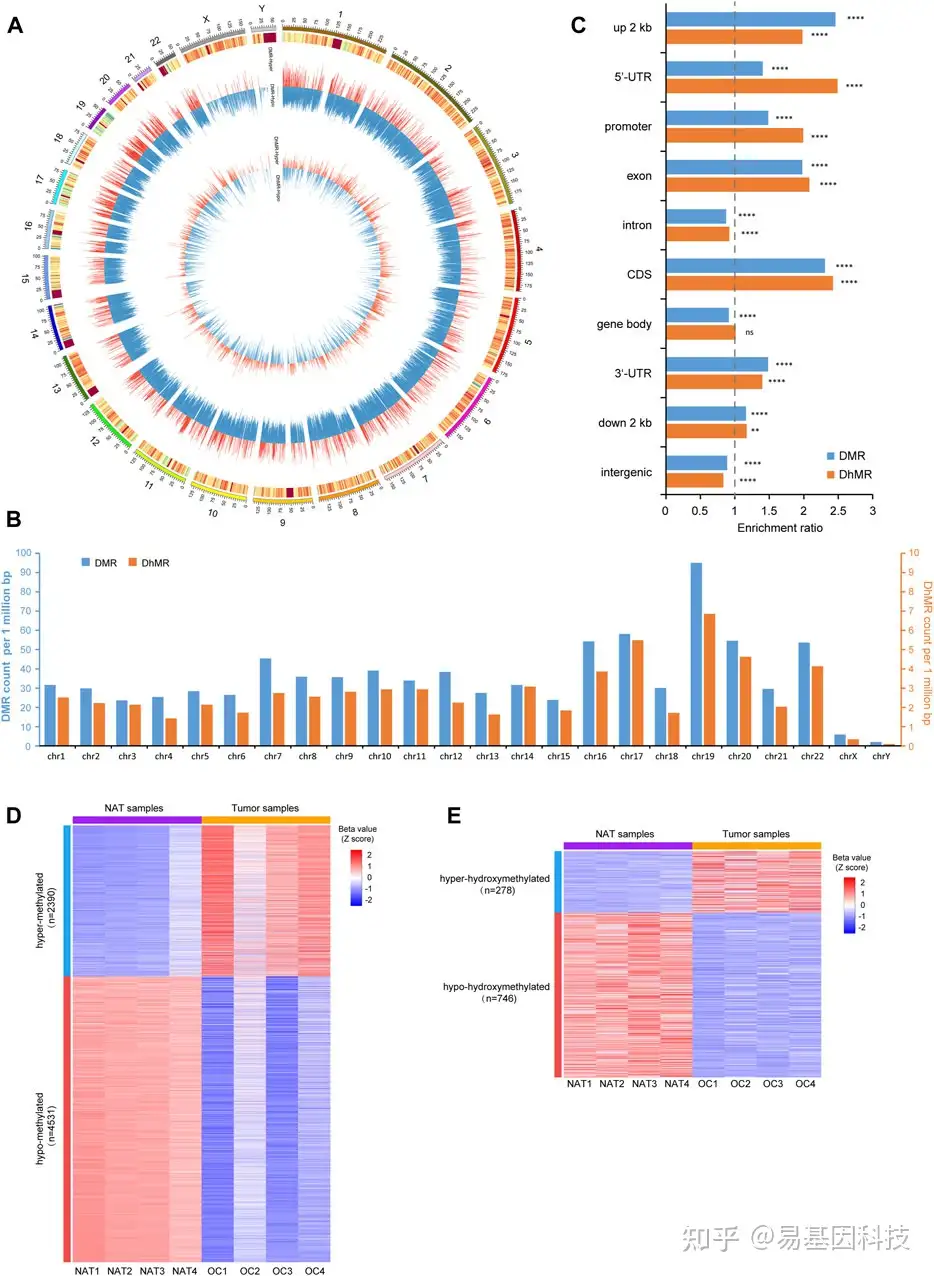
图3:p-DMRs和p-DhMRs的鉴定
- Circos图显示DMRs和DhMRs在所有染色体上的基因组分布。中间和内圈内的单独条分别代表一个DMR或一个DhMR。每个条形图的高度表示甲基化差异程度(Δβ值),红色条形图表示高甲基化,蓝色条形图表示低甲基化。
- 条形图显示DMRs和DhMRs在每条染色体上的分布。y轴表示每M碱基对中的DMRs/DhMRs数。
- 条形图显示DMRs和DhMRs在每个基因组特征中的遗传分布。y轴表示特定的基因组区域,x轴表示DMRs/DhMRs的富集比。
- 热图表示NAT和OSCC样本中p-DMRs (β值的Z评分)水平,包括2390个高甲基化和4531个低甲基化p-DMRs。
- 热图表示NAT和肿瘤样本中p-DhMRs (β值的Z评分)水平,其中278个高羟甲基化和746个低羟甲基化p-DhMRs
NAT:正常邻近组织。OSCC:口腔鳞状细胞癌。DMR:差异甲基化区域。DhMRs:差异羟甲基化区域。p-DMRs:启动子DMRs。p-DhMRs:启动子DhMRs。**p < .01。****p < .0001。
(4)甲基化、羟甲基化与基因表达的综合分析
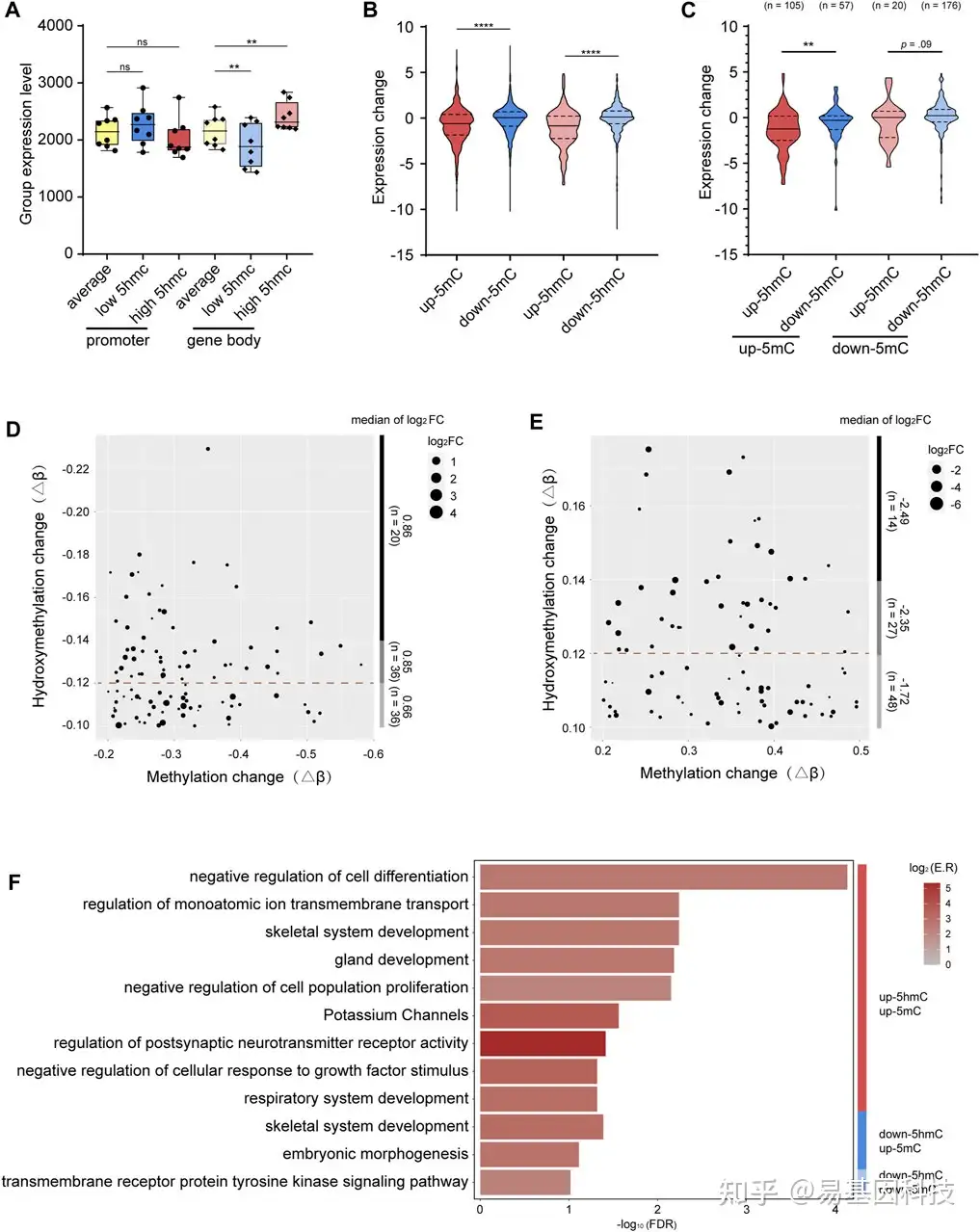
图4:5mC、5hmC与基因表达的综合分析
- 将5hmC在启动子(标准色)或基因体(颜色较浅)区域的平均水平(黄色)、低水平(蓝色)和高水平(红色)的基因表达水平(每组的平均标准化计数)分组。
- 高甲基化p-DMR(红色)、低甲基化p-DMR(蓝色)、高羟甲基化p-DhMR(浅红色)、低羟甲基化p-DhMR(浅蓝色)基因表达变化分组。
- 启动子区5mc和5hmc变化不同组合对基因表达的分组变化。up-5mC5mc /up- 5hmc(红色),up- 5mc /down-5hmc(蓝色),down-5mc /up- 5hmc(浅红色),down- 5mc /down- 5hmc(浅蓝色)。
- 点图显示启动子区单down-5mC/down-5hmC的基因上调水平与down-5hmC水平的协调关系。
- 点图显示启动子区单up-5mC/up-5hmC的基因下调水平与up-5hmC水平的协调关系。
- 条形图显示启动子区5mC和5hmC单变化或共变化的基因功能簇。
-log10 (FDR)表示-log10转换后的FDR,色条表示log2(富集率),红色表示表达水平较高,灰色表示表达水平较低。p-DMRs:启动子DMRs。p-DhMRs:启动子DhMRs。Up-5mC:hyper-methylated。Down-5mC:hypo-methylated。Up-5hmC: hyper-hydroxymethylated。Down-5hmC: hypo-hydroxymethylated。Log2FC: log2 fold change。**p < .01。****p < .0001。
(5)启动子5mC和5hmC单修饰OSCC差异表达基因的功能分析

图5:启动子5mC和5hmC单向修饰基因与HNSC的临床相关性。
A-B. 方框图显示四种亚型的HNSC肿瘤和匹配的正常组织(TCGA“实体组织正常”样本的HNSC患者)中,启动子处down-5mC/down-5hmC的前20个上调基因(A)和启动子处up-5mC/up-5hmC的前20个下调基因(B)的分组表达水平。分组表达水平定义为各组平均log2 (TPM + 1)。
C–D. Kaplan-Meier曲线显示了上述前20个上调基因标签(C)和前20个下调基因标签(D)
E-F. 低或高评分的HNSC患者的OS曲线。Kaplan-Meier曲线显示了4种HNSC病理亚型中,上述前20个上调基因(E)和上述前20个下调基因(F)评分低或高的各亚型HNSC患者的OS曲线。
蓝色线:低评分。红线:高评分。HNSC:头颈部鳞状细胞癌。TPM:transcript per million。OS:总生存期。
研究小结:
本研究首次提供了OSCC中5mC和5hmC的全基因组、单碱基分辨率、平行图谱。使用整合多组学方法,揭示了OSCC样品中启动子区域的5mC和5hmC单向修饰基因的表达变化上调。此外,具有启动子5mC和5hmC单向修饰的此类差异调控基因可能与OSCC的临床结局具有功能相关性。
关于易基因精准DNA甲基化/羟甲基化测序(oxBS-seq)
羟甲基化5hmC是哺乳动物基因组上的第六碱基,在发育、衰老、神经退行性疾病、复杂疾病及肿瘤发生过程中起重要作用。DNA羟甲基化是近年发现的一种新的DNA修饰并迅速成为研究热点。随着研究的深入,发现之前被认为是检测DNA甲基化标准的重亚硫酸盐测序并不能区分DNA甲基化(5mC)和DNA羟甲基化(5hmC)。
易基因联合剑桥大学建立了化学氧化法结合重亚硫酸盐转化的测序技术(oxidative bisulfite sequencing, oxBS-Seq),该技术不仅可以精确检测DNA甲基化,排除DNA羟甲基化的影响,还可以双文库结合同时单碱基分辨率精确检测DNA羟甲基化。
传统BS转化无法区分5mC和5hmC
传统的Bisulfite测序中,5hmC经过Bisulfite处理后变为CMS,CMS在测序中仍然被读作C碱基,因此不能区分5mC和5hmC。
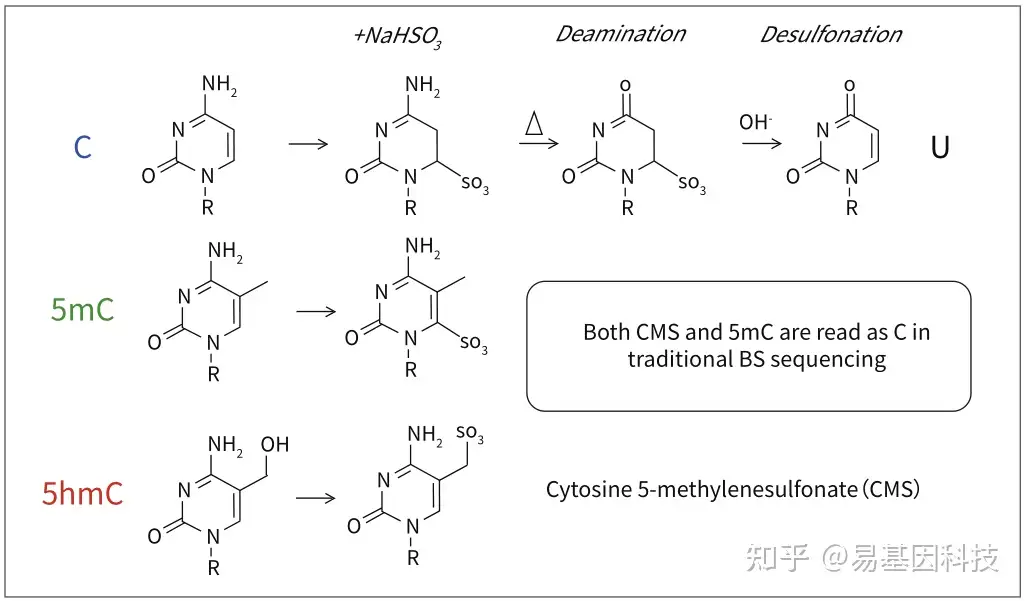
oxBS技术原理
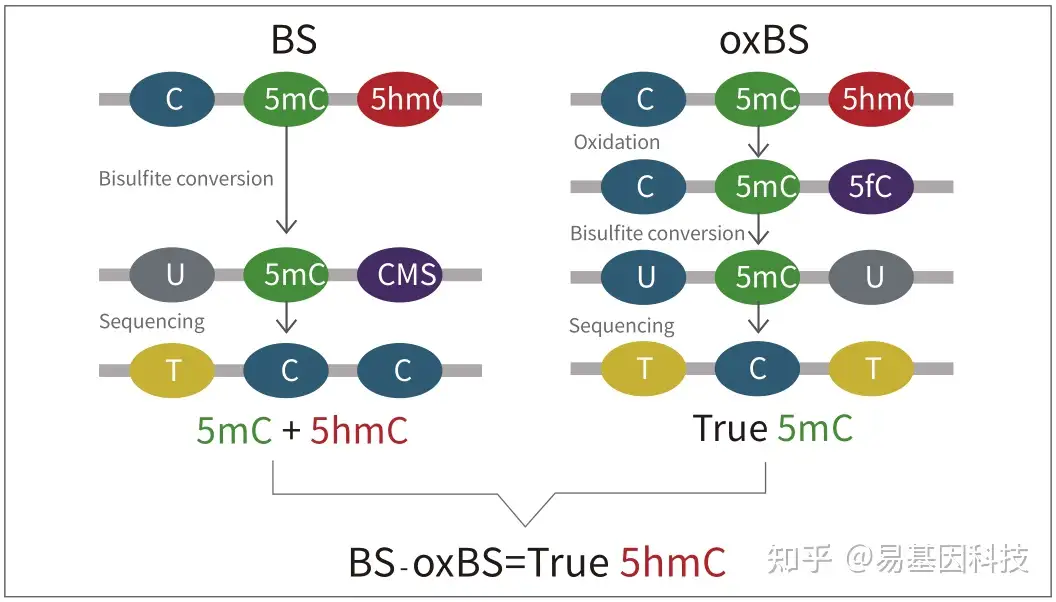
技术优势:
- DNA甲基化检测全新的“标准”;
- 单碱基检测DNA羟甲基化修饰;
- 多重质控标准检测氧化效率和Bisulfite转换率;
- 实验偏好性低,重复性高(R2>0.98);
- 易基因自主研发的甲基化特异性多重PCR引物设计软件;
- 可满足多种测序应用需求:
- 全基因组氧化甲基化测序(oxWGBS)
- 简化基因组氧化甲基化测序(oxRRBS)
- 目标区域靶基因氧化甲基化测序(Target-oxBS)。
技术路线:
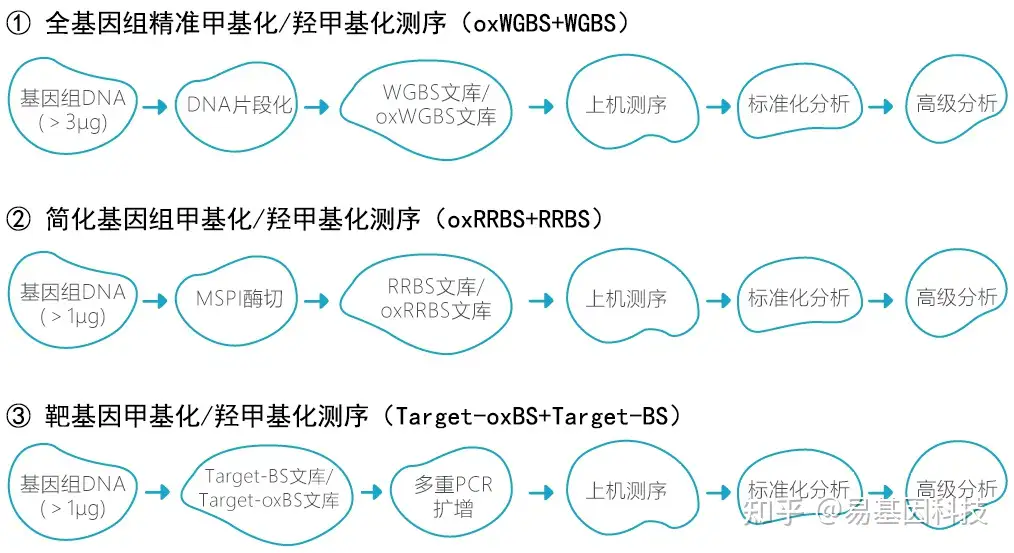
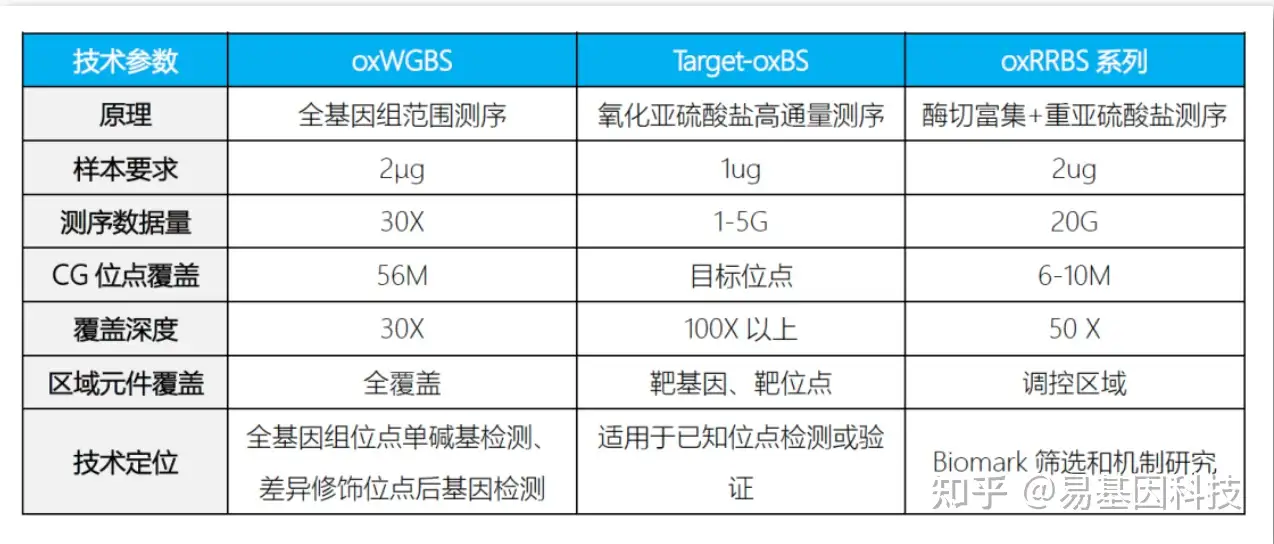
技术指标:
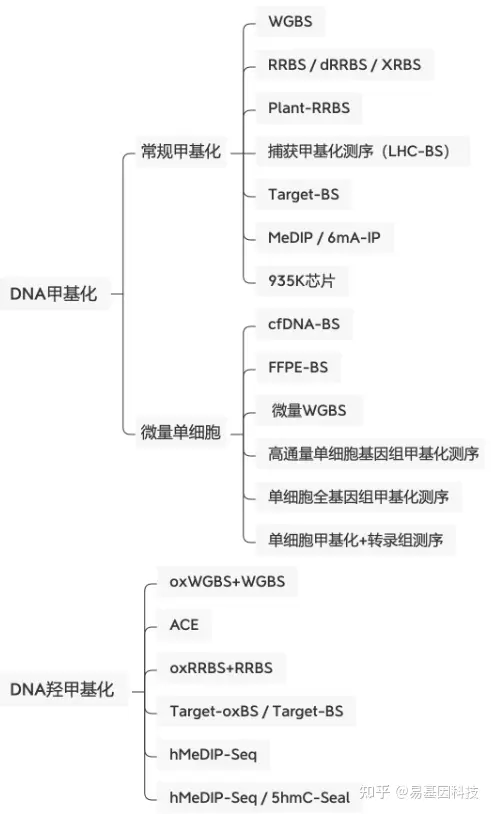
易基因科技提供全面的DNA甲基化研究整体解决方案,详询易基因:0755-28317900。
参考文献:
Zhao W, Zhu L, Gong Q, Ma S, Xiong H, Su T, Wan Z, Wang D. Unidirectional alteration of methylation and hydroxymethylation at the promoters and differential gene expression in oral squamous cell carcinoma. Front Genet. 2023;14:1269084. pii: 1269084. doi: 10.3389/fgene.2023.1269084. PubMed PMID: 37900177.
相关阅读:
项目文章:oxBS揭示复发性膀胱癌的DNA甲基化和羟甲基化变化并鉴定预测PD-L1表达标记物
项目文章 | oxBS-seq揭示了宫颈癌发生过程中的表观遗传特征变化
亮点研究|oxRRBS+RRBS揭示牦牛下丘脑在神经调节和髓鞘形成中的表观调控机制
技术推介 | 精准DNA甲基化/羟甲基化测序(oxBS-seq)
标签:OSCC,基因,甲基化,鳞癌,5mC,启动子,5hmC From: https://www.cnblogs.com/E-GENE/p/17846302.html