大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
肾上腺皮质癌(Adrenocortical carcinoma,ACC)是一种罕见的侵袭性肾上腺内分泌癌。在肾上腺皮质癌中,最近研究表明以CpG岛甲基化表型(CpG island methylator phenotype,CIMP)为特征的亚型与特别差的预后相关。然而CIMP的驱动因素仍然未知。此外,CIMP与肾上腺皮质癌患者临床预后不良之间的功能关系尚不明确。
2023年8月2日,法国巴黎大学Valentina Boeva团队在《Clinical Epigenetics》杂志发表题为“DNA hypermethylation driven by DNMT1 and DNMT3A favors tumor immune escape contributing to the aggressiveness of adrenocortical carcinoma”的研究论文,该研究通过RRBS、RNA-seq等组学研究揭示了由DNMT1和DNMT3A驱动的DNA高甲基化有利于肿瘤免疫逃逸,从而导致肾上腺皮质癌侵袭。

标题:DNA hypermethylation driven by DNMT1 and DNMT3A favors tumor immune escape contributing to the aggressiveness of adrenocortical carcinoma(由DNMT1和DNMT3A驱动的DNA高甲基化有利于肿瘤免疫逃逸,从而导致肾上腺皮质癌的侵袭性)
时间:2023-08-02
期刊:Clinical Epigenetics(临床表观遗传学)
影响因子:IF 5.7 / 1区
技术平台: RNA-seq、RRBS等
研究摘要:
本研究通过RRBS、RNA-seq等分析结果表明肾上腺皮质癌(ACC)中的CIMP与DNA甲基转移酶DNMT1和DNMT3A高表达有关,而这主要由基因拷贝数的增加和细胞过度增殖驱动。此外,本研究验证了CIMP通过促进肿瘤免疫逃逸以促进肿瘤的侵袭性,而通过用去甲基化剂5-氮杂胞苷(5-azacytidine)处理可以至少部分逆转这种效应。
本研究结果表明,与去甲基化药物联合治疗可能会提高免疫治疗的疗效,并可能为高CIMP肾上腺皮质癌患者提供一种新的治疗方法。
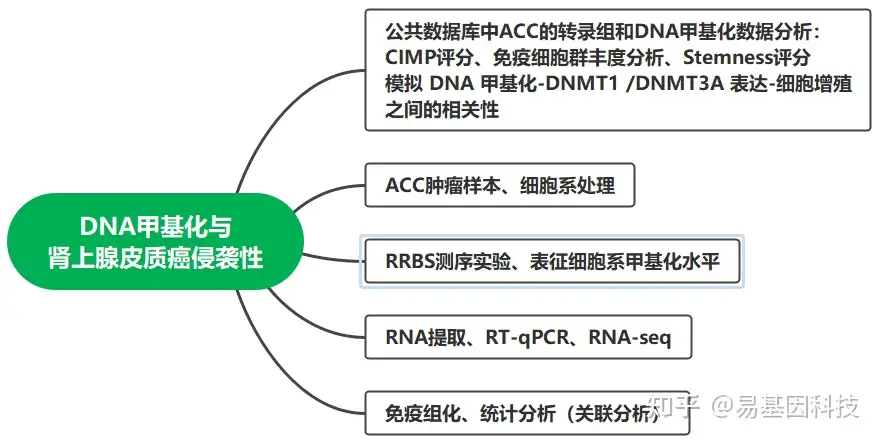
研究结果:
(1)ACC中的CIMP与DNMT1和DNMT3A高表达相关
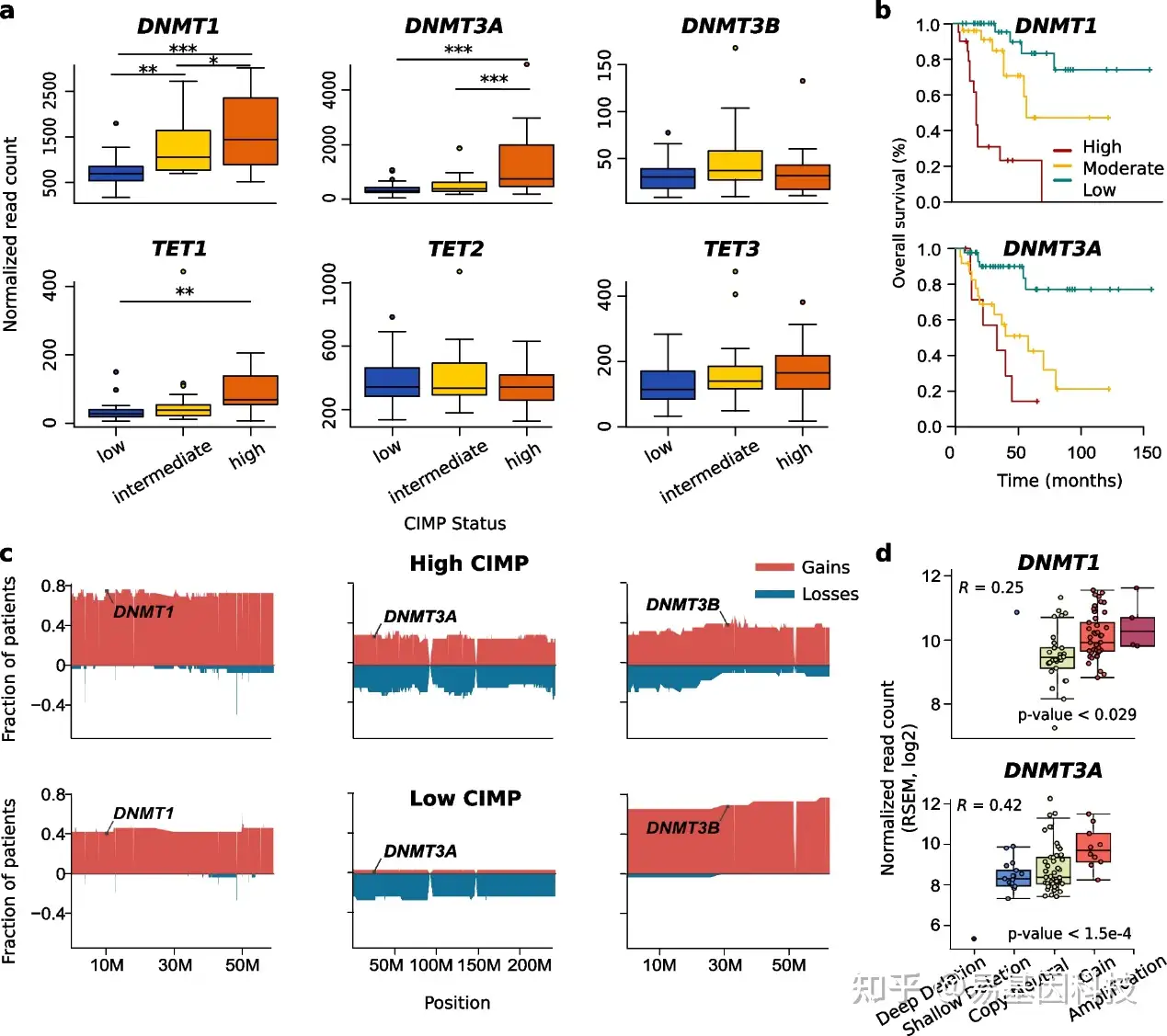
图1:DNMT1和DNMT3A的表达水平在hCIMP-ACC中上调,并与拷贝数增加相关。
- DNMT1、DNMT3A、DNMT3B、TET1、TET2和TET3在TCGA数据集患者中lCIMP (n = 32)、iCIMP (n = 26)、hCIMP (n = 21)的表达水平(双侧t检验,***p<0.005)。
- 根据TCGA数据集的DNMT1或DNMT3A表达(分为高、中、低),利用Kaplan–Meier分析ACC患者的总生存率。
- 在含有DNMT1、DNMT3A和DNMT3B基因的染色体中hCIMP-ACC和lCIMP-ACC的拷贝数变化模式。
- TCGA患者DNMT1和DNMT3A基因拷贝数状态的表达水平。
(2)拷贝数异常和增殖增加共同导致了ACC中DNMT高表达
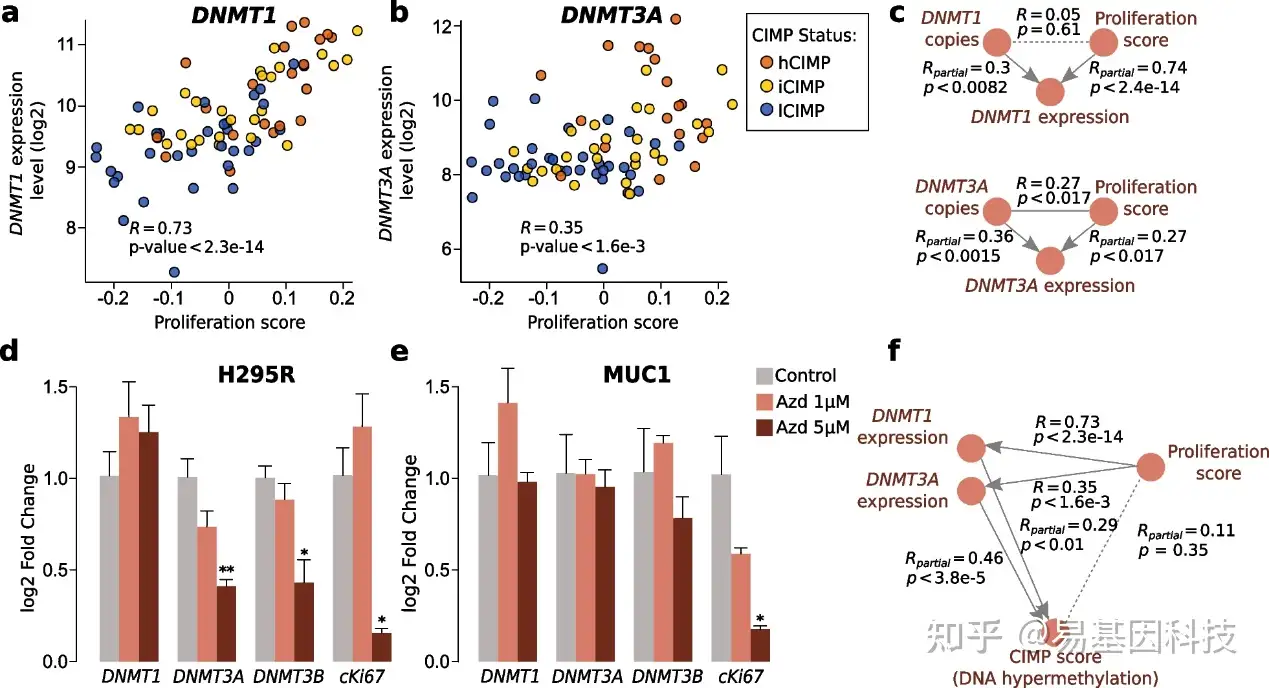
图2:hCIMP-ACC肿瘤中细胞增殖增加可能是DNMT1和DNMT3A表达上调以及DNMT基因拷贝数畸变的第二驱动因素。
a-b. DNMT1(a)或DNMT3A(b)表达与细胞增殖之间的相关性;hCIMP样本(n=21,橙色),iCIMP(n=26,黄色)和lCIMP(n = 32,蓝色)。
c. 使用TCGA数据集的偏相关分析对DNMT基因的基因组拷贝数状态、细胞增殖和基因表达之间的相互依赖性进行建模。Rpartial表示在调控第三因素时计算的Pearson偏相关系数。
d-e. 分别用1µM或5µM的增殖抑制剂AZD-5438(AZD)处理hCIMP-ACC和lCIMP-ACC细胞系H295R(d)和MUC-1(e)48h后,分析对DNMT1、DNMT3A、DNMT3B和增殖标记物MKI67表达的影响。使用DMSO作为载体。数据表示4个生物重复的平均值±SEM(Wilcoxon检验,*p<0.05,**p<0.01)。
(3)高CIMP和低CIMP肿瘤在增殖和免疫反应基因表达特征方面存在差异
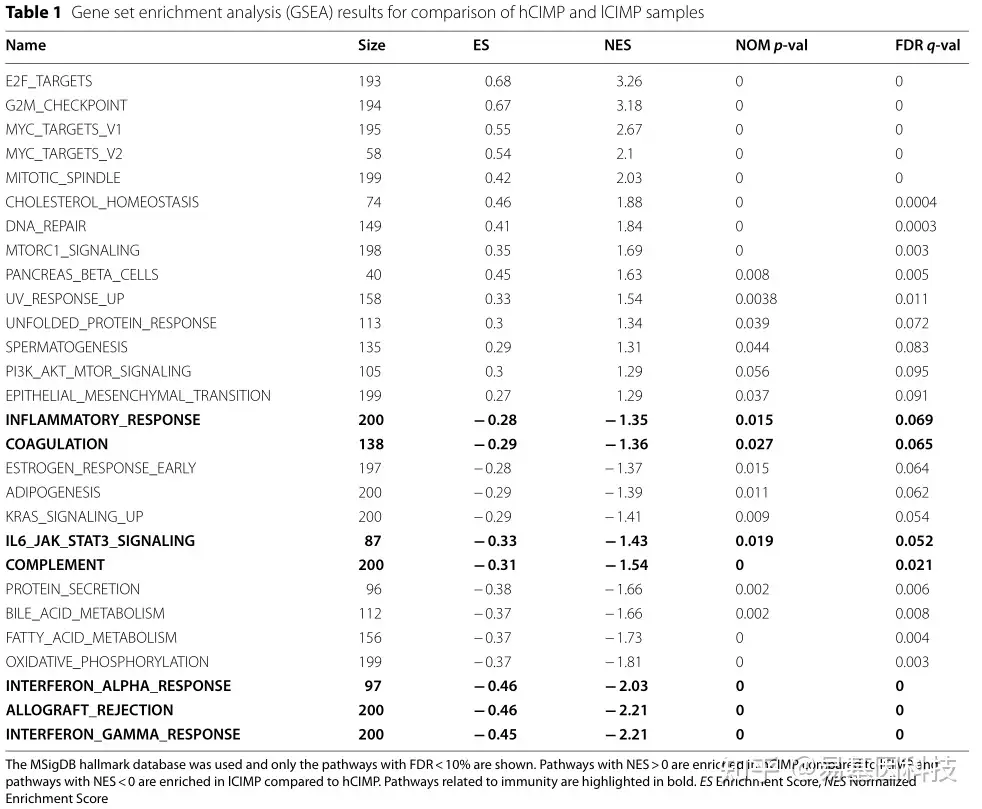
表1:hCIMP和lCIMP样本的基因集富集分析(GSEA)结果比较
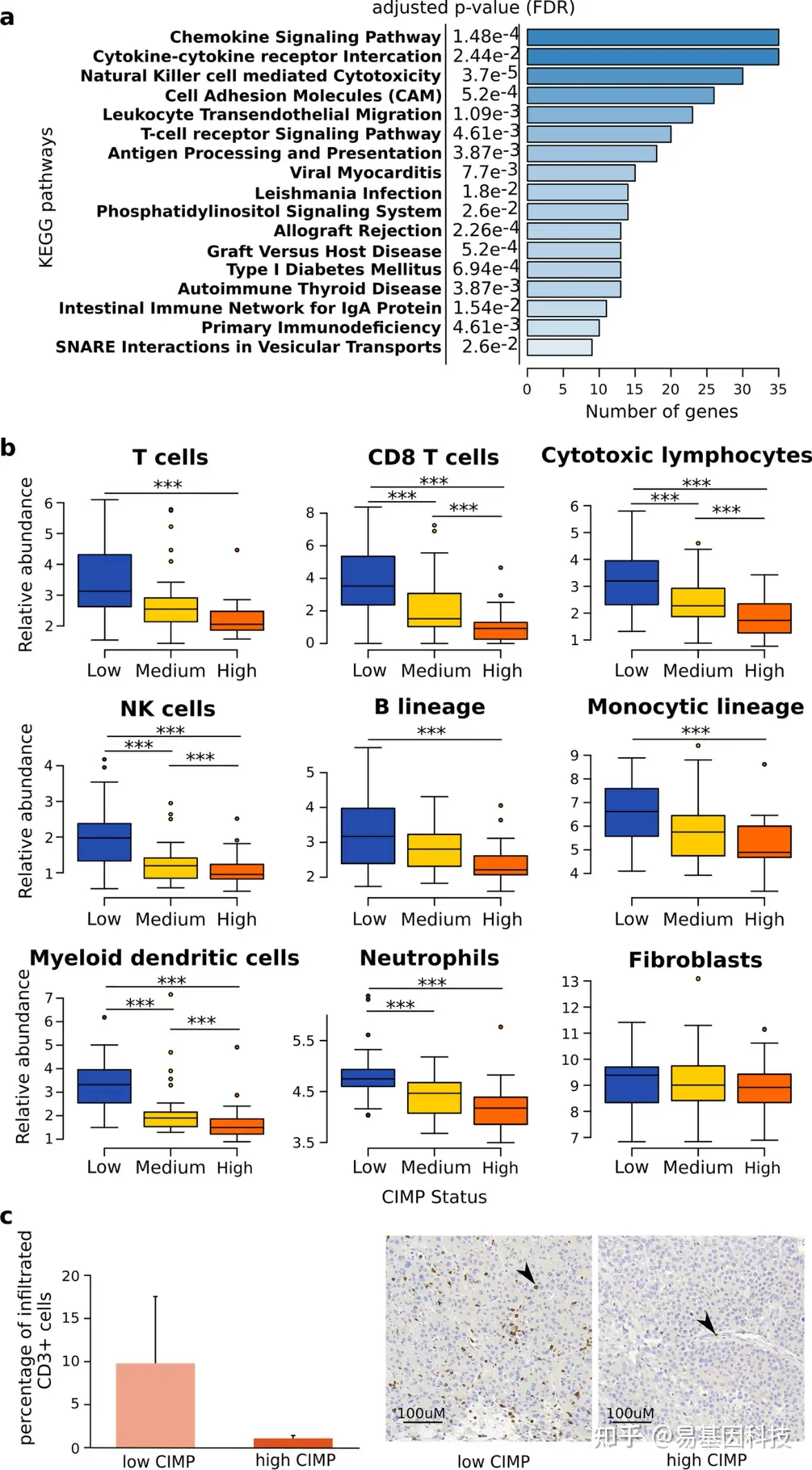
图3:hCIMP-ACC肿瘤的特征是肿瘤浸润免疫细胞丰度较低。
- 对TCGA数据集中hCIMP肿瘤中DNA甲基化抑制基因的通路富集分析。使用完整的KEGG通路数据集分析,图中只描述显著富集的通路。
- 在TCGA数据集的lCIMP (n=32), iCIMP (n=26)和hCIMP (n = 21)样本中,使用mcp计数器计算肿瘤浸润的免疫和非免疫基质细胞群的相对丰度。使用Kruskal-Wallis检验比较每个种群的相对丰度,然后使用Benjamini-Hochberg校正的Dunn检验。***p < 0.001。
- 左:肿瘤浸润CD3+ T细胞百分比的定量。数据来自至少6个生物重复的平均值±SEM。Mann-Whitney U检验p=0.12。右:高浸润lCIMP样本和低浸润hCIMP样本的代表性截图
(4)用DNA去甲基化剂处理hCIMP-ACC细胞系可重新激活与免疫系统相关的基因
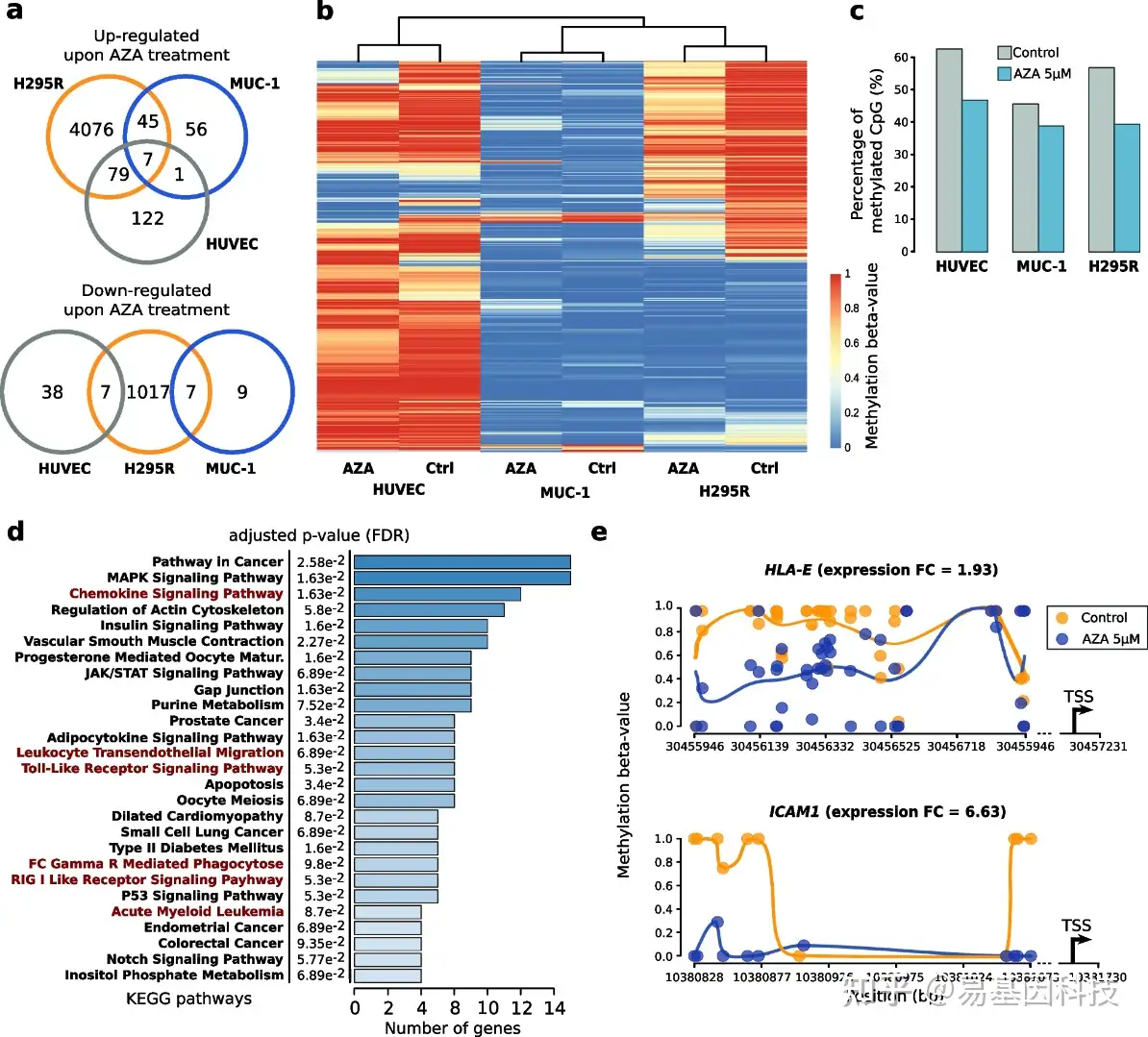
图4:去甲基化药物5-氮杂胞苷(5-azacytidine, AZA)可以部分恢复hCIMP-ACC细胞系H295R中CpG岛的DNA高甲基化,恢复免疫应答基因活性。
- AZA处理后,H295R(5242个基因)、MUC-1(125个基因)和HUVEC(254个基因)细胞系中,存在显著上调或下调的基因重叠(FDR < 0.05)。
- 经5 μM AZA处理或未处理的3种细胞系(H295R, MUC-1和HUVEC)的启动子CpG岛甲基化情况,进行无监督分层聚类。使用甲基化水平变化最大的CpG (s.d. > 0.4)进行聚类分析。
- 在RRBS reads覆盖的CpG中甲基化CpG的整体百分比。AZA诱导三种细胞系CpG甲基化水平降低,但这种作用在DNA甲基化基础水平最低的MUC-1细胞系中不太明显。
- AZA诱导H295R基因启动子去甲基化的通路富集分析。与免疫应答相关的通路用红色突出显示。
- 位于两个免疫应答基因启动子H295R的差异甲基化区域(DMRs)示例。
研究小结:
本研究结果阐明DNMT1和DNMT3A高表达是基因拷贝数增加和细胞过度增殖的结果,构建了ACC中CIMP原因和结果的表型机制研究。此外,研究揭示了CIMP通过促进肿瘤免疫逃逸对肿瘤的侵袭性产生直接影响(图5)。且通过去甲基化剂AZA处理,这一作用可以至少部分逆转。总之,本研究结果强烈表明,DNA去甲基化药物和免疫治疗药物联合治疗hCIMP-ACC患者可能对患者生存产生积极影响。
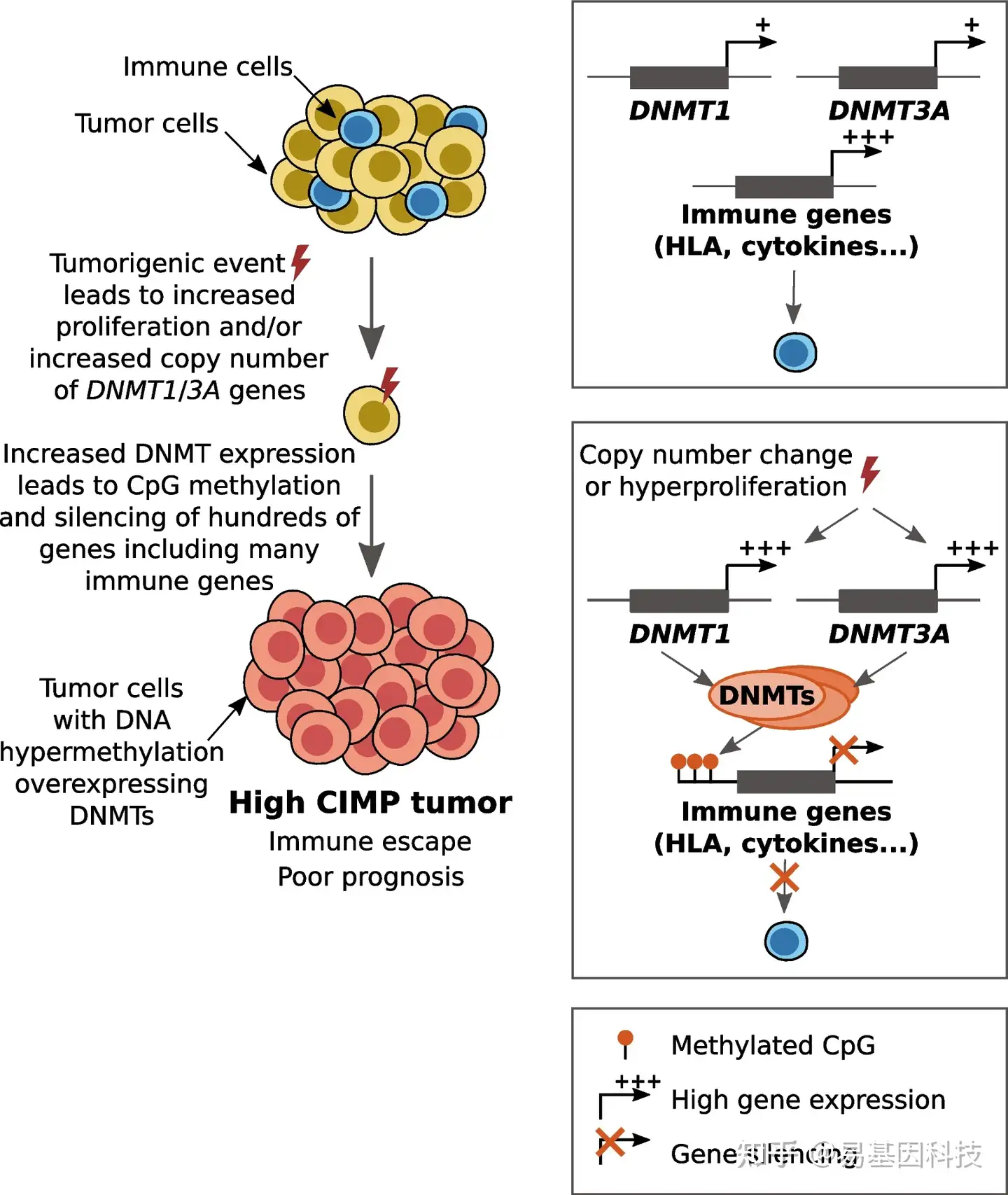
图5:本研究提出了ACC高CIMP(hCIMP)的建立模型。DNA甲基转移酶DNMT1和DNMT3A在基因组畸变和过度增殖的驱动下表达增加,导致数百个基因启动子区DNA甲基化增加;因此,免疫应答相关基因的甲基化依赖性沉默导致免疫细胞对肿瘤浸润减少。
关于易基因简化基因组甲基化测序(RRBS)研究解决方案
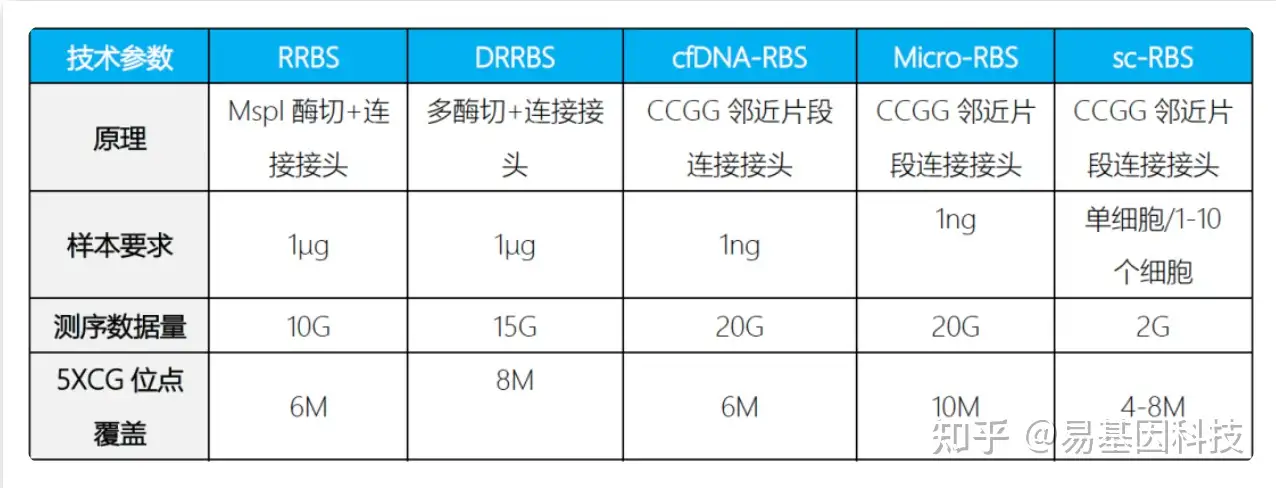
简化甲基化测序(Reduced Representation Bisulfite Sequencing,RRBS)是利用限制性内切酶对基因组进行酶切,富集启动子及CpG岛等重要的表观调控区域并进行重亚硫酸盐测序。该技术显著提高了高CpG区域的测序深度,在CpG岛、启动子区域和增强子元件区域可以获得高精度的分辨率,是一种准确、高效、经济的DNA甲基化研究方法,在大规模临床样本的研究中具有广泛的应用前景。
为适应科研技术的需要,易基因进一步开发了可在更大区域内捕获CpG位点的双酶切RRBS(dRRBS),可研究更广泛区域的甲基化,包括CGI shore等区域。
为助力适用低起始量DNA样本(5ng)量多维度甲基化分析,易基因开发了富集覆盖CpG岛、启动子、增强子、CTCF结合位点的甲基化靶向基因组测序方法:extended-representation bisulfite sequencing(XRBS),实现了高灵敏度和微量样本复用检测,使其具有高度可扩展性,并适用于有限的样本和单个细胞基因组CG位点覆盖高达15M以上。
技术优势:
- 起始量:100ng gDNA;
- 单碱基分辨率;
- 多样本的覆盖区域重复性可达到85%-95%、测序区域针对高CpG调控区域,数据利用率更高;
- 针对性强,成本较低;
- 基因组CG位点覆盖高达10-15M,显著优于850K芯片。
应用方向:
RRBS/dRRBS/XRBS广泛应用于动物,要求全基因组扫描(覆盖关键调控位点)的:
- 队列研究、疾病分子分型、临床样本的甲基化 Biomarker 筛选
- 复杂疾病及肿瘤发病机制等甲基化研究
- 模式动物发育和疾病甲基化研究
易基因提供全面的表观基因组学和表观转录组学、DNA与蛋白互作测序方案,详询易基因:0755-28317900.

参考文献:
Kerdivel G, Amrouche F, Calmejane MA, Carallis F, Hamroune J, Hantel C, Bertherat J, Assié G, Boeva V. DNA hypermethylation driven by DNMT1 and DNMT3A favors tumor immune escape contributing to the aggressiveness of adrenocortical carcinoma. Clin Epigenetics. 2023 Aug 2;15(1):121.
相关阅读:
项目文章 | RRBS揭示基于DNA甲基化驱动基因的肾透明细胞癌预后模型的鉴定和验证
一文读懂|精准简化基因组甲基化测序(RRBS+oxRRBS)分析怎么做
3文一览:简化甲基化测序(RRBS)技术优势及研究成果(医学+物种保护+农学)
标签:ACC,DNMT3A,DNA,DNMT1,基因,甲基化,RRBS From: https://www.cnblogs.com/E-GENE/p/17805457.html