大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
2023年3月23日,海南大学三亚南繁研究院/热带作物学院博士研究生郭昊等为第一作者、王守创教授为通讯作者在《Science China Life Sciences》杂志发表题为“Population analysis reveals the roles of DNA methylation in tomato domestication and metabolic diversity”的研究论文,该研究通过对野生品种、地方品种和栽培品种的番茄群体进行全基因组重亚硫酸盐测序(WGBS)、转录组测序(RNA-seq)和代谢组学等分析,解析了番茄群体代谢多样性与育种过程中DNA甲基化变化的关系,构建了多组学关联网络并完善了番茄多酚等代谢物的合成通路。

标题:Population analysis reveals the roles of DNA methylation in tomato domestication and metabolic diversity(群体分析揭示DNA甲基化在番茄驯化和代谢多样性中的作用)
时间:2023-03-23
期刊:Sci China Life Sci
影响因子:IF 10.372
技术平台:WGBS、RNA-seq、代谢组学分析等
研究摘要:
DNA甲基化是一种重要的表观遗传学标记,但其多样性和其在群体水平上番茄育种中的作用在很大程度上仍未知。本研究对包括野生品种、地方品种和栽培品种番茄在内的群体进行了全基因组重亚硫酸盐测序(WGBS)、RNA-seq和代谢组学分析,共鉴定出8375个差异甲基化区域(DMR),其在驯化和改良过程中的甲基化水平逐渐降低,研究结果表明20%以上的DMR与选择性清除(selective sweep)重叠。番茄中80%以上的DMR与单核苷酸多态性(single-nucleotide polymorphisms,SNP)没有显著相关性,而DMR与相邻SNP具有强关联。此外,研究还对来自364个不同种质的339种代谢物进行代谢组学分析,并进一步进行基于SNP和DMR的代谢关联研究,分别通过SNP和DMR标记检测到971和711个大效应位点。结合多组学共鉴定出13个候选基因并更新了多酚生物合成通路。本研究结果表明,DNA甲基化变化可以补充代谢物多样性的SNP图谱。总之,本研究通过WGBS测序等组学研究为不同种质绘制了全面的DNA甲基化图谱,并表明DNA甲基化变化可能是植物代谢多样性的遗传基础。
项目设计:
(1)样本选取:
野生品种(S.pimpinellifolium)、地方品种(S.lycopersicum var cerasiforme)、栽培品种(S.lycopersicum)共纳入96份不同品种番茄样本。每种品种至少20株,并在种子发芽后40天随机收集叶片快速置于液氮中,用于DNA、RNA和代谢物提取及后续测序分析。
(2)项目设计流程图:
结果图形
(1)不同番茄品种的DNA甲基化图谱
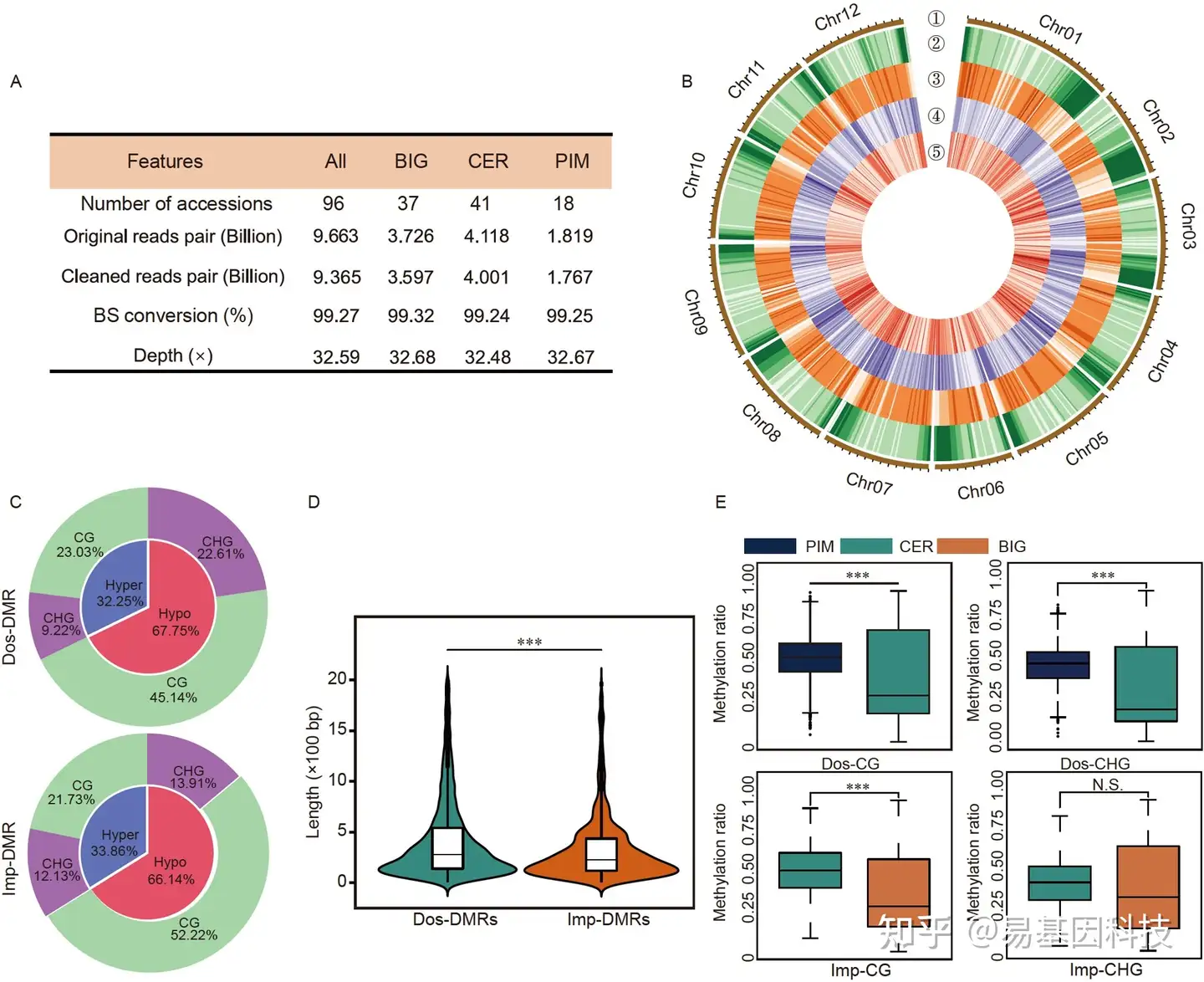
图1:驯化和改良过程中的DNA甲基化变化
- 全基因组重亚硫酸盐测序(WGBS)数据。
- DMR在番茄染色体上的分布及其与其他基因组表征的关系。从①到⑤为番茄的染色体、基因、TE、Dos DMR和Imp DMR。
- 番茄驯化和改良过程中的DMR。内圈表示Hyper-DMR和Hypo-DMR比率,外圈表示对应CG和CHG位点DMR。
- 不同育种过程和不同DMR背景下的DMR长度比较。(***P<0.001,Wilcoxon检验)
- 驯化和改良过程中群体之间的DMR信号比较。(***P<0.001,Wilcoxon检验,N.S表示不显著)
(2)DMR与选择性清除(selective sweep)重叠分析表型多样性
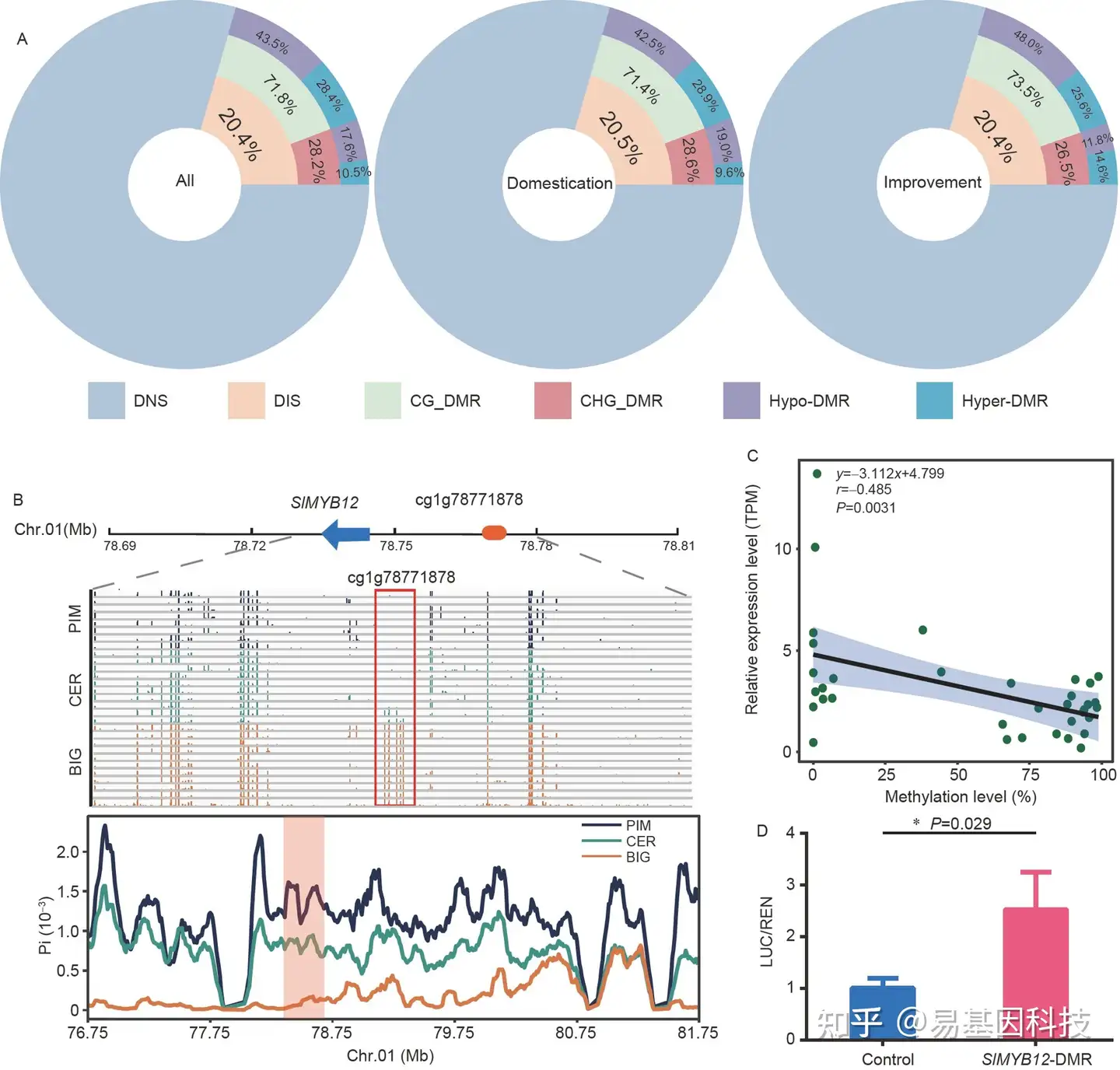
图2:差异甲基化区域(DMR)与选择性清除(selective sweep)的重叠分析。
- 驯化和改良过程中的DMR比例与选择性清除重叠。从内到外是DMR数、不同背景和不同程度的统计数据。DNS表示DMR和选择性清除不重叠,DIS表示重叠的DMR和选择性清除区域。
- 上图和中图显示了PIM、CER和BIG群体中名为cg01g78771878的DMR周围甲基化区域和水平。红色突出部分表示cg01g78771878位点。下图是位于选择性清除(selective sweep)区域的SlMYB12,BIG中的核苷酸多样性比CER或PIM中低得多。
- cg01g78771878甲基化水平与SlMYB12转录水平的关联分析。
- 三个生物学重复的LUC/REN表达比率。(误差线表示SD。t检验 *P<0.05。)
(3)DMR和SNP之间的meQTL分析
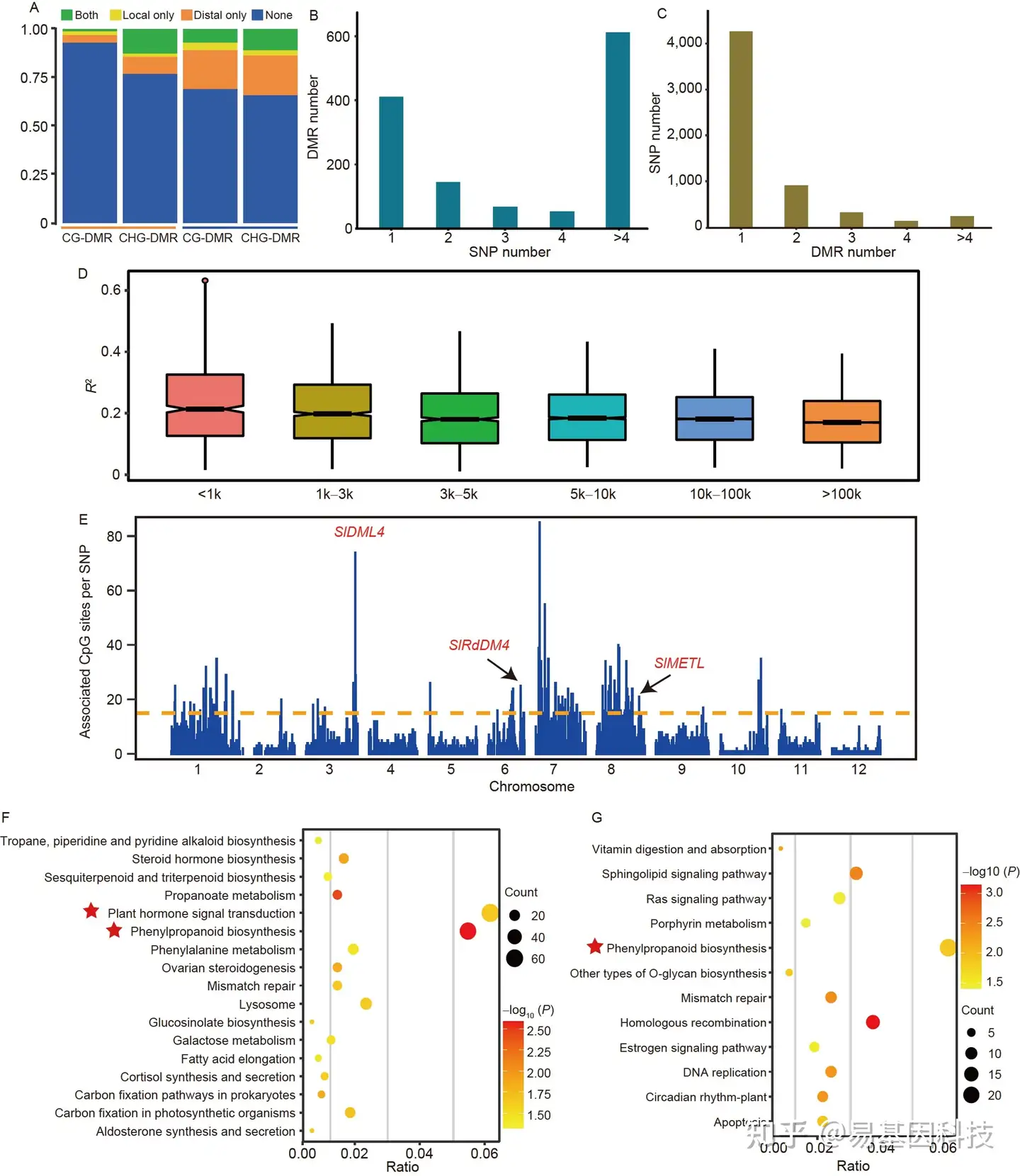
图3:DMR的遗传基础。
- 不同背景DMR的遗传基础概述。红线代表Dos-DMR,蓝线代表Imp-DMR。
- 每个DMR的重要SNP分布。
- 每个SNP标记的DMR分布。
- 不同距离DMR甲基化的LD水平。
- meQTL信号在番茄基因组中的分布。
F&G. 在CG和CHG环境中pure DMR相关基因的KEGG富集分析。
(4)构建代谢组多样性与甲基化变化关联网络
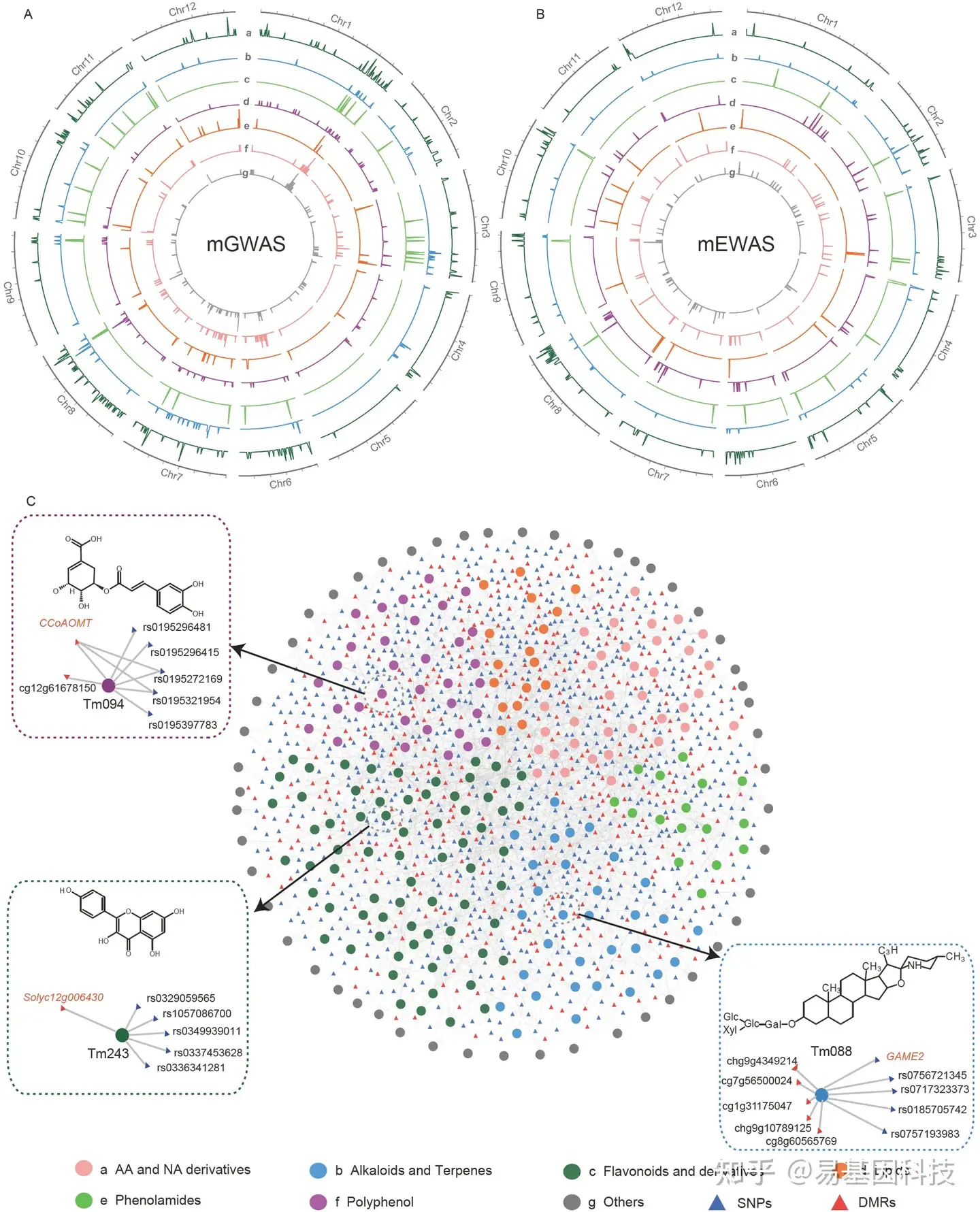
图4:多组学关联网络的整合和构建。
A&B. mEWAS和mGWAS的基因组分布。代谢物分为七类,并用不同的颜色和标签(AG)标记。
C. 代谢物、SNP和DMR之间关联分析的多组学网络。点表示与A和B中相似颜色的代谢物。SNP和DMR分别显示为小的红色和蓝色三角形。
(5)SNP和DMR解析番茄的代谢多样性
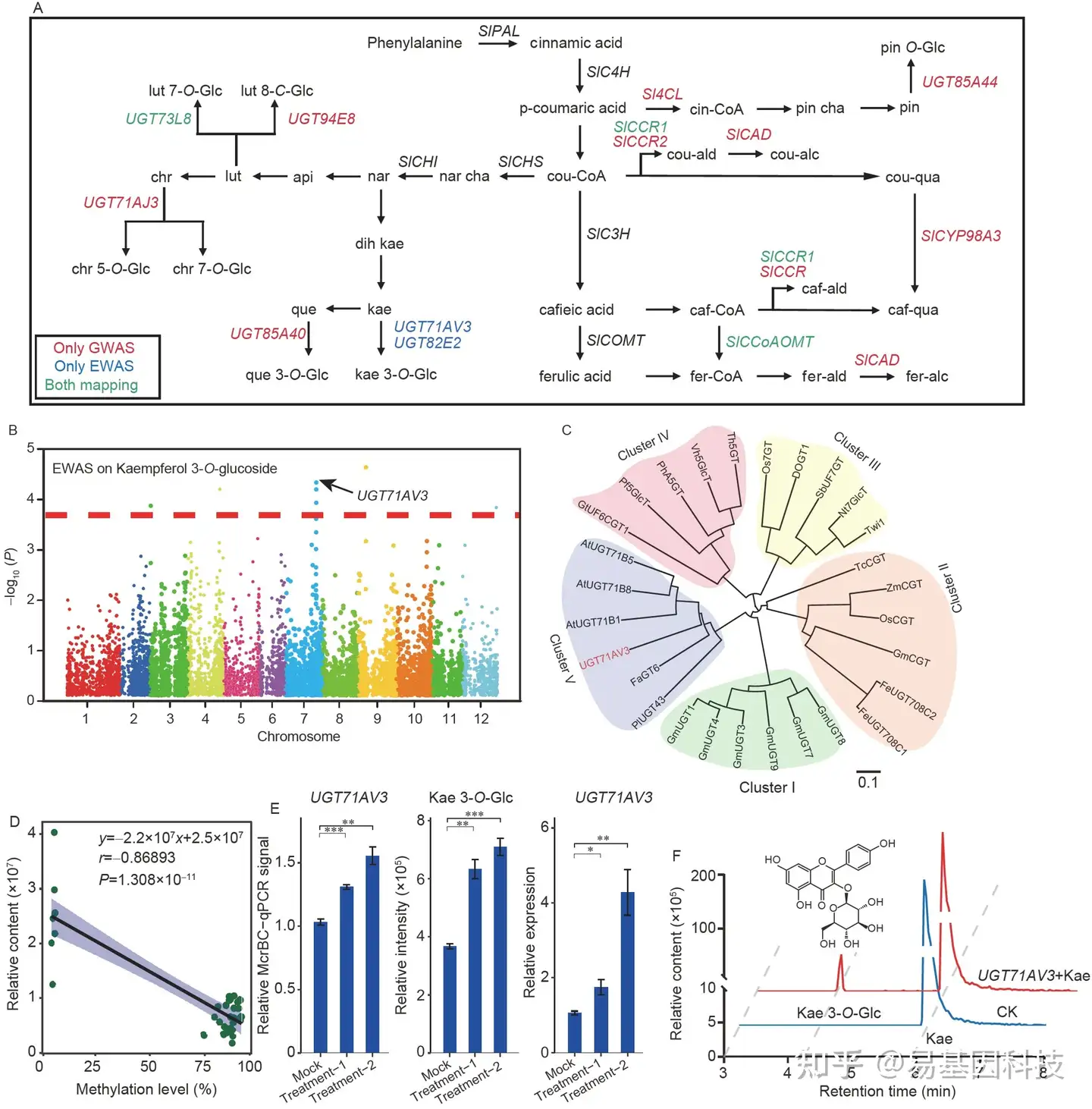
图5:基于mGWAS和mEWAS绘制的番茄多酚生物合成通路。
- 该研究中鉴定的多酚生物合成通路的已知基因和候选基因。红色、蓝色和绿色的基因分别代表仅与mGWAS相关、mEWAS相关、与两种方法均显著相关的基因。
- 山奈酚-3-O-葡萄糖甙(Kaempferol-3-O-glucoside)的EWAS曼哈顿图。虚线表示阈值。
- 类黄酮糖基转移酶基因的无根系统发育树,红色基因是候选基因(bar:每个位点0.1个氨基酸)。
- DMR(chg7g56698981)甲基化水平与山奈酚3-O-葡萄糖苷代谢物水平的关联分析。
- mock和5'-Aza处理的叶片DNA甲基化水平、UGT71AV3相对表达、山奈酚3-O-葡萄糖苷的相对含量分析(误差线表示SD。*P<0.05,**P<0.01,***t检验 P<0.001)。
- UGT71AV3和山奈酚作为底物的体外活性色谱图。
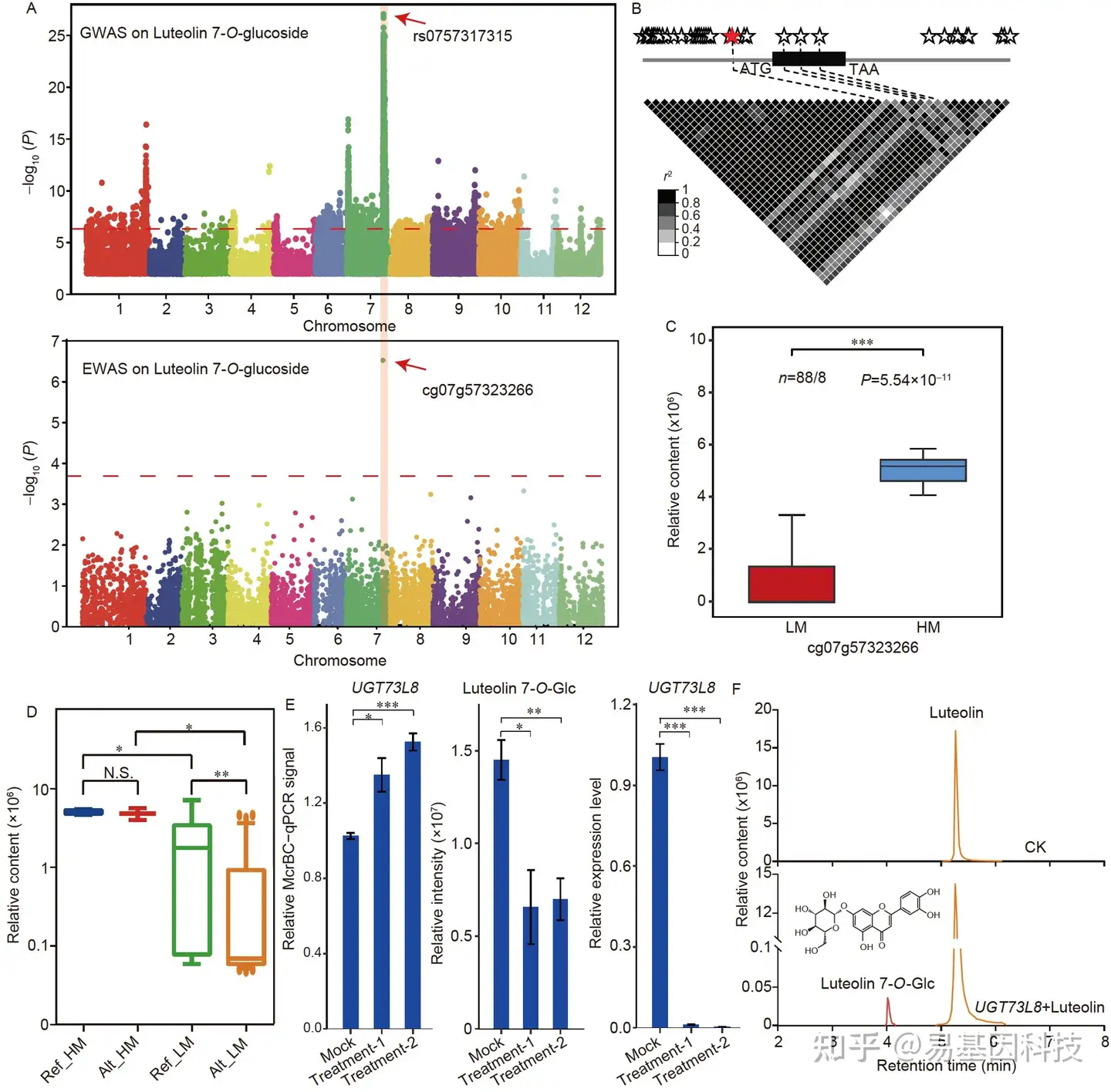
图6:遗传变异和表观遗传变异协同促进类黄酮多样性。
- 木犀草素7-O-葡萄糖苷(luteolin 7-O-glucoside)的GWAS(上图)和EWAS(下图)曼哈顿图。虚线表示GWAS和EWAS的阈值。GWAS和EWAS重叠信号由红色阴影列突出显示。强相关SNP(rs0757317315)和DMR(cg07g57323266)用红色箭头标记。
- UGT73L8中多态性位点之间成对r 2值(连锁不平衡量度)。
- 低甲基化水平(LM)和高甲基化水平(HM)中木犀草素O-glc含量箱形图(***P<0.001,Student's t-检验)。
- 具有不同单倍型的LM和HM中木犀草素7-O-葡萄糖苷含量箱形图
- 在mock和5'-Aza处理的叶片的DNA甲基化水平、UGT73L8相对表达以及木犀草素7-O-葡糖苷的相对含量分析(误差线表示SD。*P<0.05,**P<0.01,***通过t检验P<0.001)。
- UGT73L8和木犀草素作为底物的体外活性色谱图。
关于易基因全基因组重亚硫酸盐测序(WGBS)技术
全基因组重亚硫酸盐甲基化测序(WGBS)可以在全基因组范围内精确的检测所有单个胞嘧啶碱基(C碱基)的甲基化水平,是DNA甲基化研究的金标准。WGBS能为基因组DNA甲基化时空特异性修饰的研究提供重要技术支持,能广泛应用在个体发育、衰老和疾病等生命过程的机制研究中,也是各物种甲基化图谱研究的首选方法。
易基因提供的全基因组甲基化测序技术通过T4-DNA连接酶,在超声波打断基因组DNA片段的两端连接接头序列,连接产物通过重亚硫酸盐处理将未甲基化修饰的胞嘧啶C转变为尿嘧啶U,进而通过接头序列介导的 PCR 技术将尿嘧啶U转变为胸腺嘧啶T。
应用方向:
WGBS广泛用于各种物种,要求全基因组扫描(不错过关键位点)
- 全基因组甲基化图谱课题
- 标志物筛选课题
- 小规模研究课题
技术优势:
- 应用范围广:适用于所有参考基因组已知物种的甲基化研究;
- 全基因组覆盖:最大限度地获取完整的全基因组甲基化信息,精确绘制甲基化图谱;
- 单碱基分辨率:可精确分析每一个C碱基的甲基化状态。
易基因科技提供全面的DNA甲基化研究整体解决方案。
参考文献:Guo H, Cao P, Wang C, Lai J, Deng Y, Li C, Hao Y, Wu Z, Chen R, Qiang Q, Fernie AR, Yang J, Wang S. Population analysis reveals the roles of DNA methylation in tomato domestication and metabolic diversity. Sci China Life Sci. 2023 Mar 23.
相关阅读:
项目文章|WGBS+RNA-seq揭示PM2.5引起男性生殖障碍的DNA甲基化调控机制
14种全基因组DNA甲基化测序(WGBS)标准分析比对软件的比较 | 生信专区
文献速递 | 微量细胞WGBS技术揭示克隆胚胎异常DNA再甲基化新机制
标签:DNA,组学,基因组,甲基化,DMR,番茄,WGBS From: https://www.cnblogs.com/E-GENE/p/17297011.html