大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
2022年05月16日,《Cancer Res》杂志发表了题为“M6A RNA Methylation Regulates Histone Ubiquitination to Support Cancer Growth and Progression”的研究论文,该研究通过对骨肉瘤细胞系进行MeRIP-seq、RNA-seq、RIP、qRT-PCR等实验,揭示RNA去甲基化酶ALKBH5上调USP22和RNF40以抑制组蛋白H2A泛素化并诱导关键DNA复制和DNA损伤修复相关基因的表达,从而促进骨肉瘤(OS)进展。

标题:M6A RNA Methylation Regulates Histone Ubiquitination to Support Cancer Growth and Progression
时间:2022.05.16
期刊:Cancer Research
影响因子:IF 13.312
技术平台:MeRIP-seq、RNA-seq、RIP、qRT-PCR等
样本实验:
研究摘要:
骨肉瘤是最常见的骨恶性肿瘤,但受缺乏可以靶向的反复突变、缺乏有效的化疗替代方案的阻碍,骨肉瘤患者生存率在过去30年中几乎没有变化。本研究通过分析mRNA甲基化的表观遗传变化,对更好理解骨肉瘤生长机制和开发靶向治疗方法具有巨大的前景。在骨肉瘤患者中,RNA去甲基化酶ALKBH5扩增且高表达与拷贝数变化相关。ALKBH5对于促进骨肉瘤生长和转移至关重要,但它对于正常细胞生存却并非不可或缺。甲基化RNA免疫沉淀测序(MeRIP-seq)分析和功能研究表明,ALKBH5通过调控组蛋白去泛素化酶USP22和泛素连接酶RNF40的m6A水平来介导其促癌功能。骨肉瘤中ALKBH5介导的m6A缺失导致USP22和RNF40表达增加,诱导组蛋白H2A单泛素化抑制和关键促癌基因,从而驱动不受调控的的细胞周期进程、持续DNA复制和DNA损伤修复。已知泛素化H2B 的RNF40通过与DDB1-CUL4泛素E3连接酶复合体互作并影响其稳定性来抑制癌症中的H2A泛素化。总之,本研究直接将ALKBH5活性增加与癌症中的USP22/RNF40和组蛋白泛素化失调联系起来。结果表明m6A RNA甲基化与其他表观遗传机制协同作用以调控癌症生长。
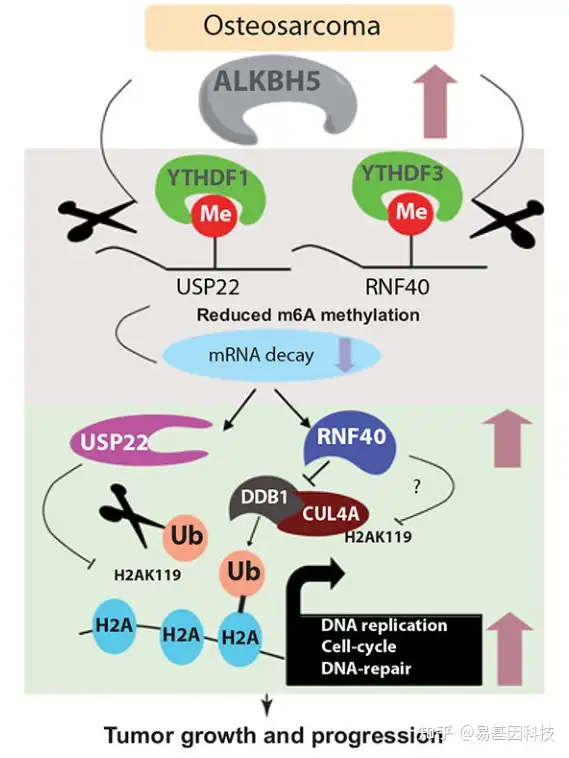 图:ALKBH5促进骨肉瘤生长和进展机制模型
图:ALKBH5促进骨肉瘤生长和进展机制模型
结果图形
(1)ALKBH5在骨肉瘤中扩增且高表达,并促进癌症生长和进展
与其他癌症类型相比,在所有m6A调节因子中,ALKBH5在骨肉瘤中最高表达。通过对97个原发性骨肉瘤样品的比较基因组杂交和转录组测序(RNA-seq)分析表明,与METTL3、METTL14或FTO相比,ALKBH5在骨肉瘤中高度扩增(>26%–40%),且其表达与拷贝数变化高度相关。研究人员分析了骨肉瘤中m6A水平与ALKBH5表达之间的关系。
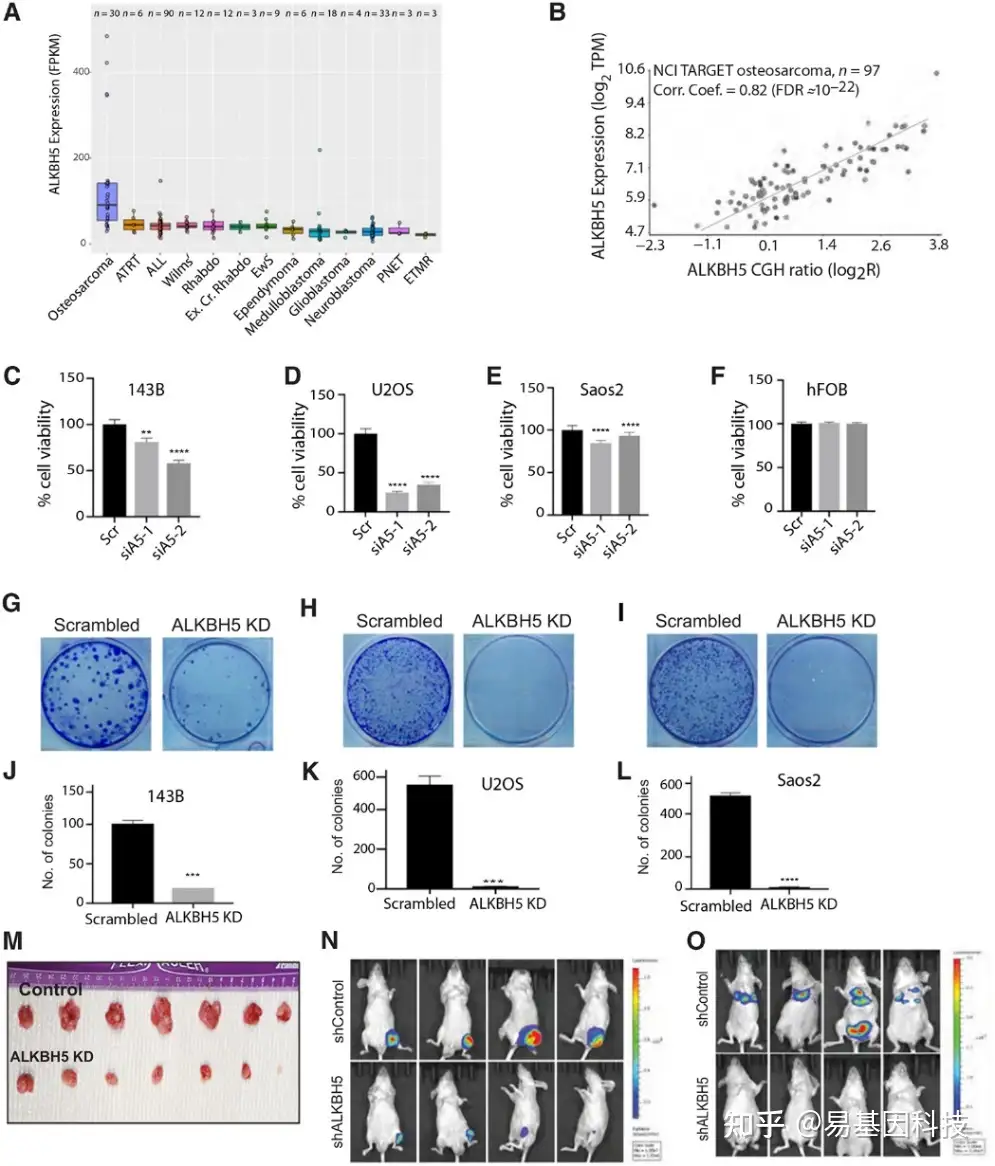
图1:RNA去甲基化酶ALKBH5在骨肉瘤中扩增并促进骨肉瘤生长和进展。
- 儿科临床前试验联盟(PPTC)数据集的泛癌分析显示ALKBH5基因在患者衍生异种移植物中表达,数据使用PCAT生成。
- TARGET数据库的比较基因组杂交和RNA-seq数据显示ALKBH5基因扩增以及骨肉瘤样品中ALKBH5表达和拷贝数变化之间的强相关性。
C-F. 通过加扰siRNA(Scr)、ALKBH5 siRNA-1(siA5-1)或ALKBH5 siRNA-2(siA5-2)转染的143B(C)、U2OS(D)、Saos2(E)和hFOB细胞(F)的细胞活力百分比(通过CellTiter-Glo发光或Alamar Blue细胞活力试验检测)。
G-I. 利用加扰siRNA或ALKBH5 siRNA(ALKBH5 KD)转染的143B(G),U2OS(H)和Saos2(I)细胞的克隆形成实验。
J-L. 图1G-I中计数的结晶紫染色克隆的平均数条形图。
M. 注射了稳定表达shControl(n=7)或shALKBH5(n=7)的1×106 143B细胞的裸鼠体内代表性肿瘤生长。
N. 在小鼠胫骨内植入用对照(shControl;N=5)或ALKBH5 shRNA(shALKBH5;N=5)稳定转染的143B细胞后的代表性肿瘤生长。
O. 静脉注射用对照(shControl;n=5)或ALKBH5 shRNA(shALKBH5;n=5)稳定转染的143B细胞的小鼠代表性转移病灶。C–F和J–L中显示的数据是平均值±SEM(n≥3个独立实验)。C-F中的P值使用单因素方差分析和Dunnett多重比较检验计算,**,P<0.01;***,P<0.001;****,P<0.0001。
(2)ALKBH5调控DNA复制、细胞周期和DNA损伤修复基因的表达
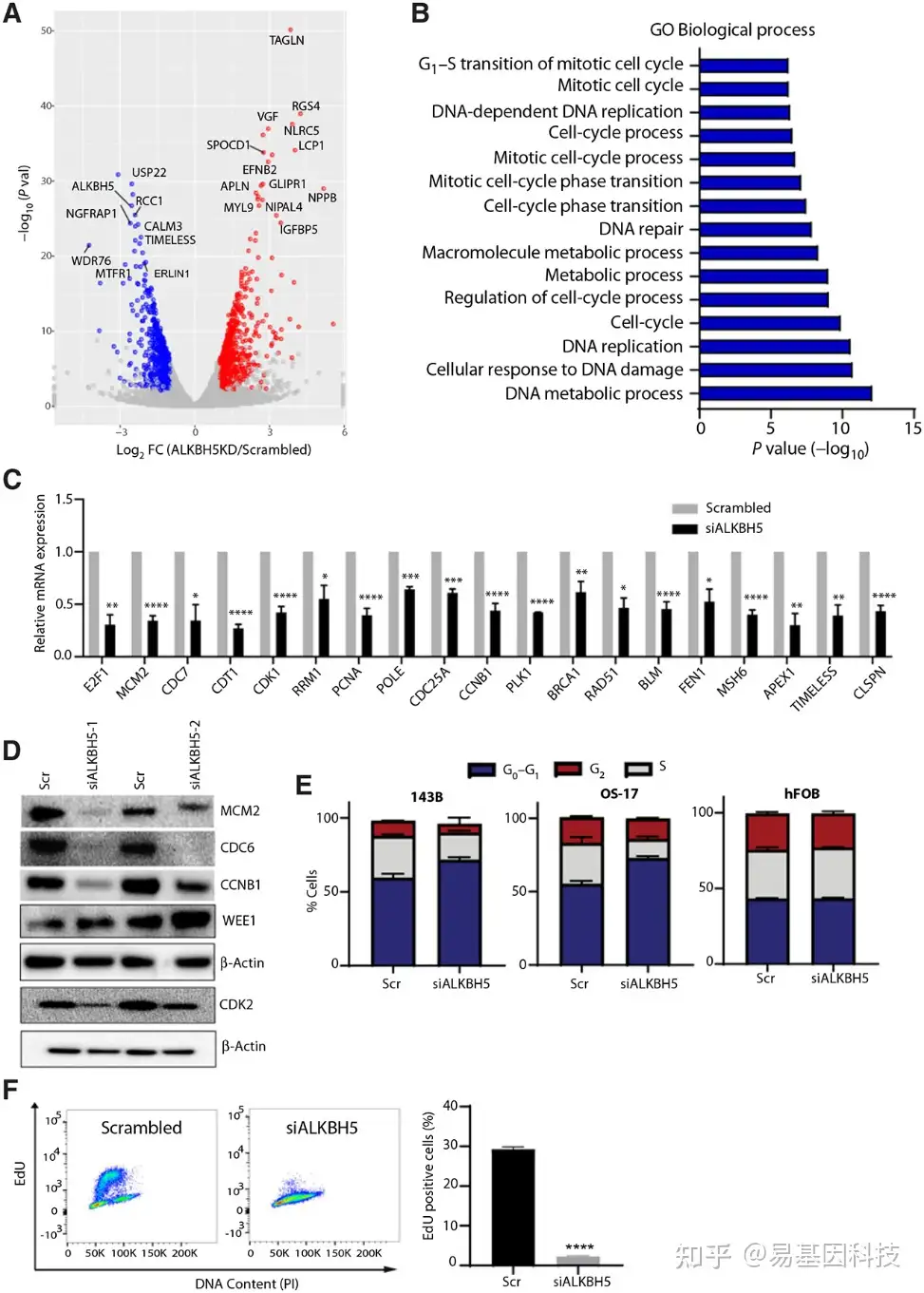
图2:ALKBH5通过调控细胞周期和DNA复制相关基因表达来促进细胞周期进程。
- 用加扰siRNA或siALKBH5(KD)转染48小时的143B细胞中的基因表达变化火山图。差异表达基因的截止标准包括绝对log2倍数变化>1和P<0.05(红色,差异增加;蓝色,减少基因)。
- GO分析结果显示ALKBH5 KD 143B细胞(减少基因)中高度富集的生物过程。
- 加扰siRNA或ALKBH5 siRNA-1(siALKBH5)转染的143B细胞中靶基因表达的qRT-PCR分析结果。
- 使用所示蛋白抗体在加扰siRNA和两种ALKBH5 siRNA(siALKBH5-1和siALKBH5-2)转染的143B细胞中靶基因的Western blot分析。用β-肌动蛋白作为负载对照。凝胶照片代表至少三次独立实验。显示MCM2、CDC6、CCNB1、WEE1和β-肌动蛋白的Western blot实验在一个凝胶上运行(剥离后),而CDK2和β-肌动蛋白在单独的凝胶上运行。
- 加扰siRNA或ALKBH5 siRNA转染的143B、OS-17和hFOB细胞的细胞周期分布。碘化丙啶染色DNA含量,并用FlowJo软件进行流式细胞术分析以鉴定细胞周期不同阶段的细胞群。
- EdU免疫荧光与用EdU脉冲标记的U2OS细胞的DNA含量的双变量细胞周期分析。条形图显示加扰siRNA和ALKBH5 siRNA转染的U2OS细胞中的EdU细胞定量。使用标准Student t检验计算P值。*,P<0.05;**,P<0.01;***,P<0.001;****,P<0.0001。条形图表示至少三次独立实验的平均值±SEM+
(3)ALKBH5缺失诱导复制应激并损害DNA损伤修复
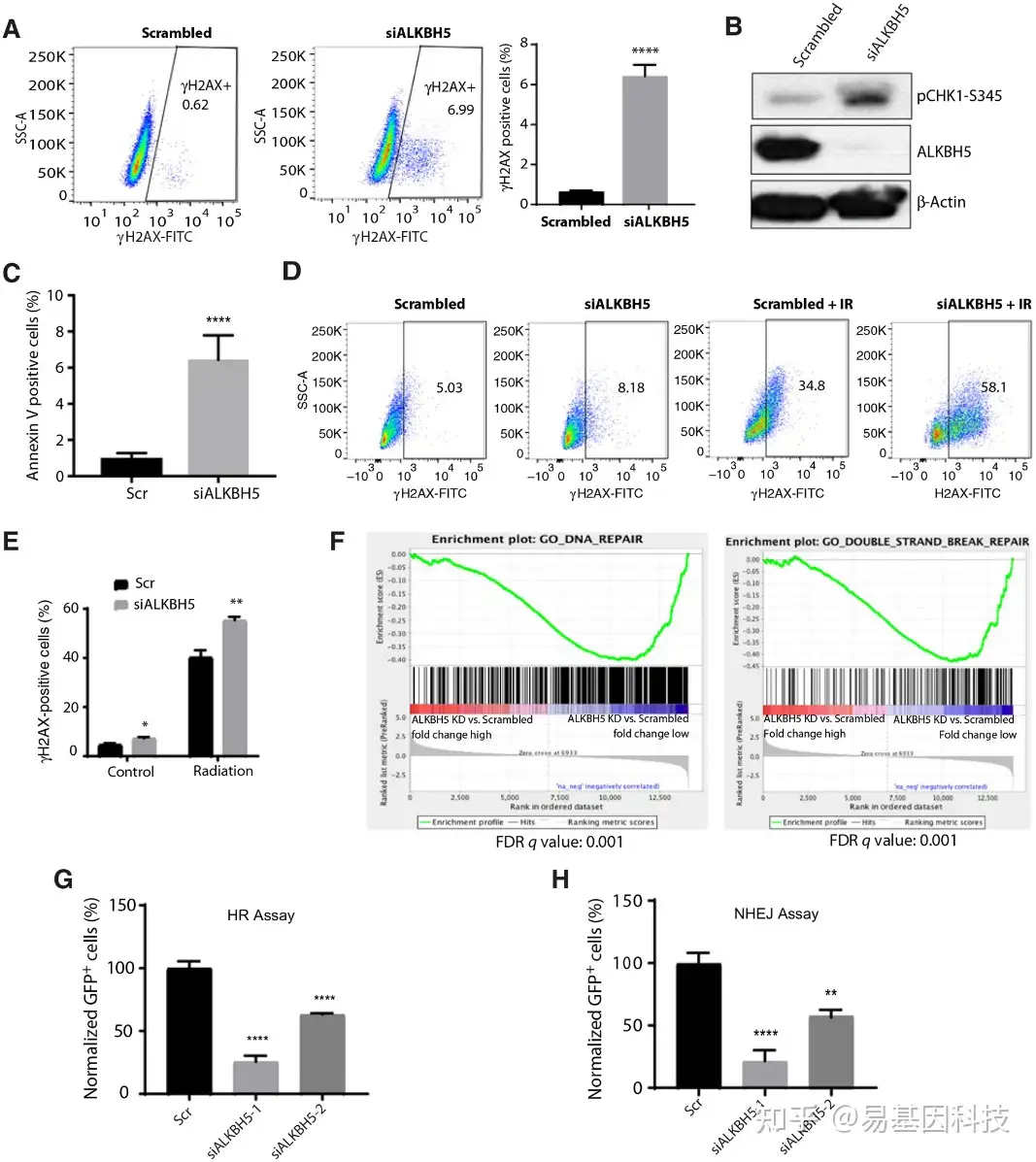
图3:ALKBH5调节癌细胞中的DNA损伤修复事件。
- 加扰siRNA或ALKBH5 siRNA转染的U2OS细胞中的γH2AX阳性细胞代表性点图。在流式细胞术分析之前,用加扰siRNA或ALKBH5siRNA转染U2OS细胞72小时。条形图显示平均值±SEM(n=3)。
- 使用所示蛋白抗体对加扰siRNA和ALKBH5 siRNA转染的U2OS细胞进行Western Blot分析。β-肌动蛋白作为负载对照。
- 用加扰siRNA或ALKBH5 siRNA转染的膜联蛋白(Annexin)V-FITC阳性U2OS细胞的百分比。
- 流式细胞术分析结果显示,γH2AX阳性细胞存在于加扰siRNA和ALKBH5 siRNA转染的143B骨肉瘤细胞中。无电离辐射(IR)或10Gy IR处理细胞.24小时后,对细胞进行γH2AX染色以估计未修复的双链断裂。
- D中显示的γH2AX阳性细胞的定量条形图。
- 与加扰siRNA转染的143B细胞相比,GSEA显示ALKBH5 KD细胞中DNA损伤修复通路富集。
- DR-GFP报告基因实验结果显示DSB诱导的HR修复。用加扰siRNA或ALKBH5 siRNA转染U2OS-DR-GFP细胞,然后用表达I-SceI核酸内切酶的pCAGGS载体或作为对照的空载体转染。如果通过HR修复,I-SceI核酸内切酶表达诱导DSB通过流式细胞术导致GFP细胞。
- EJ5-GFP报告实验显示总NHEJ。用加扰siRNA或ALKBH5 siRNA转染EJ5-GFP U2OS细胞,然后用表达I-SceI核酸内切酶的pCAGGS载体或作为对照的空载体转染。如果通过NHEJ修复,I-SceI核酸内切酶诱导的DSB通过流式细胞术产生GFP细胞。
(4)转录组范围的MeRIP-seq鉴定组蛋白去泛素化蛋白USP22为ALKBH5靶点
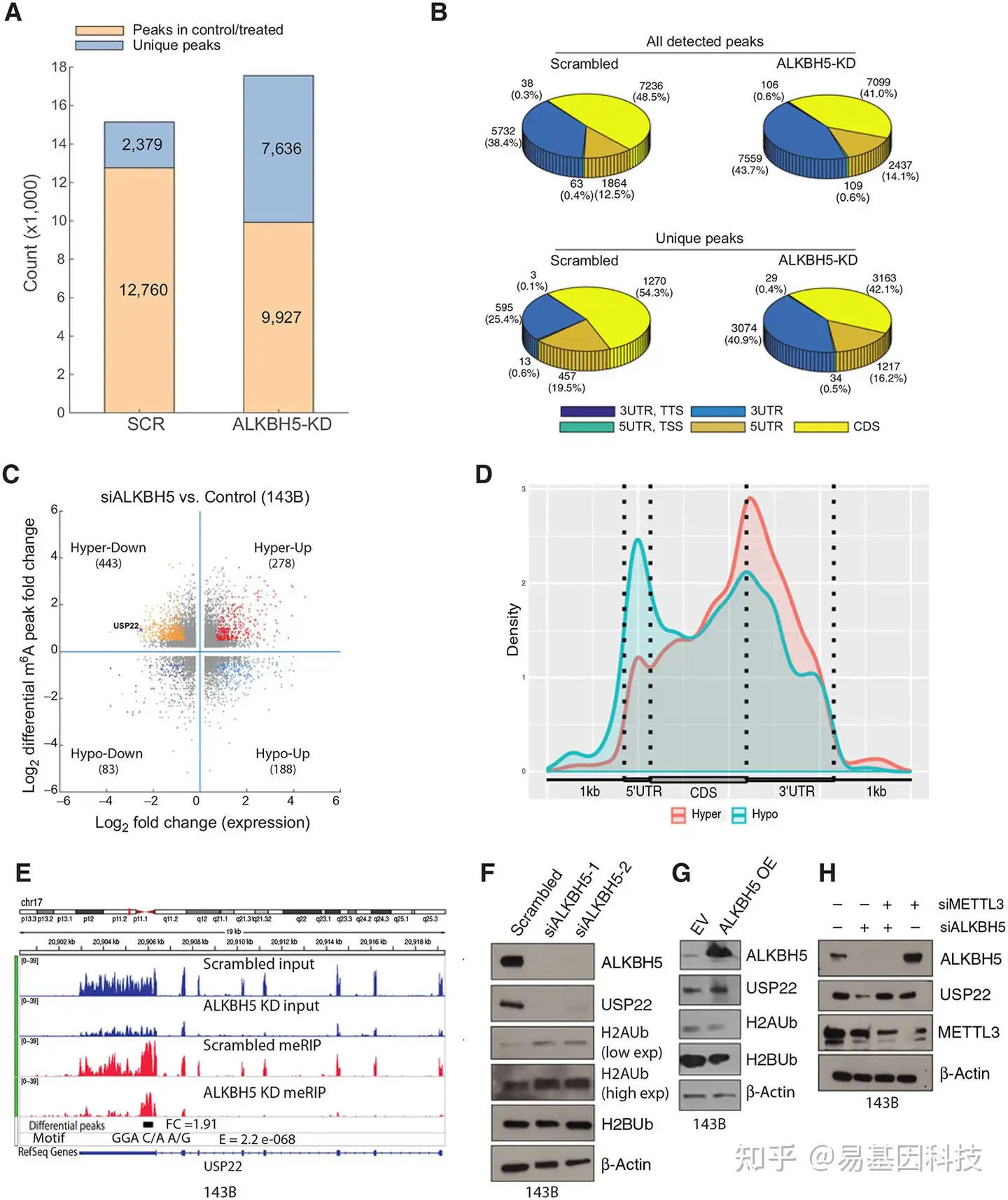
图4:对照组和ALKBH5敲除骨肉瘤(OS)细胞的m6A甲基化分析。
- 使用MeRIP-seq在加扰siRNA(SCR)和ALKBH5 siRNA(ALKBH5 KD)转染的143B细胞中鉴定的peaks数。常见m6A基因共享至少一个共同peak,而特异性m6A基因在加扰siRNA和ALKBH5 KD转染的OS细胞之间没有共同peaks。
- 加扰siRNA和ALKBH5 siRNA(ALKBH5 KD)转染的143B细胞中不同基因区域中整体(上)和特异性(下)m6A peaks比例。
- 与加扰siRNA转染的细胞相比,ALKBH5敲低143B细胞中m6A甲基化水平和表达水平显著变化的基因分布分析。
- 高和低m6A peaks在mRNA不同区域的分布。
- USP22在ALKBH5 siRNA或加扰siRNA转染的143B细胞中显示出显著富集的m6A peaks(adj P<0.05)。顶部两个轨迹(蓝色)表示input,底部两个轨迹分别表示加扰siRNA和ALKBH5 siRNA转染的143B细胞的MeRIP结果。通过使用MeTDiff程序将整体m6A值除以特定基因的靶位点表达水平来计算检测到的差异m6A peaks位点及其倍数变化值(差异峰轨迹)。将MeRIP-seq数据进行MEME-ChIP(E值=2.2E-068)来确定m6A motif(显示为motif GGA C/A A/G)分布。
- 使用针对所示的蛋白抗体,在加扰siRNA和ALKBH5 siRNA转染的143B OS细胞中靶基因的Western Blot分析。
- 空载体或ALKBH5表达质粒转染的143B细胞中靶基因的Western Blot分析
- 加扰siRNA、ALKBH5 siRNA,METTL3 siRNA或ALKBH5+METTL3 siRNA转染的143B细胞中靶基因的Western Blot分析。凝胶照片表示至少三个独立实验。
(5)ALKBH5通过USP22和RNF40调控H2A K119单泛素化
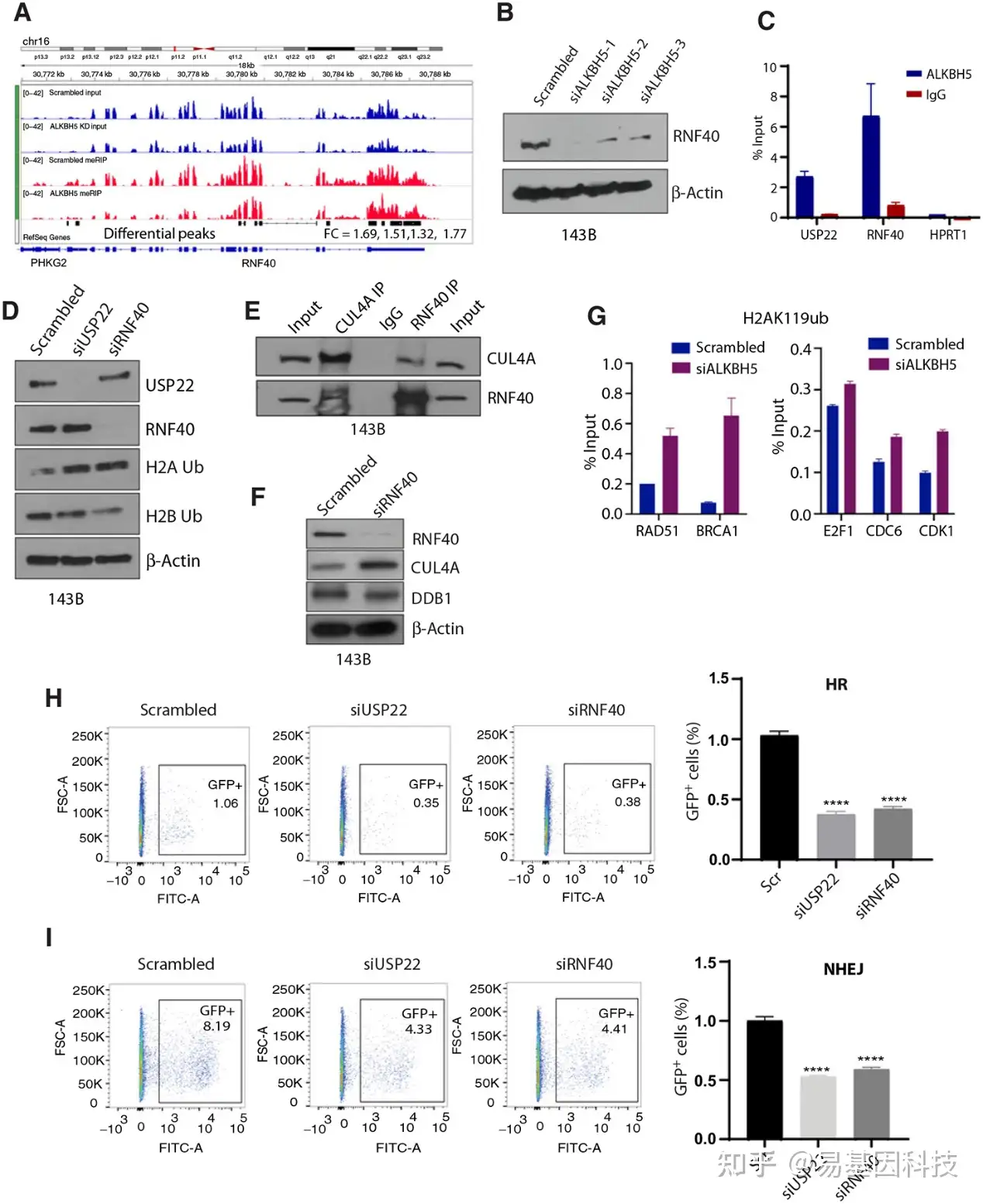
图5:ALKBH5-USP22/RNF40轴调控组蛋白H2AK119单泛素化和DNA损伤修复。
- 与加扰siRNA转染的细胞相比,RNF40在ALKBH5 siRNA转染的143B细胞中显著富集了m6A peaks(adj P<0.05)。顶部两个轨道(蓝色)分别代表input,底部两个轨迹显示加扰siRNA和ALKBH5 siRNA转染的143B细胞的MeRIP结果。
- 使用RNF40抗体对加扰siRNA和ALKBH5 siRNA转染的143B细胞进行Western Blot分析。
- qRT-PCR结果显示ALKBH5或IgG的抗体进行RNA免疫沉淀后,143B细胞中ALKBH5靶基因USP22和RNF40富集。HPRT1为阴性对照。
- Western Blot分析显示所示蛋白抗体在加扰USP22 siRNA或RNF40 siRNA转染的143B细胞中RNF40和USP22以及单泛素化的H2A(H2A-ub)和H2B(H2B-ub)水平。
- 使用143B裂解液用RNF40、CUL4A或IgG抗体进行免疫沉淀分析。显示了RNF40和CUL4A的免疫印迹。
- Western Blot分析显示加扰或RNF40 siRNA转染的143B细胞中RNF40、CUL4A和DDB1水平。
- 加扰siRNA或ALKBH5 siRNA转染的143B细胞使用H2Aub K119抗体进行ChIP qRT-PCR。条形图显示图中所示基因的启动子区域附近的组蛋白标记富集。
- DR-GFP实验的GFP阳性细胞反映DSB诱导的HR修复的FACS分析。用加扰siRNA、USP22 siRNA(siUSP22)或RNF40 siRNA(siRNF40)转染U2OS-DR-GFP细胞,然后用表达I-SceI核酸内切酶的pCAGGS载体或空载体转染作为对照。
- EJ5-GFP报告基因分析的GFP阳性细胞反映了总NHEJ的 FACS。用加扰siRNA、USP22 siRNA(siUSP22)或RNF40 siRNA(siRNF40)转染EJ5-GFP U2OS细胞,然后用表达I-SceI核酸内切酶的pCAGGS载体或空载体转染作为对照。
(6)AUSP22和RNF40介导ALKBH5的促癌功能
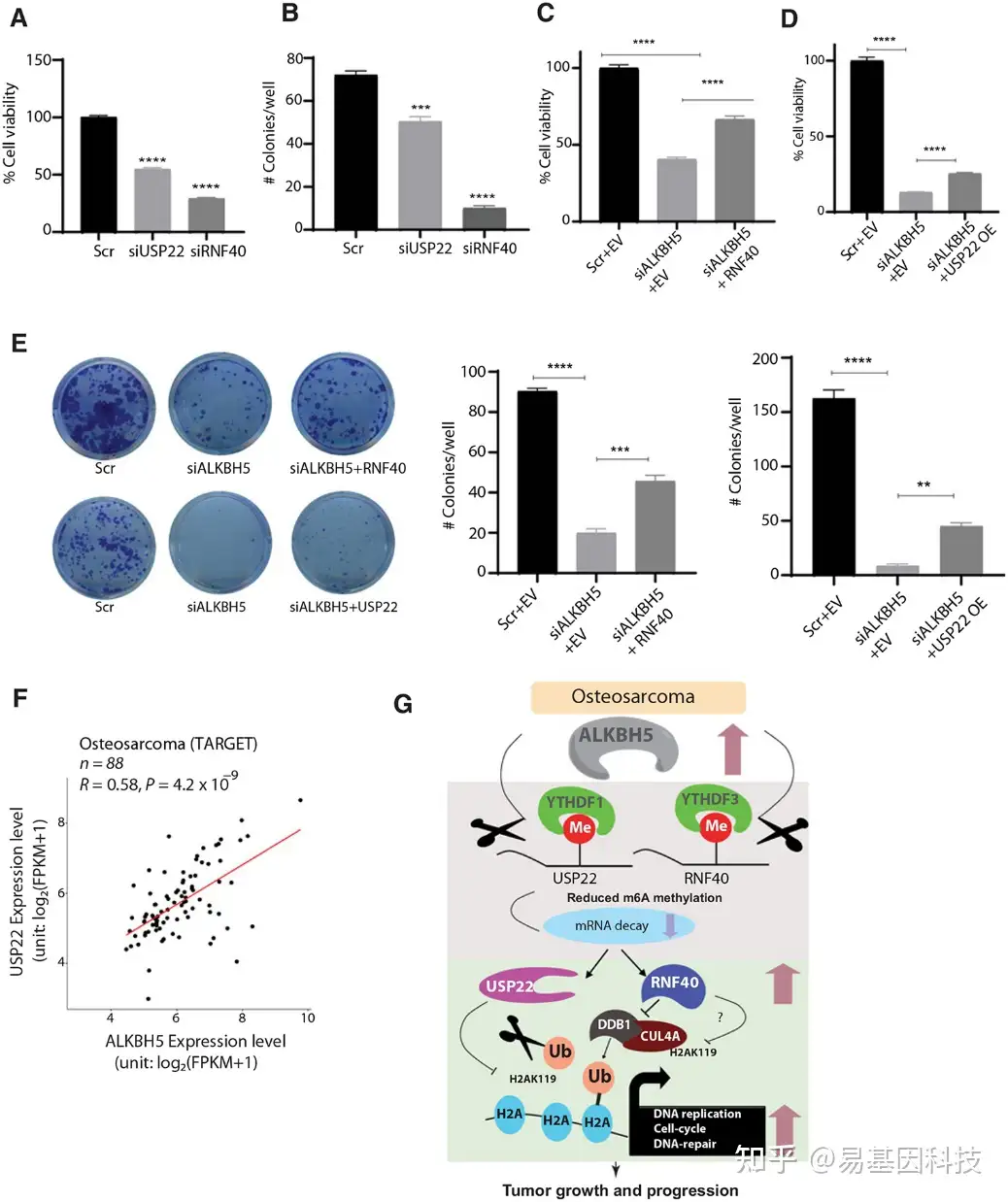
图6:USP22和RNF40介导ALKBH5 ALKBH5-USP22/RNF40轴的促癌效应,以调控组蛋白H2AK119单泛素化和DNA损伤修复。
- 经加扰siRNA(Scr)、USP22 siRNA(siUSP22)或RNF40 siRNA(siRNF40)转染的143B细胞中细胞活力百分比条形图。通过CellTiter-Glo发光细胞活力实验检测细胞活力。
- 加扰siRNA、USP22 siRNA或RNF40 siRNA转染的143B细胞克隆形成实验。条形图显示每孔计数的结晶紫染色克隆数。
- 加扰siRNA(Scr)+空载体(EV)或ALKBH5 siRNA+EV或ALKBH5 siRNA+RNF40过表达质粒共转染的143B细胞的细胞活力百分比。
- Scr+空载体(EV)或ALKBH5 siRNA+EV或ALKBH5 siRNA+USP22过表达质粒共转染的143B细胞的细胞活力百分比。
- 加扰siRNA+EV或ALKBH5 siRNA+EV或ALKBH5 siRNA+USP22或RNF40过表达质粒共转染的143B细胞的克隆形成实验结果。
- TARGET数据库中骨肉瘤肿瘤样品显示ALKBH5和USP22表达之间的显著相关性。
- ALKBH5促进骨肉瘤生长和进展机制模型。研究结果表明,ALKBH5扩增介导其过表达导致m6A甲基化水平降低,从而增加组蛋白去泛素化酶USP22和泛素连接酶RNF40在骨肉瘤中的稳定性。USP22稳定性增加反过来导致去泛素化增加和单泛素化组蛋白H2A水平降低。另一方面,ALKBH5过表达骨肉瘤中的RNF40稳定性导致DDB1-CUL4A连接酶复合体的互作和抑制增加,进而介导单泛素化组蛋白H2A水平降低。单泛素化组蛋白H2A(其压缩染色质并充当转录抑制标记)降低,反式激活关键的生长/促瘤基因,导致不受调控的细胞周期进展、连续的DNA复制和DNA损伤修复以支持骨肉瘤生长。
总结:
本研究有几个关键结果。
- 本研究表明m6A RNA甲基化对于维持组蛋白去泛素化酶和泛素连接酶的最佳比例以调控H2A泛素化至关重要。
- 组蛋白去泛素化酶USP22和泛素连接酶RNF40在癌症中的表达受到调控。这一发现意义重大,因为人们对癌症中组蛋白泛素化的调控知之甚少。此外,研究揭示了导致USP22在癌症中过表达的机制将阐明其功能,并可能为靶向其促癌作用的方法提供信息。
- 研究首次表明了RNF40-DDB1-CUL4A在调控H2AK119泛素化中的作用。
- 最后,研究结果支持ALKBH5激活构建了一个异常的表观遗传程序来促进癌症的观点。因此,依赖于ALKBH5致癌信号,通过靶向ALKBH5的方法来治疗癌症将具有巨大的临床转化潜力。
关于易基因RNA m6A甲基化测序(MeRIP-seq)技术
易基因MeRIP-seq技术利用m6A特异性抗体富集发生m6A修饰的RNA片段(包括mRNA、lncRNA等rRNA去除所有RNA),结合高通量测序,可以对RNA上的m6A修饰进行定位与定量,总RNA起始量可降低至10μg,最低仅需1μg总RNA。广泛应用于组织发育、干细胞自我更新和分化、热休克或DNA损伤应答、癌症发生与发展、药物应答等研究领域;可应用于动物、植物、细胞及组织的m6A检测。
大样本量m6A-QTL性状关联分析,传统MeRIP单个样品价格高,通常难以承担。易基因开发建立MeRIP-seq2技术,显著提成IP平行性,实现不同样本间相对定量,降低检测成本。
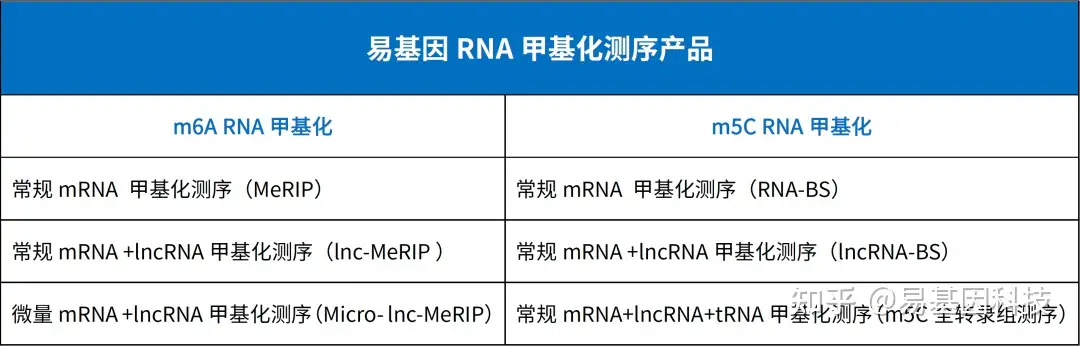
易基因提供适用于不同科研需求的MeRIP技术:
- m6A甲基化-常量mRNA 甲基化测序(MeRIP-seq)
- m6A甲基化-常量mRNA +lncRNA甲基化测序(lnc-MeRIP-seq)
- m6A甲基化-微量mRNA +lncRNA甲基化测序(Micro-lnc-MeRIP-seq)
- 高通量m6A甲基化-常量mRNA甲基化测序(MeRIP-seq2)
技术优势:
- 起始量低:样本起始量可降低至10-20μg,最低仅需1μg总RNA;
- 转录组范围内:可以同时检测mRNA和lncRNA;
- 样本要求:可用于动物、植物、细胞及组织的m6A检测;
- 重复性高:IP富集重复性高,最大化降低抗体富集偏差;
- 应用范围广:广泛应用于组织发育、干细胞自我更新和分化、热休克或DNA损伤应答、癌症的发生与发展、药物应答等研究领域。
研究方向:
m6A甲基化目前主要运用在分子机制的理论性研究
- 疾病发生发展:肿瘤、代谢疾病(如肥胖/糖尿病)、神经和精神疾病(如阿尔兹海默症/抑郁症)、炎症…
- 发育和分化:早期胚胎发育、个体/组织/器官生长发育、干细胞分化与命运决定、衰老
- 环境暴露与响应:污染、抗逆、生活方式
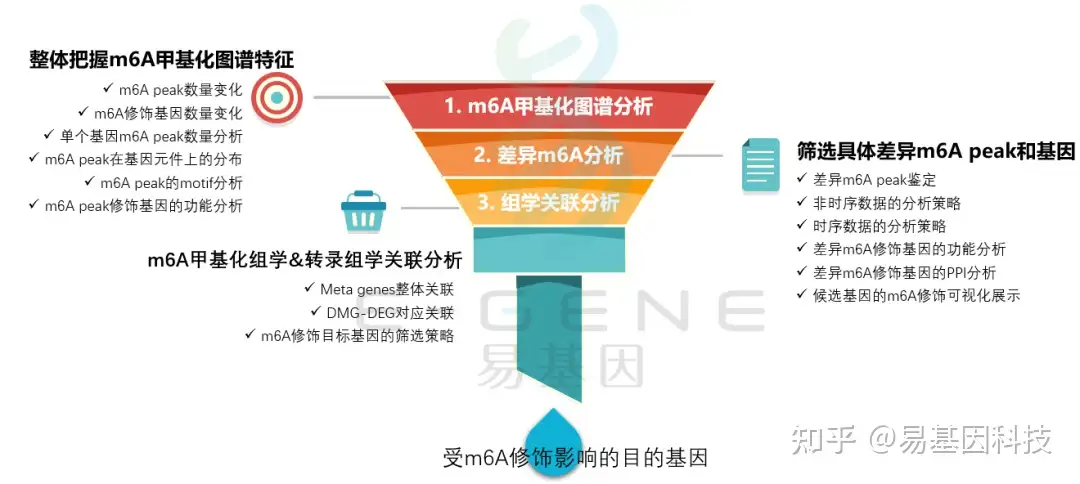
关于m6A甲基化研究思路
(1)整体把握m6A甲基化图谱特征:m6A peak数量变化、m6A修饰基因数量变化、单个基因m6A peak数量分析、m6A peak在基因元件上的分布、m6A peak的motif分析、m6A peak修饰基因的功能分析
(2)筛选具体差异m6A peak和基因:差异m6A peak鉴定、非时序数据的分析策略、时序数据的分析策略、差异m6A修饰基因的功能分析、差异m6A修饰基因的PPI分析、候选基因的m6A修饰可视化展示
(3)m6A甲基化组学&转录组学关联分析:Meta genes整体关联、DMG-DEG对应关联、m6A修饰目标基因的筛选策略
(4)进一步验证或后期试验
易基因科技提供全面的RNA甲基化研究整体解决方案,技术详情了解请致电易基因。
参考文献:
Yadav P, Subbarayalu P, Medina D, Nirzhor S, Timilsina S, Rajamanickam S, Eedunuri VK, Gupta Y, Zheng S, Abdelfattah N, Huang Y, Vadlamudi R, Hromas R, Meltzer P, Houghton P, Chen Y, Rao MK. M6A RNA Methylation Regulates Histone Ubiquitination to Support Cancer Growth and Progression. Cancer Res. 2022 May 16;82(10):1872-1889.
相关回顾:
项目集锦 | 易基因近期m6A甲基化(MeRIP-seq)研究成果
干货:m6A RNA甲基化MeRIP-seq测序分析实验全流程解析
深度综述:癌症中RNA修饰机制的遗传和表观遗传失调(m6A+m1A+m5C+ψ)
 标签:化来,seq,m6A,细胞,甲基化,143B,转染,ALKBH5,siRNA
From: https://www.cnblogs.com/E-GENE/p/17136725.html
标签:化来,seq,m6A,细胞,甲基化,143B,转染,ALKBH5,siRNA
From: https://www.cnblogs.com/E-GENE/p/17136725.html