PCA降维
PCA是一种无监督降维算法,它是最常用的降维算法之一,可以很好的解决因变量太多而复杂性,计算量增大的弊端。
本质上讲,PCA就是将高维的数据通过线性变换投影的方式映射到低维空间上去,并且保证在投影的维度上,原数据的信息量最大(损失最小)。
PCA的具体步骤
- 1.特征中心化。每一维的数据都减去该维的均值。这里的“维”指的就是一个特征(或属性),变换之后每一维的均值都变成了0。每一列减去该列均值后,得到矩阵B
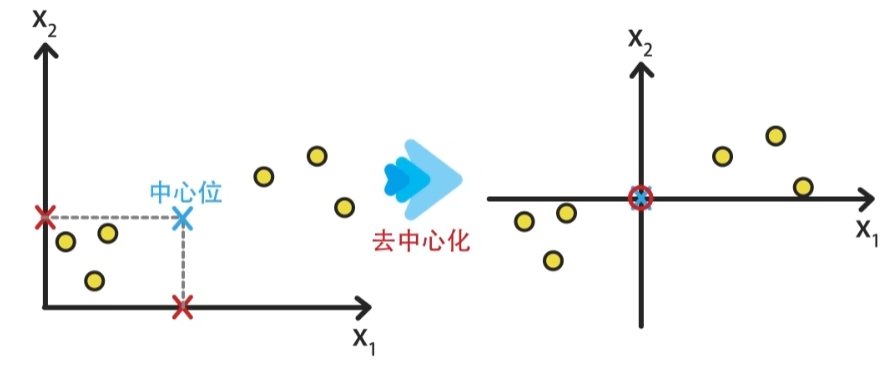
去中心化不会影响样本的分布性质,可以简化PCA算法的推倒过程
- 计算B的协方差矩阵C
- 求出协方差矩阵C的特征值和对应的特征向量
- 从大到小排序特征值,取得最前的k个特征向量P
- 将数据转换到k个特征向量构建的新空间中
import matplotlib
import numpy as np
from sklearn.decomposition import PCA
from sklearn.datasets import fetch_olivetti_faces
import matplotlib.pyplot as plt
import cv2
matplotlib.use('TkAgg') # 指定交互式框架为TkAgg
# 加载人脸数据集
faces = fetch_olivetti_faces()
pic_seed = 10
X = faces.data
# 随机选择一张人脸图片
face = X[pic_seed].reshape(64, 64)
# 将人脸数据进行PCA降维
# 将数据降维到50个主成分
# n_components = [50]
n_components = [50,40,30,20,10]
# 显示结果
fig, axs = plt.subplots(1, len(n_components)+1, figsize=(10, 10))
axs[0].imshow(face, cmap='gray')
axs[0].set_title('Original Face')
index = 1
for items in n_components:
pca = PCA(n_components=items)
X_pca = pca.fit_transform(X)
# 将降维后的数据进行逆转换
X_restored = pca.inverse_transform(X_pca)
face_restored = X_restored[pic_seed].reshape(64, 64)
axs[index].imshow(face_restored, cmap='gray')
axs[index].set_title(str(items))
index += 1
plt.show()

左一为原始的64*64图像,通过pca降维成50、40、30、20、10个维度之后
通过逆转换还原图像得到结果
还有一个是葡萄酒数据集
# 读取数据()
df_wine = pd.read_csv('wine.data',header=None)
# 设置列索引
df_wine.columns = ['Class label', 'Alcohol', 'Malic acid', 'Ash',
'Alcalinity of ash', 'Magnesium', 'Total phenols',
'Flavanoids', 'Nonflavanoid phenols', 'Proanthocyanins',
'Color intensity', 'Hue',
'OD280/OD315 of diluted wines', 'Proline']
数据维度为178*14
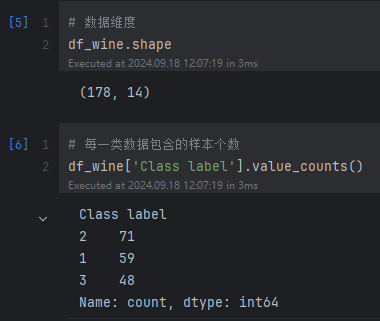
该数据集是UCI的公开数据集,是对意大利同一地区种植的葡萄酒进行分析的结果,数据集共14列数据,第一个属性是类标识符,分别是1/2/3来表示,代表葡萄酒的三个分类。剩余的13个属性是,酒精、苹果酸、灰、灰分的碱度、镁、总酚、黄酮类化合物、非黄烷类酚类、原花色素、颜色强度、色调等。
数据集划分
# 数据集设置:X为样本特征数据,y为目标数据,即标注结果
X, y = df_wine.iloc[:, 1:].values, df_wine.iloc[:, 0].values
# 数据集划分: 将数据集划分为训练集和测试集数据(测试集数据为30%,训练集为70%)
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3,
stratify=y,
random_state=0)
数据标准化
# 实例化
sc = StandardScaler()
# 对数据集进行标准化(一般情况下我们在训练集中进行均值和方差的计算,直接在测试集中使用)
X_train_std = sc.fit_transform(X_train)
X_test_std = sc.transform(X_test)
PCA实现
# 计算协方差矩阵
cov_mat = np.cov(X_train_std.T)
# 对协方差矩阵进行特征值分解
eigen_vals, eigen_vecs = np.linalg.eig(cov_mat)
得到特征值
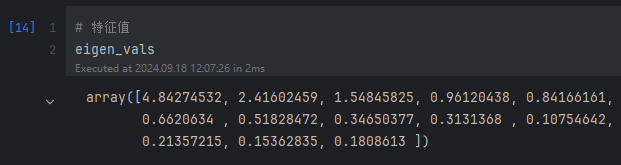
特征值分布
# 特征值之和
tot = sum(eigen_vals)
# 对特征进行排序,并计算所占的比例
var_exp = [(i / tot) for i in sorted(eigen_vals, reverse=True)]
# 累计求和
cum_var_exp = np.cumsum(var_exp)
# 绘制图像
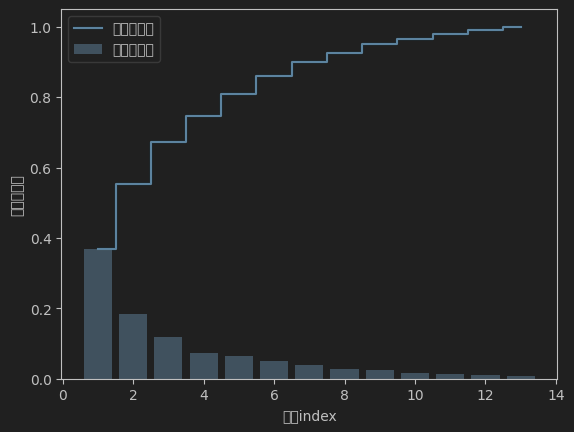
plt.figure()
plt.bar(range(1, 14), var_exp, alpha=0.5, align='center',
label='特征值分布')
plt.step(range(1, 14), cum_var_exp, where='mid',
label='累计特征值')
plt.ylabel('特征值比例')
plt.xlabel('特征index')
plt.legend(loc='best')
特征降维
# 创建列表,由(eigenvalue, eigenvector)元组构成
eigen_pairs = [(np.abs(eigen_vals[i]), eigen_vecs[:, i])
for i in range(len(eigen_vals))]
# 按特征值从大到小对列表(eigenvalue, eigenvector)排序
eigen_pairs.sort(key=lambda k: k[0], reverse=True)

# 取前两个特征值对应的特征向量作为主要成分
w = np.hstack((eigen_pairs[0][1][:, np.newaxis],
eigen_pairs[1][1][:, np.newaxis]))
进行特征压缩
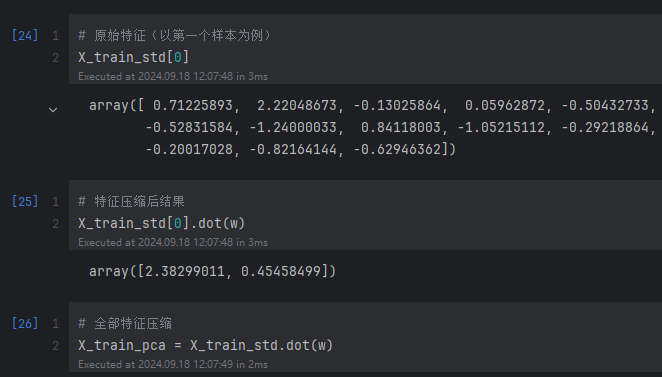
结果展示
# 特征压缩后结果展示
colors = ['r', 'b', 'g']
markers = ['s', 'x', 'o']
for l, c, m in zip(np.unique(y_train), colors, markers):
# 按照样本的真实值进行展示
plt.scatter(X_train_pca[y_train == l, 0],
X_train_pca[y_train == l, 1],
c=c, label=l, marker=m)
plt.xlabel('PC 1')
plt.ylabel('PC 2')
plt.legend(loc='lower left')
plt.tight_layout()
plt.show()
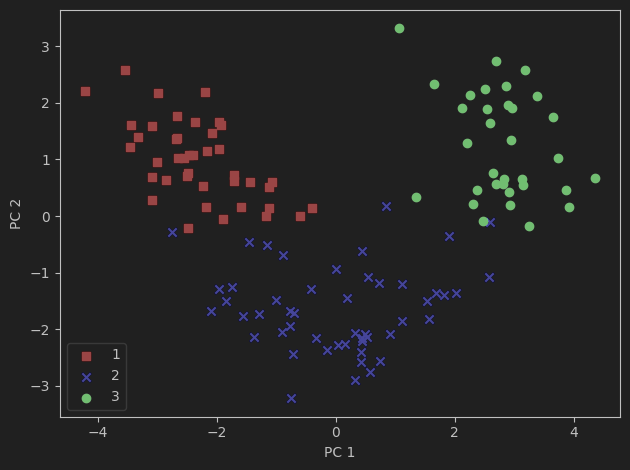
这个结果有助于之后进行分类
标签:特征值,plt,eigen,降维,神经网络,train,PCA,数据 From: https://www.cnblogs.com/jinwan/p/18424009