文章题目
False signals induced by single-cell imputation
中文名:
单细胞插补引起的假信号
文章地址:
https://f1000research.com/articles/7-1740/v2
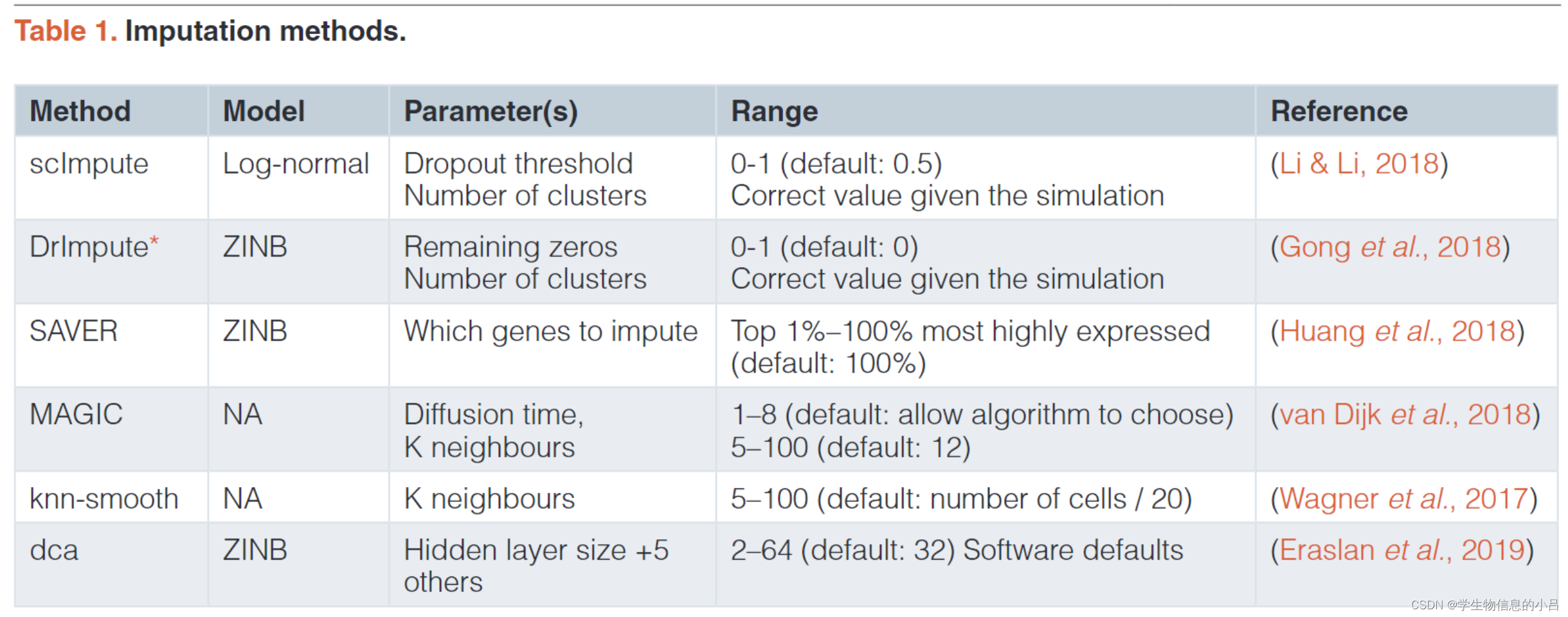
评价插补方法:
SAVER,DrImpute,scImpute,DCA,MAGIC,knn-smooth
上述方法基于原理不同
SAVER,scImpute,drImpute基于模型,knn-smooth和MAGIC基于高斯平滑的思路,DCA是一种使用自编码器的基于深度学习的方法
评价指标构造方法:
1.构造简单的负二项数据集
1000个细胞 500个基因(平均表达确定在一个区间水平内) 细胞类型2类
数据集中不存在dropout现象(没有0值)
数据集中基因 一半处于差异表达状态 另外一半独立绘制 不存在差异表达
鉴定方法:通过SPearman相关性鉴定细胞间相关性,相关性确定后,用Bonferroni矫正相关性
假阳性设定:
不涉及DE基因或方向不正确的相关性被视为假阳性
结果
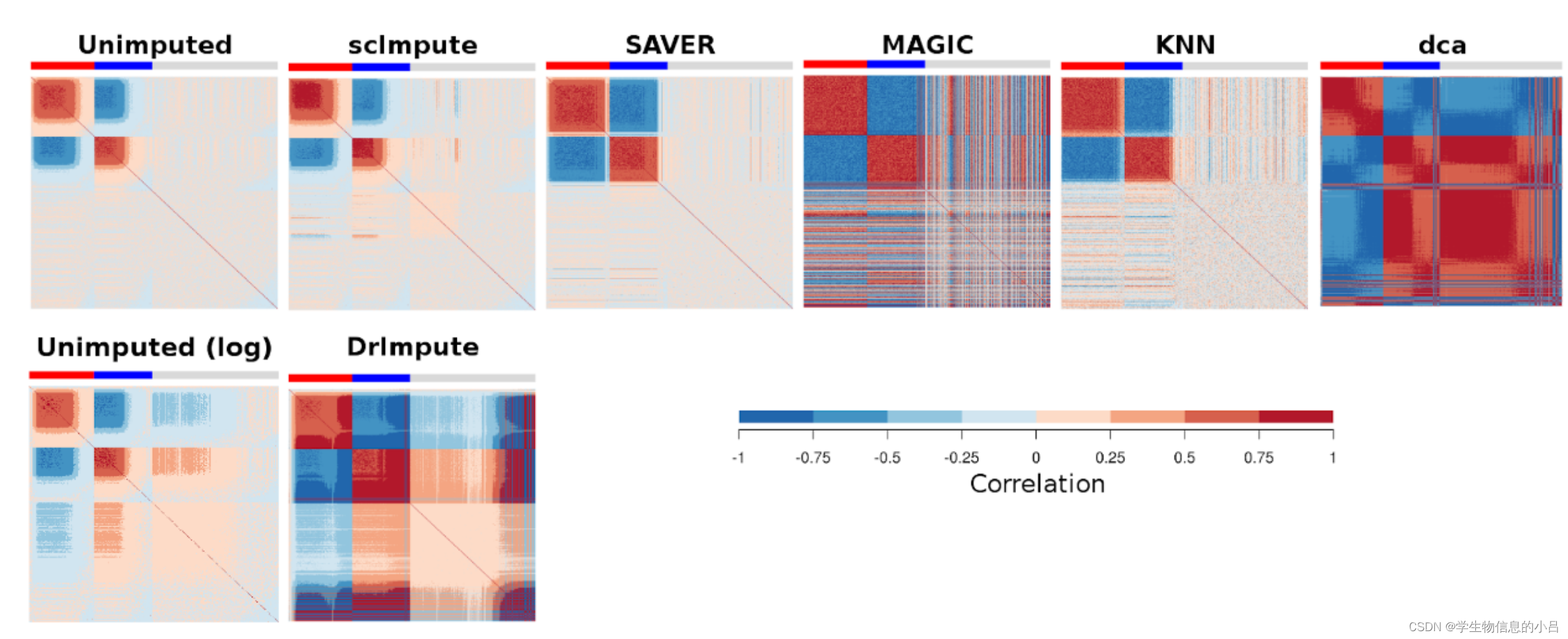
结果说明
所有插补方法都提高了检测低表达DE基因相关性的敏感性。然而,只有SAVER增强了低表达DE基因之间的相关性,而没有在独立绘制的基因之间产生假阳性基因相关性。
2.构造基于Splatter的数据模型
生成60个模拟scRNA-seq矩阵matrix
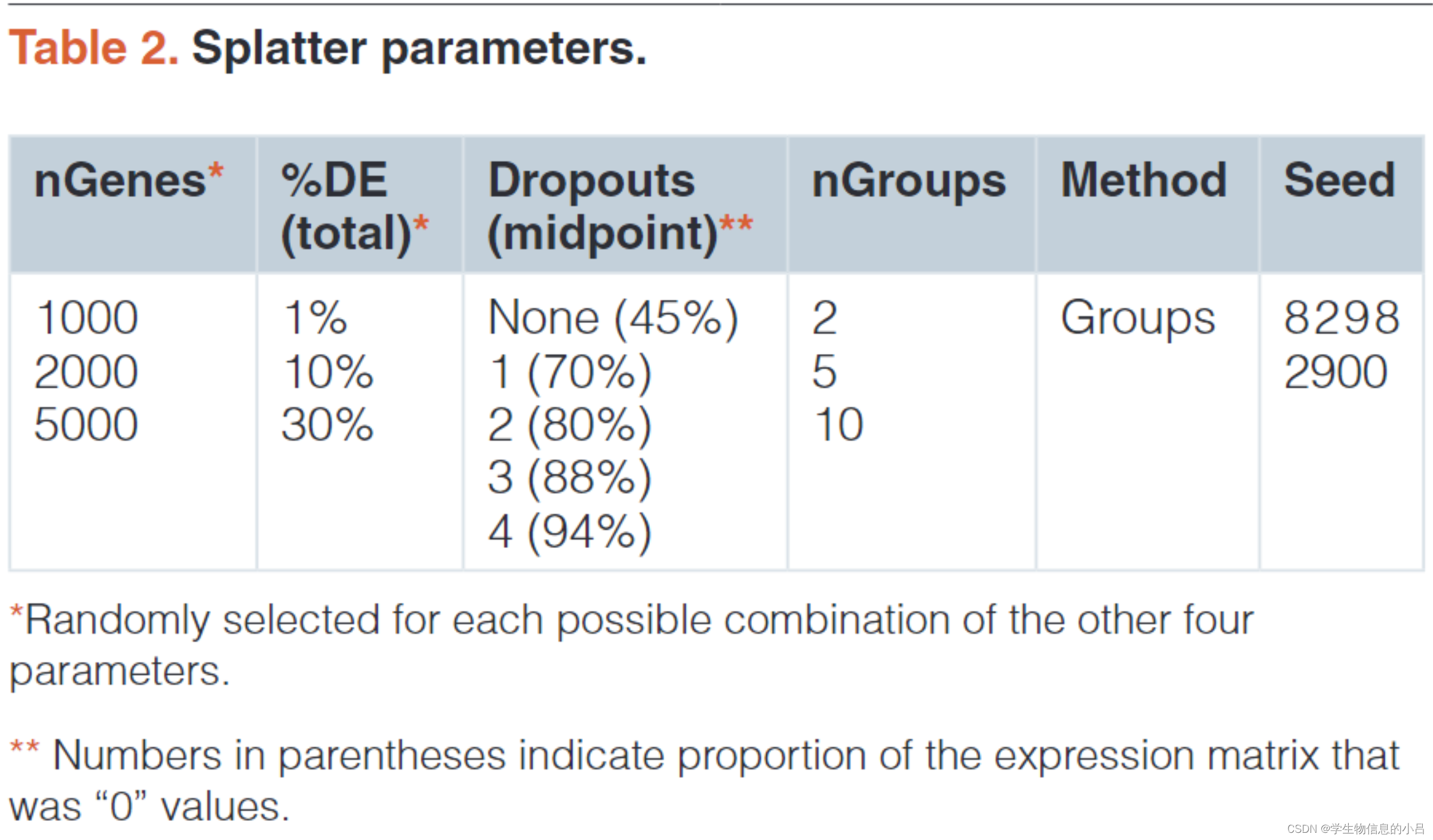
模拟数据集中的DE差异基因占比和dropout率各不相同,此外
每种方法的组也不相同
通过测试各组之间的差异表达基因来评估插补带来的假阳性可能
使用Kruskal Wallis检验来验证插补后数据的分布是否出现变化
真正的差异表达基因定义为:
gene大小为所有成对簇的最大对数2倍变化且在5%FDR后显著的基因才被称为DE gene
假阳性设定
构造的splatter数据集本身具有不同数据的原始值 设定为reference 这个值可以作为ground truth使用
插补前后的数据集本身的DE gene 与真实情况的出入视为假阳性和假阴性来源
结果
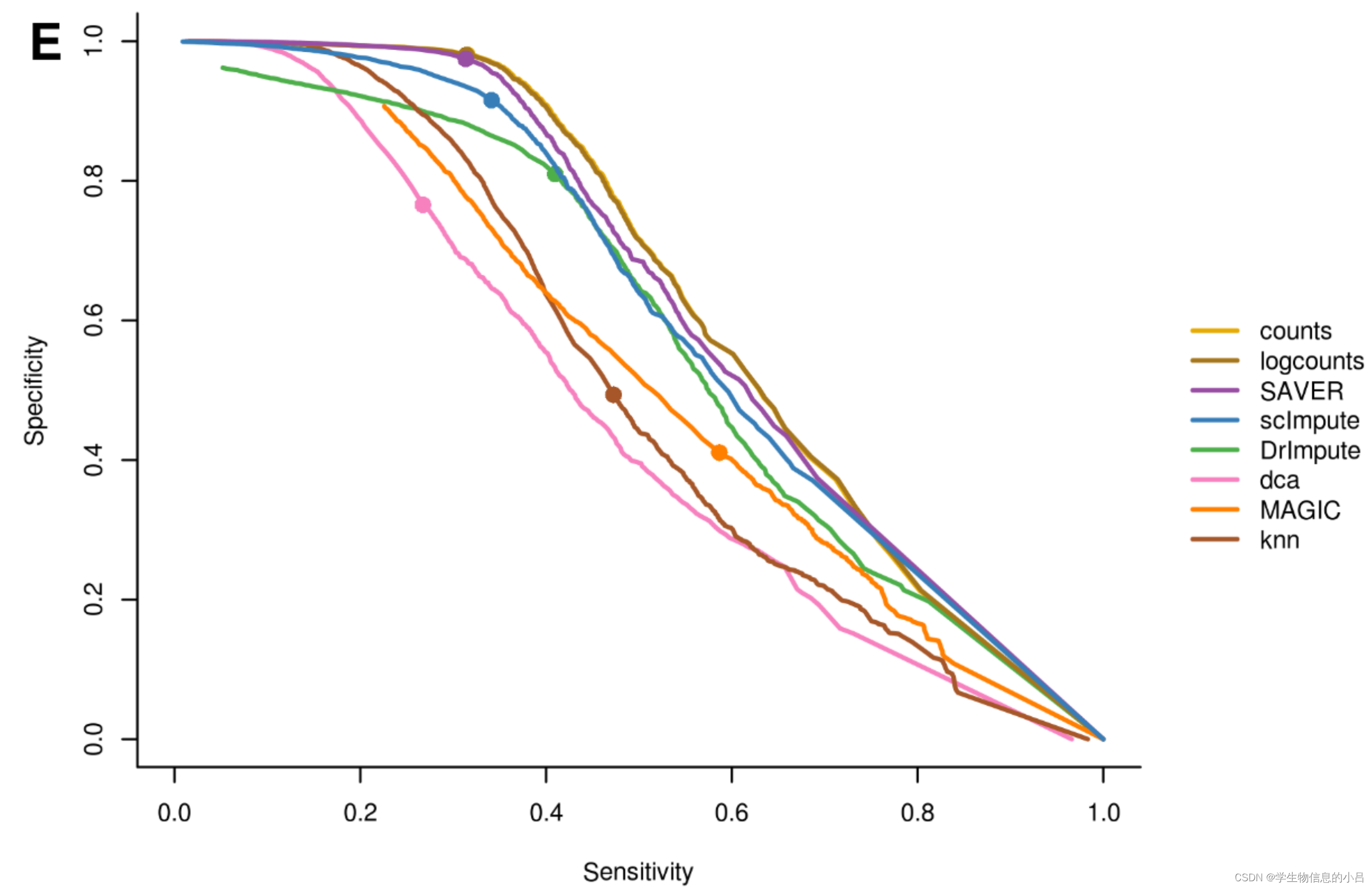
![[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-WPvqDXQo-1665413613958)(https://note.youdao.com/yws/res/4098/WEBRESOURCEd962f266815475908182724581a06e88)][外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传(img-ic962sjV-1665413613959)(https://note.youdao.com/yws/res/4100/WEBRESOURCEf29038aad197a13bd99502195620c03f)]](/i/ll/?i=5dab52d134a04142bd11343e5ae7dc45.png)
结果解读
总的来说,当同时考虑敏感性和特异性时,基于模型的方法比平滑方法表现更好
3.对Tabula Muris数据集进行插补改装
从Tabula Muris中选择了6个10X 12个Smart-seq2的数据集
1.首先做归一化:
至少有两种细胞类型含有>5%的总细胞数目,过滤后有500-5000个细胞(表S1)。对每个数据集进行预处理,以删除占总细胞数小于5%的细胞类型,以及未分配给命名细胞类型的任何细胞。对基因进行过滤,以去除在不到5%的细胞中检测到的基因。
2.然后基于欧氏距离选择每种数据集中最相近的两个细胞类型
3.随后在选定细胞类型中计算基因差异表达
4.应用Mann-Whitney-U检验测试两种选定细胞类型之间的差异表达,评估每个插补引入的假阳性。采用Bonferroni多重检测校正,以确保预期总误报率低于1
5.留下不差异表达的基因,对其进行插补去噪
假阳性设定
插补去噪后进行上述步骤,差异表达基因如果存在即代表假阳性出现。
结果
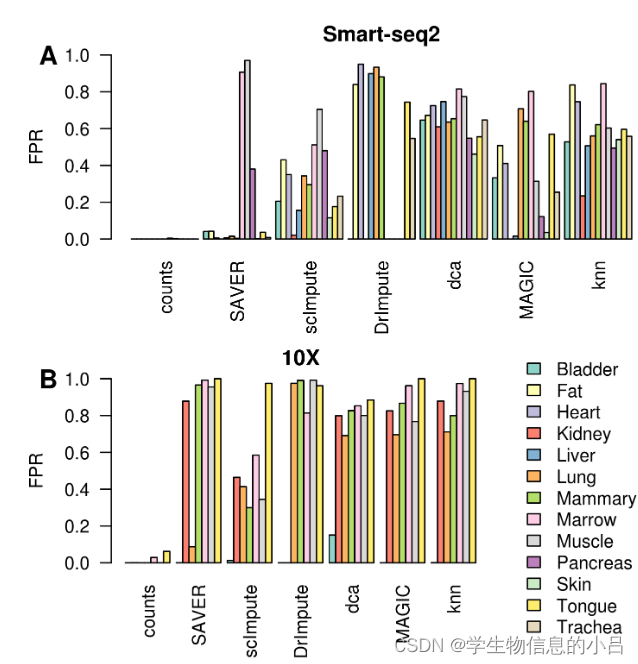
结果解读
同一种方法在不同数据集上假阳性可能性不同。
4.构造可再现性的marker指标
上一步骤讲述的Tabula Muris数据集在该步骤继续使用
通过Mann-Whitney-U检验方法来确定标记基因Marker
Marker gene是一种不同于DE gene的指标 每一个gene都会被分配一个自己的marker所属细胞类型
判定标准:将基因分配给AUC值最高的细胞类型
使用5%的FDR和超过特定阈值的AUC为每个输入数据集定义重要标记基因
通过这种方法可以将每个基因分配给数据集中的单个细胞类型 而不是全局细胞类型
假阳性设定
设定为marker的gene在插补后是否是可再现的
可再现性分数定义为:
在两个数据集中都是显著标记的、也是同一细胞类型标记的标记的分数
结果
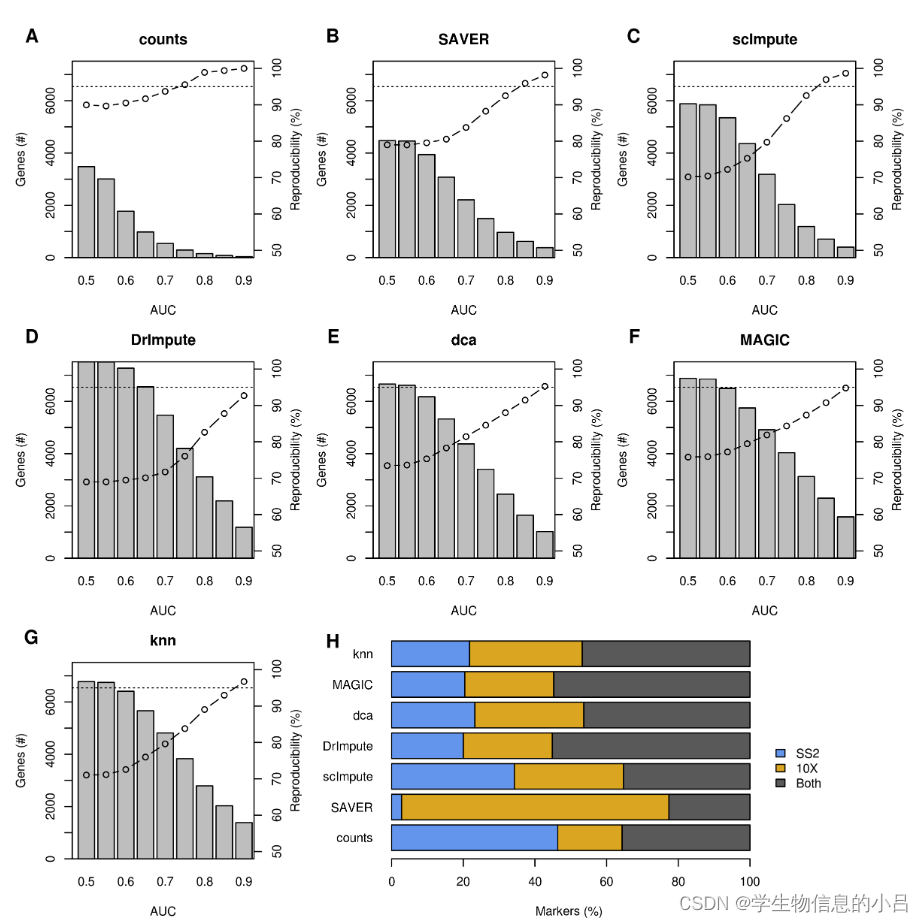
结果解读
存在大量的不可重现的标记marker gene 说明在不同数据集中的可定义为某个细胞类型的marker其实是有差别的。同一个marker gene在不同的数据集中属于不同的细胞类型。
如果不进行插补,两个数据集中95%的显著标记基因在同一细胞类型中高度表达。插补后,根据AUC阈值(可以划归为marker的阈值)的升高,这一数字大幅下降。在估算的Smart-seq2和10X Chromium数据集中,降低幅度阈值会导致更多标记分配给相互矛盾的细胞类型。
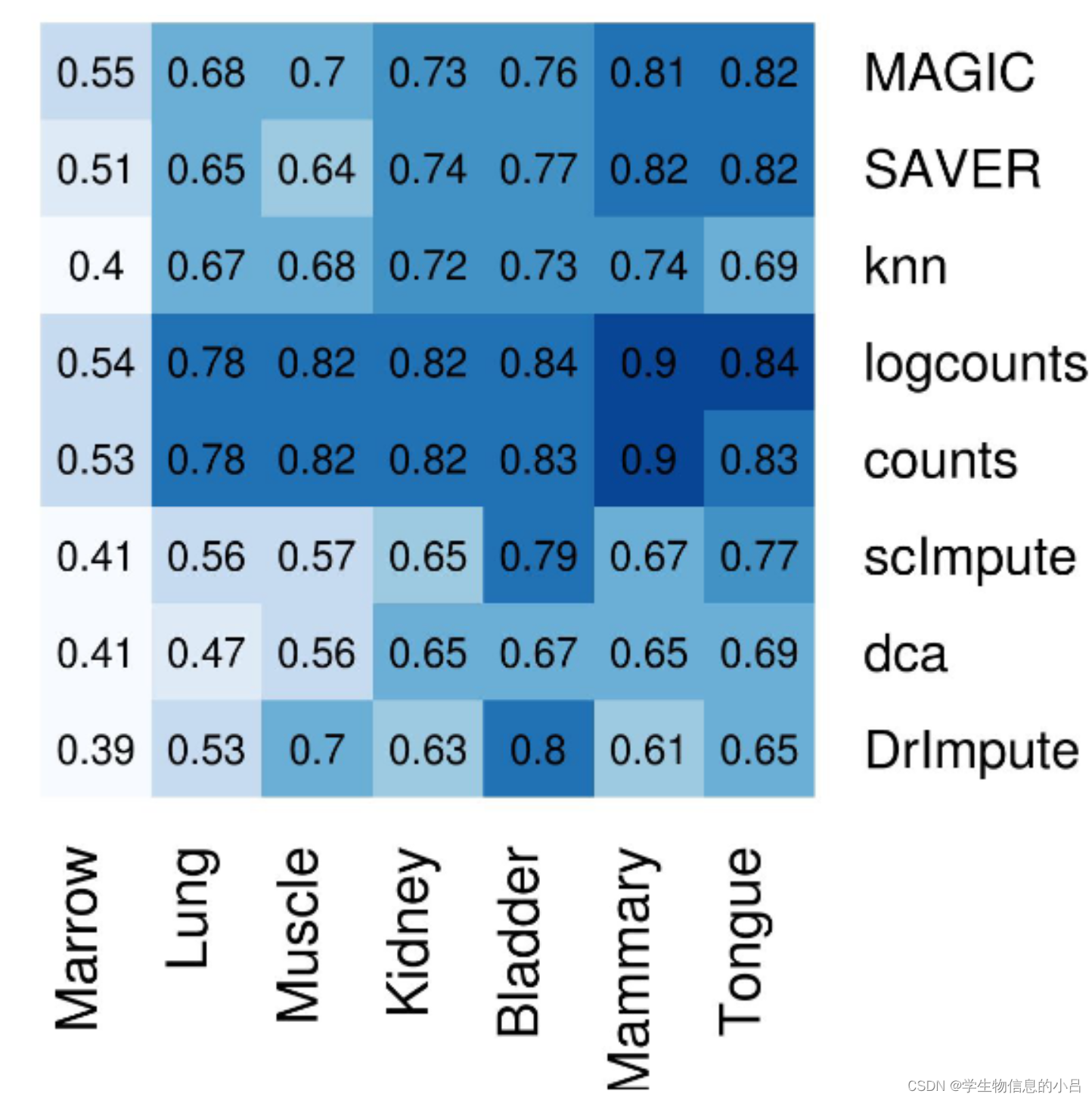
未经插补过的数据实际上获得了最高比例的一致性marker
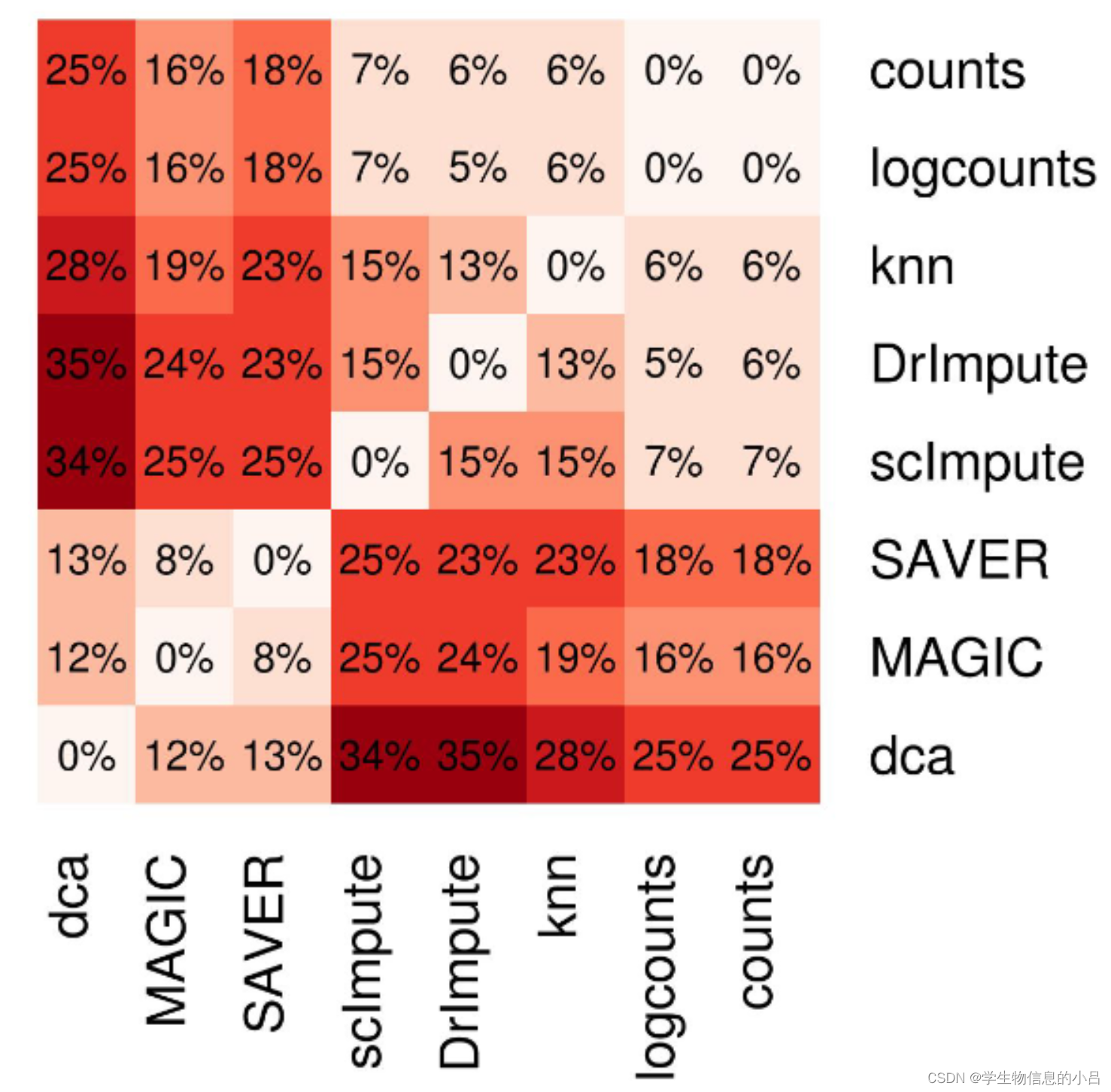
插补之间的marker存在矛盾,同一个数据集中,通过不同插补方法分配给不同细胞类型的重要标记(FDR 5%)的比例亦不相同。
根据所用的插补方法,总共有5-35%的markergene 分配给不同细胞类型。
且存在偏向性 一部分属于MAGIC、SAVER和dca,另一部分属于scImpute、DrImpute和knn-smooth。
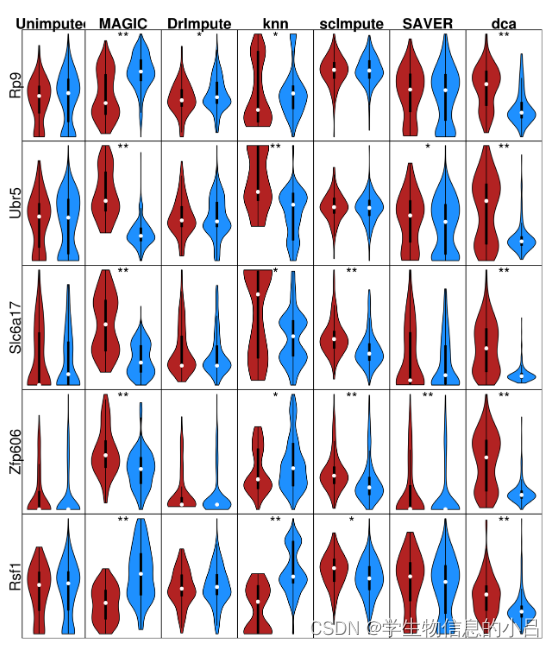
同样的数据集经过不同的插补方法处理后,同一数据集的两种不同细胞(红,蓝)出现了DE基因的假阳性变化。例如,使用MAGIC插补后,Zfp606在PP细胞中的表达高于A细胞,但使用knn光滑插补后则相反。
总结
- 1.各类插补方法都会导致假阳性无可避免的存在
- 2.平衡sensitivity和specificity之间的基本平衡不可靠插补来打破
- 3.真实数据集相比于仿真数据集(splatter)变化更多,一些本来不会产生假阳性的方法在真实数据集上还是会产生假阳性
- 4.不同的插补方法既有利于敏感性,也有利于特异性,但没有一种方法能够全面改善差异表达的检测
- 5.当前单细胞RNASeq插补方法的基本局限性,即仅使用原始数据中的信息。因此,没有获得新的信息,这类似于简单地降低应用于数据的任何统计检验的显著性阈值
- 6.验证多个数据集或多个插补方法的结果再现性可以消除一些假阳性。