大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
2021年03月02日,杜克大学医学中心的分子遗传学和微生物学系Stacy M. Horner教授团队在《Cell Reports》(IF: 9.995)杂志发表了题为“Post-transcriptional regulation of antiviral gene expression by N6-methyladenosine”的研究论文,研究通过MeRIP-seq(m6A-seq)等实验揭示了m6A在抗病毒限制的I型干扰素(interferon,IFN)响应期间对IFN刺激基因(IFN-stimulated genes ,ISG)翻译的转录调控机制。

标题:Post-transcriptional regulation of antiviral gene expression by N6-methyladenosine
时间:2021.03.02
期刊:Cell Reports
影响因子:IF 9.995
技术平台:MeRIP-seq、RNA-seq、Ribo-seq、MeRIP-qRT-PCR、荧光素酶活性分析实验等
研究摘要
I型干扰素(interferon,IFN)通过诱导数百种IFN刺激基因(ISG)来响应病毒感染,必须调节这些ISG的诱导以实现有效和可调控的抗病毒感染响应,但这些基因的转录调控机制尚不明确。本研究首先鉴定了RNA碱基修饰N6-甲基腺苷(m6A)在ISG调控中的作用,通过Ribo-seq和定量质谱法结合m6A免疫沉淀测序(MeRIP-seq)分析,鉴定出包含IFITM1的ISG亚群,其翻译通过m6A和m6A甲基转移酶蛋白METTL3和METTL14增强。研究进一步表明m6A识别蛋白(readers)YTHDF1以m6A结合依赖性方式上调IFITM1表达,且m6A甲基转移酶复合体可以促进I型IFN的抗病毒活性。总之,本研究表明m6A在抗病毒限制的I型IFN响应期间对ISG翻译的转录调控中发挥作用。
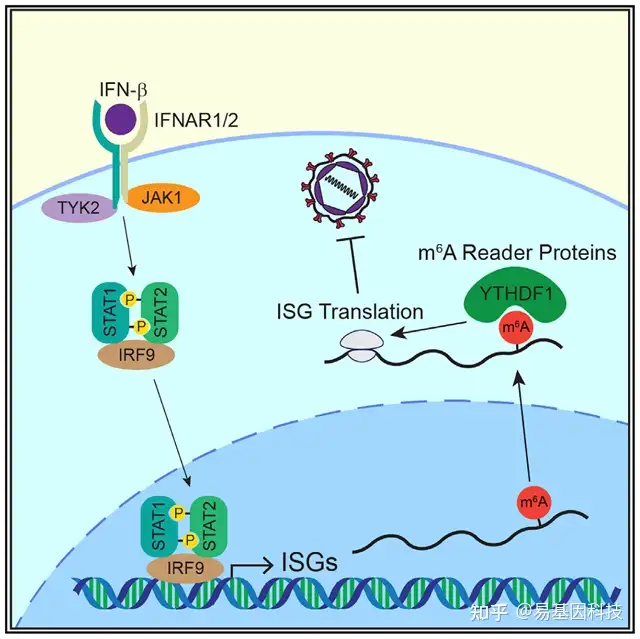
实验结果
(1)METTL3/14调节某些ISG的翻译
IFN-β通过诱导ISG转录形成对病毒感染的响应。为研究m6A是否调节I型IFN响应,本研究对Huh7细胞中m6A甲基转移酶复合体METTL3/14缺失后,IFN-β诱导的几种具有抗病毒功能的ISG表达进行检测。在METTL3/14敲除后,IFN-β诱导的ISG IFITM1和MX1蛋白表达降低(图1A),同时在A549细胞、原代新生儿人真皮成纤维细胞(NHDF)和Huh7细胞中IFN-β处理后的多个时间点(8h、16h和24h)观察到相似结果;但MX1蛋白水平在A549和NHDF细胞中的影响不如在Huh7细胞中强烈。而为响应IFN-β,METTL3/14过表达增加了Huh7细胞中的IFITM1和MX1丰度,但没有增加ISG15和EIF2AK2的丰度(图1B)。METTL3/14调节的ISG IFITM1和MX1在没有IFN-β的情况下不表达,表明METTL3/14动态变化对内源性IFN-β的任何混杂效应可以忽略不计(图1A和1B)。
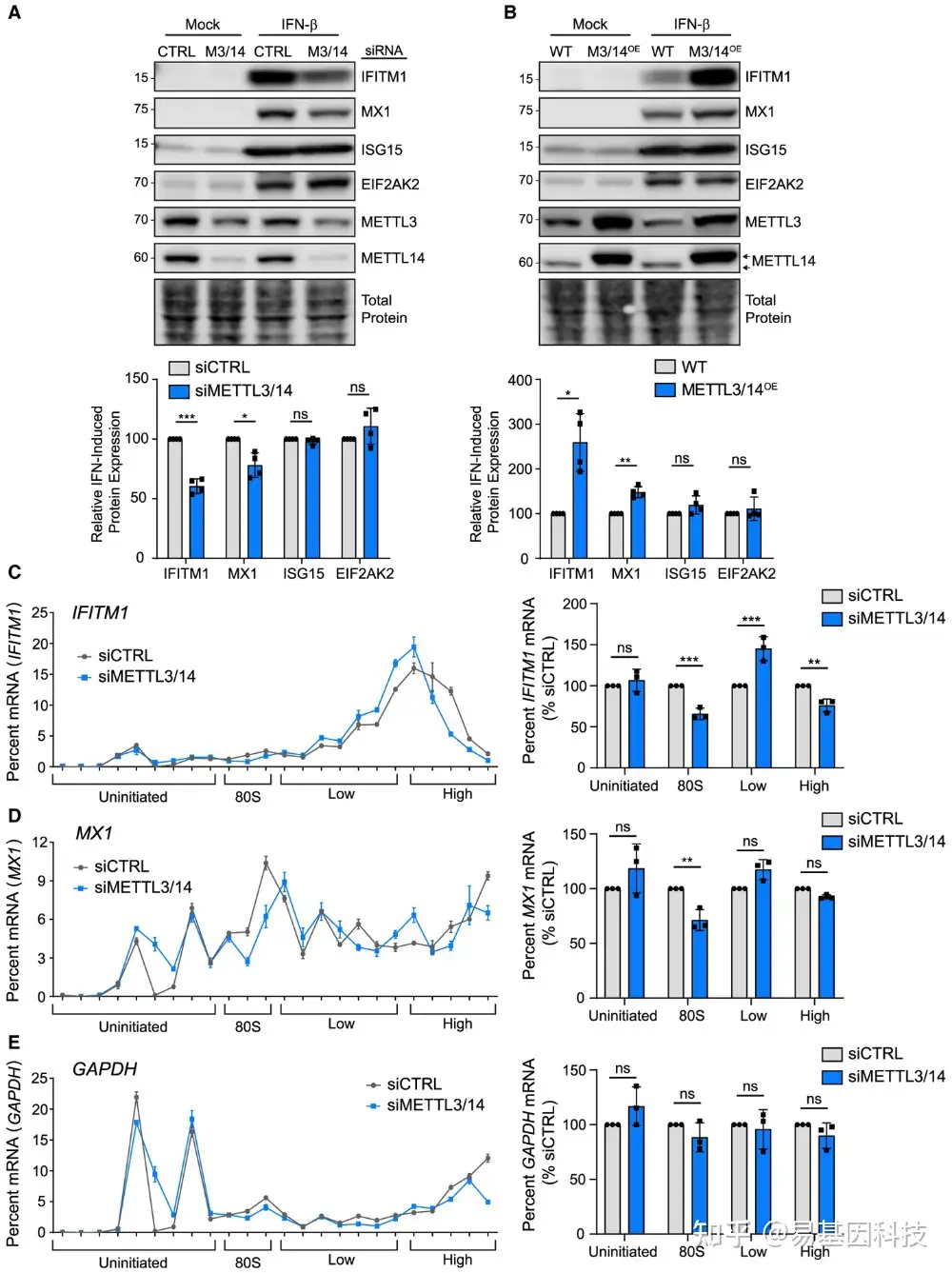
图1:METTL3/14调节某些ISG的翻译
(A-B)在模拟或IFN-β(24h)处理之前,用siRNA转染METTL3/14(M3/14)或对照(CTRL)(A)或稳定过表达FLAG-METTL14(M3/14OE;上箭头表示FLAG-METTL14;下箭头表示内源性METTL14)(B)的Huh7细胞提取物的免疫印迹分析。相对于非靶向对照(siCTRL)+IFN-β(A)或WT+IFN-(B),对A和B中4个重复的相对ISG表达进行定量。
(C-E)用CTRL或METTL3/14 siRNA转染后经IFN-β处理的Huh7细胞提取物中分离24个蔗糖梯度级分中的IFITM1(C)、MX1(D)和GAPDH(E)mRNA相对百分比qRT-PCR分析。未初始化(游离、40S和60S亚基)、初始化(80S)、低分子量或高分子量多核糖体。右边的图表显示了IFITM1、MX1或GAPDH组合级分中的mRNA百分比。将各部分的百分比相加得出各类别的总百分比。
数值为4个生物学重复(A和B)的平均值±SEM,3个技术重复平均值±SD,表示3个实验(C–E,左图)和3个生物学重复的平均值±SEM(C–E,右图)。*非配对Student's t检验(A和B)和Sidak多重比较检验的双向方差分析(C–E),*p<0.05,***p<0.01,***p<0.005,ns,不显著。
为研究METTL3/14如何调节某些ISG的蛋白水平,研究首先分析METTL3/14缺失是否导致ISG mRNA对IFN-β响应减少。同时,在IFN-β处理后使用RNA测序(RNA-seq),结果显示METTL3/14缺失对核心ISG的mRNA表达丰度影响很小,表明ISG RNA稳定性不受METTL3缺失的影响。
由于METTL3/14缺失导致IFITM1和MX1蛋白减少而不影响其转录水平,本研究分析METTL3/14是否调节其蛋白稳定性。研究结果表明METTL3/14不能检测到Huh7细胞中这些ISG的出核或蛋白质稳定性。
为了检测METTL3/14是否调节IFITM1翻译,作者检测了其在对照细胞或IFN-β处理后METTL3/14缺失的细胞中的多核糖体(polysome)百分比。结果显示METTL3/14缺失不改变总多核糖体水平,但METTL3/14缺失会导致80S级分中IFITM1 mRNA水平较低,且从重多核糖体级分转换为轻多核糖体级分(图1C),表明METTL3/14缺失后IFITM1翻译受损。另外,在MX1(图1D)中也观察到类似但不太明显的变化,但在GAPDH(图1E)中没有观察到。总之结果表明,METTL3/14调节某些ISG的翻译,如IFITM1和MX1。
(2)METTL3/14调节的ISG被m6A修饰
为确定METTL3/14调节的ISG IFITM1和MX1以及其他ISG是否被m6A修饰,研究通过使用甲基化RNA免疫沉淀测序(MeRIP-seq)在Huh7细胞中绘制了IFN诱导的转录组m6A图谱,在鉴定出ISG后,将m6A免疫沉淀后reads覆盖率的peaks,与使用MeTDiff m6A peaks calling的input进行比较。结果显示mRNA peaks在编码序列末端和3′UTR起始位点富集(图2A)。peaks内最富集的RNA序列motif为[U/A]GGAC,这与DRAmC已知m6A motif匹配(图2A)。约85%的ISG(分类为IFN后上调超过4倍的ISG)被m6A修饰,而Huh7细胞表达转录组中比例为74%(平均覆盖率≥10)(图2B)。该结果与先前的研究一致,该研究发现ISG以与转录组相似的百分比进行m6A修饰。m6A修饰的ISG百分比在其他类别的ISG中相似,包括脊椎动物中进化保守的“核心”ISG和具有已知抗病毒功能的核心ISG的14个亚群(图2B)。接下来,利用MeRIP-seq数据生成了IFITM1、MX1、ISG15和EIF2AK2图,并使用m6A peaks calling 方法MeTDiff和meRIPPer揭示METTL3/14调节基因IFITM1和MX1具有m6A peaks(图2C和2D),而ISG15和EIF2AK2没有m6A peaks(图2E和2F),这些图表明ISG15的3'UTR也可能包含m6A位点(图2E)。将MeRIP-seq实验中ISG的m6A状态与已发表的研究数据进行比较,比较结果显示在所有研究中核心抗病毒ISG的m6A状态预测一致。dsDNA处理有效地激活IFN产生并诱导本实验中发现的相同核心抗病毒ISG的m6A修饰。HCMV感染也导致某些ISG的m6A修饰,这种病毒编码抑制IFN信号因子;因此ISG可能不像dsDNA或IFN-β处理那样强烈诱导。m6A在许多ISG上的存在表明m6A可以调节抗病毒I型IFN响应。
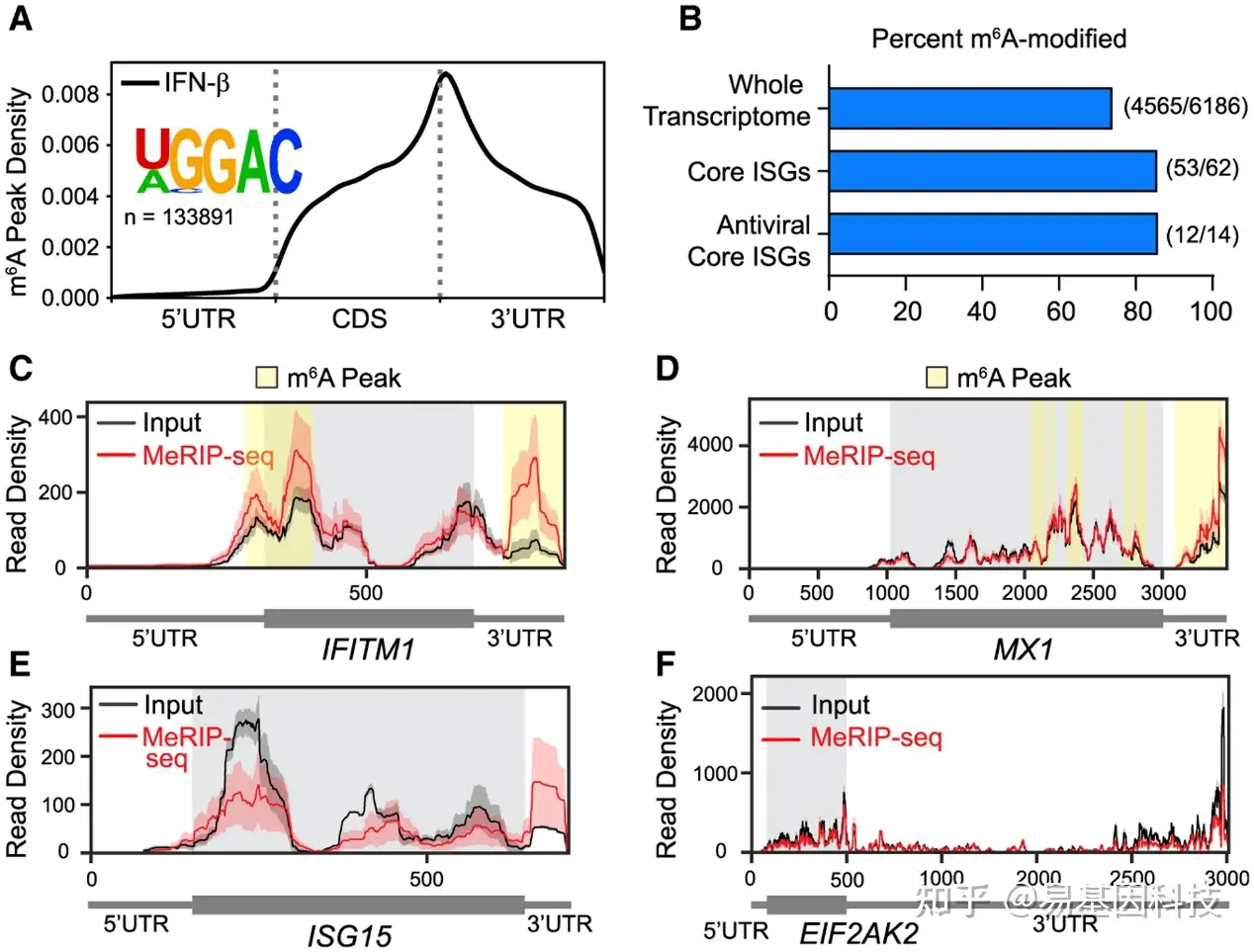
图2:METTL3/14调节的ISG由m6A修饰
(A) 在IFN-β处理(8h)后,转录组范围内的m6A分布Metagene图,绘制了具有统计学意义的peaks 中DRACH motif位点相对位置,以及peaks中最富集的motif。
(B) 在表达的转录组中由m6A修饰的基因百分比,响应于IFN-β处理(ISG)的mRNA诱导≥4倍的基因,脊椎动物中保守的核心ISG,或具有抗病毒功能的核心ISG亚群。
(C-F)在IFITM1(C)、MX1(D)、ISG15(E)和EIF2AK2(F)转录本中MeRIP(红色)和input(黑色)reads覆盖图。生物学重复的差异由reads覆盖范围周围的红色和黑色阴影表示,灰色阴影表示编码序列,黄色阴影表示由MeTDiff和meRIPPer的m6A peaks calling软件。所有分析均在3个生物学重复上进行。
(3)IFITM1在3′UTR中的m6A修饰增强了其翻译
m6A增强了某些mRNA的翻译。具体而言,m6A识别蛋白(readers)YTHDF1在3′UTR内识别m6A,并与真核翻译起始因子(如eIF3)结合,以增强m6A修饰转录本的翻译。为METTL3/14对ISG的翻译调节是否是通过m6A诱导,研究使用IFITM1作为METTL3/14调节的ISG模型。首先确定METTL3/14缺失对IFITM1 m6A修饰的影响。MeRIP-qRT-PCR结果表明IFITM1 mRNA在m6A阴性ISG EIF2AK2和m6A阴性合成RNA中富集,证实其含有m6A。METTL3/14缺失降低了IFITM1 mRNA的m6A富集,但没有降低m6A阴性EIF2AK2转录本或m6A阴性合成RNA的富集(图3A和3B),数据表明IFITM1是由METTL3/14修饰的m6A。
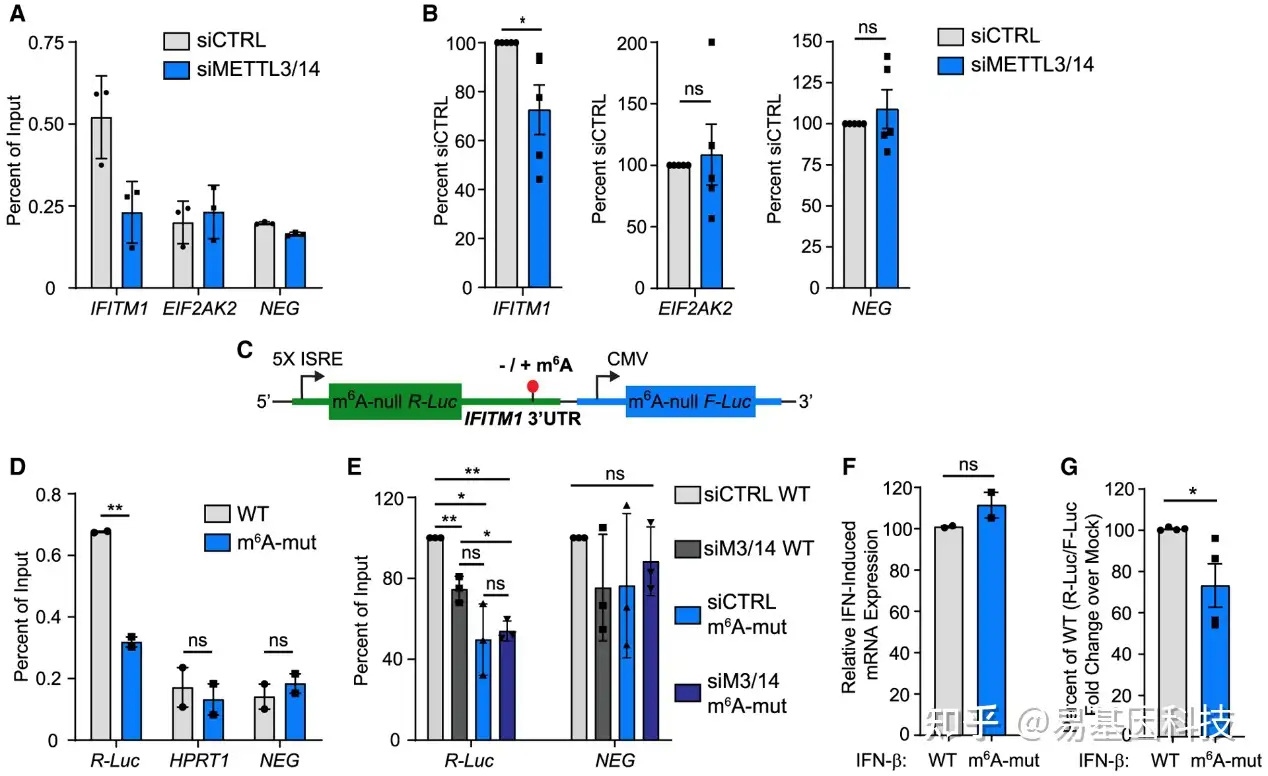
图3:IFITM1在 3'UTR中的m6A修饰增强了翻译
(A) 在用siCTRL或METTL3/14 siRNA处理并掺入m6A阴性(NEG)寡核苷酸的Huh7细胞中,由IFN-β(8h)诱导的ISG的相对m6A水平的代表性MeRIP qRT-PCR分析。
(B) A中5个生物学重复每个基因的相对富集百分比,归一化为siCTRL。
(C) 野生型(WT)和突变型ISRE-m6A缺失的Renilla荧光素酶(R-Luc)IFITM1 3'UTR报告基因示意图,该报告基因也从单独的启动子表达m6A缺失的萤火虫荧光素酶(F-Luc)。
(D)经IFN-β(8h)处理的转染Huh7细胞的WT和m6A-mut IFITM1 3'UTR报告RNA的相对m6A水平的MeRIP-qRT-PCR分析。
(E)用siRNA转染的Huh7(24h)、然后进行报告基因转染(24h)和用IFN-β处理(8h)的WT和m6A-mut IFITM1 3'UTR报告基因RNA的相对m6A水平的MeRIP-qRT-PCR分析,。
(F) 报告基因转染(24h)和IFN-β处理(8h)后,在Huh7细胞中归一化为HPRT1的WT和m6A-mut IFITM1 3'UTR报告基因mRNA表达的qRT-PCR分析。
(G) IFN-β诱导的WT和m6A-mut IFITM1 3′UTR报告基因中的相对萤光素酶活性(R-Luc/F-Luc)(相对于模拟8h)。
在鉴定出IFITM1是m6A修饰之后,接下来生成了一个荧光素酶报告基因,其中包含IFN刺激的响应元件(ISRE)-启动子驱动的Renilla萤光素酶,所有DRAC motif被敲除(m6A-null R-Luc),然后与野生型(WT)IFITM1 3′UTR或类似的3′UTR序列结合,IFITM1中3′UTR m6A peaks中的四个推定m6A motif从A→G转换(m6A-mut)(图3C)。分析数据结果表明, METTL3/14通过向3'UTR添加m6A修饰来调节IFITM1翻译,并且IFITM1 3'UTR内的m6A修饰足以增强其翻译。
(4)YTHDF1以m6A依赖性方式增强IFITM1蛋白表达
为检测YTHDF1是否诱导m6A对ISG的翻译促进作用,研究人员在Huh7细胞中稳定过表达YTHDF1(H7Y1)或m6A结合缺陷型YTHDF1蛋白(H7Y1mut)并分析其相对于亲本Huh7细胞(H7)在24h后IFN诱导的ISG表达。YTHDF1过表达增加响应IFN-β的IFITM1蛋白表达,而m6A结合缺陷型YTHDF1蛋白(H7Y1mut)的过表达不增加IFITM1丰度(图4A和4B)。且野生型(WT)和突变型YTHDF1过表达不会显著影响IFN-β处理后的IFITM1 mRNA水平,表明YTHDF1不直接调控IFN信号或IFITM1 mRNA稳定性(图4C)。YTHDF1过表达显著改变IFN诱导的含m6A的ISG MX1表达,或不含m6A的ISG ISG15和EIF2AK2表达(图4A和4B)。而在IFN-β处理后,YTHDF1缺失会导致IFITM1蛋白表达降低,但MX1、ISG15和EIF2AK2未受影响(图4D)。另外,WT YTHDF1与IFITM1、MX1、ISG15和m6A阳性对照SON的转录本结合,而m6A结合缺陷型YTHDF1突变蛋白则没有结合。含有非m6A的mRNA EIF2AK2和RPL30不与任一蛋白结合(图4E和4F)。总之,这些结果表明YTHDF1与IFITM1 mRNA上的m6A结合,且通过其m6A结合活性增强其翻译是必要和充分的。YTHDF1与ISG15 mRNA明显的m6A依赖性结合表明ISG15 mRNA实际上是m6A修饰。在ISG15 mRNA的input reads与MeRIP-seq reads的关联图揭示了其3'UTR中m6A富集的潜在区域(图2E)。因此,YTHDF1在促进翻译中具有转录本特异性作用,因为它结合了IFITM1、MX1和ISG15的转录本,但其过表达仅足以显著增加IFITM1的蛋白生成。
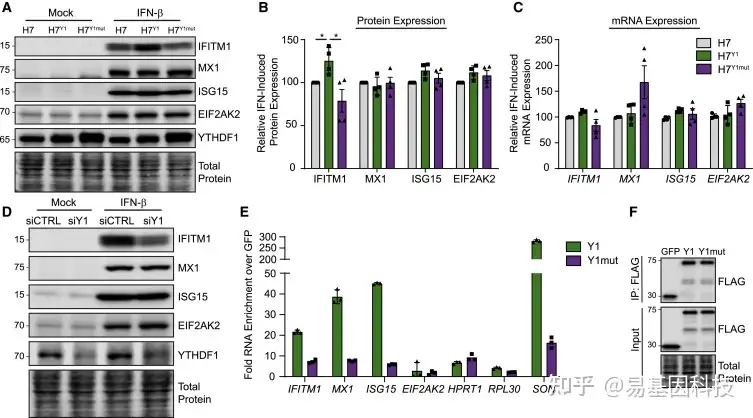
图4:YTHDF1以m6A依赖性方式增强IFITM1蛋白表达
(A) 在模拟或IFN-β(24h)处理后稳定过表达FLAG-YTHDF1 WT(Huh7Y1)或FLAG-YTHDF1 W465A(Huh7Y1mut)的Huh7细胞提取物的免疫印迹分析。
(B) A的3个独立实验中IFN-β处理后ISG表达定量,归一化为总蛋白并相对于siCTRL绘图。
(C) 在IFN-β(24h)处理后稳定过表达FLAG-YTHDF1 WT(Huh7Y1)或W465A(Huh7Y1mut)的Huh7细胞中归一化为HPRT1的ISG mRNA表达的qRT-PCR分析。
(D) 在模拟或IFN-β(24h)处理之前,用siRNA转染YTHDF1(siY1)或siCTRL的Huh7细胞提取物的免疫印迹分析。数据代表3个独立的生物学实验。
(E) 与IFN-β(8h)处理后Huh7细胞的FLAG-GFP相比,FLAG-YTHDF1 WT(Y1)或W465A(Y1mut)的免疫沉淀(IP)后mRNA富集的qRT-PCR分析。将IP值归一化为input值并绘制为GFP的倍数富集。
(F) E中使用的FLAG免疫沉淀和input级分的免疫印迹分析。
(5)METTL3/14和m6A促进ISG亚群的翻译
为鉴定蛋白表达受METTL3/14调节的其他ISG,使用基于定量质谱的蛋白质组学和氨基酸稳定同位素标记(SILAC)来比较IFN-β处理后siCTRL和siMETTL3/14细胞的蛋白质组。与siCTRL相比,siMETTL3/14对蛋白丰度的影响集中在大多蛋白质的对数比为0,表明METTL3/14缺失对IFN-β处理后的蛋白质水平没有整体影响。在先前的RNA-seq实验中,将ISG定义为通过IFN-β处理上调>2倍的基因来确定哪些蛋白是ISG。尽管质谱法检测ISG有限(n=18),但鉴定出一些METTL3/14调节的ISG(图5A;MS)。METTL3/14缺失后,大多数这些ISG的蛋白表达降低,且这些ISG包括先前鉴定的m6A修饰的IFITM1(对应于IFITM1/2/3的肽)和MX1,以及其他抗病毒ISG如OAS2和不同的HLA-C链(图5A),均由m6A修饰。通过将这些数据与之前的RNA-seq实验数据进行比较,结果表明METTL3/14对这些ISG蛋白水平的影响不由其mRNA表达调节来确定,因为METTL3/14缺失后,本实验中在蛋白水平上降低的ISG的mRNA丰度没有降低,表明翻译调控如先前对IFITM1和MX1的多核糖体分析所表明结果一致(图1C和1D;图5A,RNA)。并非所有通过质谱法鉴定的m6A修饰的ISG都受METTL3/14缺失调节(图5A;m6A),表明METTL3/14和m6A调节ISG的亚群并支持其蛋白表达。
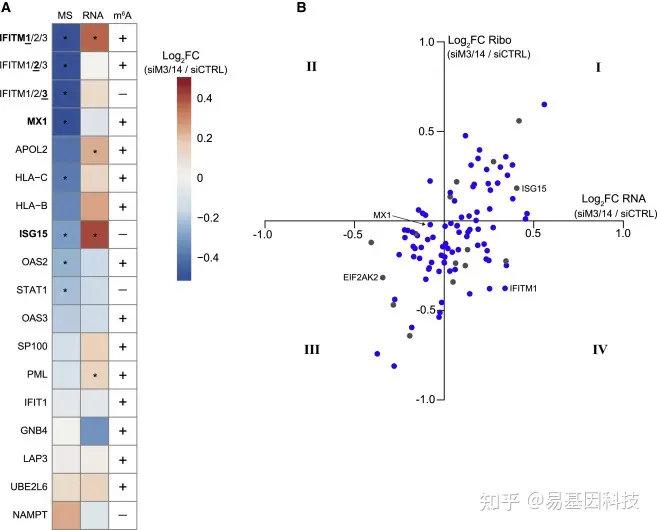
图5:METTL3/14调节ISG亚群的翻译
(A) 3列热图显示了IFN-β处理后METTL3/14缺失对Huh7细胞中ISG表达的影响。第一列显示定量质谱法检测蛋白质估计值的log2倍变化(siMETTL3/14与siCTRL + IFN-β 24 h;n=2个生物学重复)。第二列显示独立RNA-seq实验(siMETTL3/14与siCTRL + IFN-β 8 h;n=3个生物学重复)的mRNA reads的log2倍变化。第三列显示MeRIP-seq数据的m6A状态(+表示m6A阳性;-表示m6A阴性)(+IFN-β 8h;n=3个生物学重复)。基因包括RNA-seq的IFN诱导超过2倍的任何ISG,这些ISG也可以通过质谱法检测到,其他图中的ISG以粗体显示。由于IFITM1/2/3相似,因此使用此表示法表示从该蛋白质家族中检测到的肽;RNA-seq倍数变化和m6A状态对应于带下划线的数字,调整后*p<0.05。
(B) METTL3/14缺失对ISG表达影响的四象限散点图。y轴是Ribo-seq的核糖体保护片段的log2倍变化(siMETTL3/14超过siCTRL),x轴是来自独立RNA-seq实验的mRNA读数的log2倍变化(siMETTL3/14与siCTRL)。m6A修饰(蓝色)或m6A阴性(灰色)基因。对其他图中的ISG进行了标记。
(6)METTL3/14增强了IFN响应的抗病毒作用
METTL3/14在I型IFN响应期间增强ISG亚群表达,表明它可能是最佳抗病毒所必需。研究人员检测了METTL3/14动态变化后I型IFN限制负义链RNA病毒水泡性口炎病毒(vesicular stomatitis virus,VSV)感染的能力。VSV基因组包含了m6Am帽修饰,但由于这种修饰沉积不受METTL3/14调控,因此VSV复制可能不会受到METTL3/14动态变化的直接影响,而对VSV复制的任何影响可能由宿主转录本的甲基化介导。通过使用小干扰RNA(siRNA)或过表达来干扰METTL3/14的表达,然后使用显微镜在存在和不存在低剂量IFN-β预处理(6h;40U/mL)的情况下,检测感染后6小时被VSV感染的细胞百分比。在感染后早期时间点检测VSV感染,可以在感染直接诱导的ISG细胞上调之前检测到病毒复制。结果显示,在没有IFN-β预处理的情况下,任何条件下都没有看到VSV诱导ISG(图6A和6B)。通过免疫印迹或定量感染细胞百分比分析来检测VSV复制,在没有IFN-β处理情况下,METTL3/14缺失或过表达没有变化(图6)。在IFN-β预处理后,METTL3/14缺失导致METTL3/14调节的ISG表达降低(图6A),而METTL3/14过表达则上调其表达(图6B)。尽管IFN-β预处理减少了VSV病毒复制,但METTL3/14缺失降低了IFN-β限制VSV的能力,而METTL3/14过表达则会增强IFN介导的VSV限制(图6)。总之这些数据表明METTL3/14增强了I型IFN的抗病毒特性,是有效IFN介导的抗病毒响应所必需的。
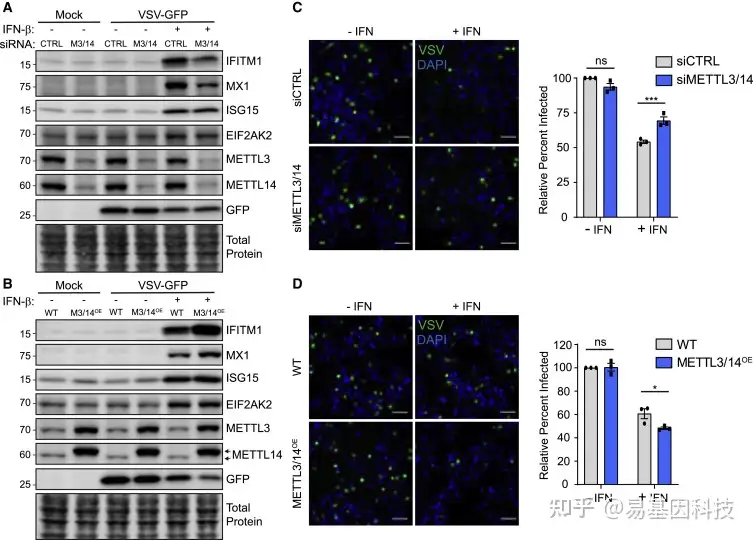
图6:METTL3/14增强了I型IFN响应的抗病毒作用
(A-B) 用siRNA(A)转染或稳定过表达FLAG-METTL14的Huh7细胞提取物的代表性免疫印迹分析(n=3),其也增强METTL3表达(M3/14OE);用IFN-β(6h)或模拟物处理,然后进行VSV感染(MOI=2;6h)(B)。箭头表示FLAG-METTL14(上)和内源性METLL14(下)。
(C-D) 用siCTRL或METTL3/14 siRNA(C)或稳定过表达FLAG-METTL14(METTL3/14OE;D)处理的Huh7细胞的代表性显微图,这些细胞用IFN-β预处理(6h),然后进行VSV感染(MOI=2;6h),对3个独立实验感染的细胞百分比进行定量,每个条件5个视野,每个视野>150个细胞,归一化为未经IFN处理的siCTRL或WT,如右图所示。比例尺:100μm。
关于易基因RNA m6A甲基化测序(MeRIP-seq)技术
易基因MeRIP-seq技术利用m6A特异性抗体富集发生m6A修饰的RNA片段(包括mRNA、lncRNA等rRNA去除所有RNA),结合高通量测序,可以对RNA上的m6A修饰进行定位与定量,总RNA起始量可降低至10μg,最低仅需1μg总RNA。广泛应用于组织发育、干细胞自我更新和分化、热休克或DNA损伤响应、癌症发生与发展、药物响应等研究领域;可应用于动物、植物、细胞及组织的m6A检测。
大样本量m6A-QTL性状关联分析,传统MeRIP单个样品价格高,通常难以承担。易基因开发建立MeRIP-seq2技术,显著提成IP平行性,实现不同样本间相对定量,降低检测成本。
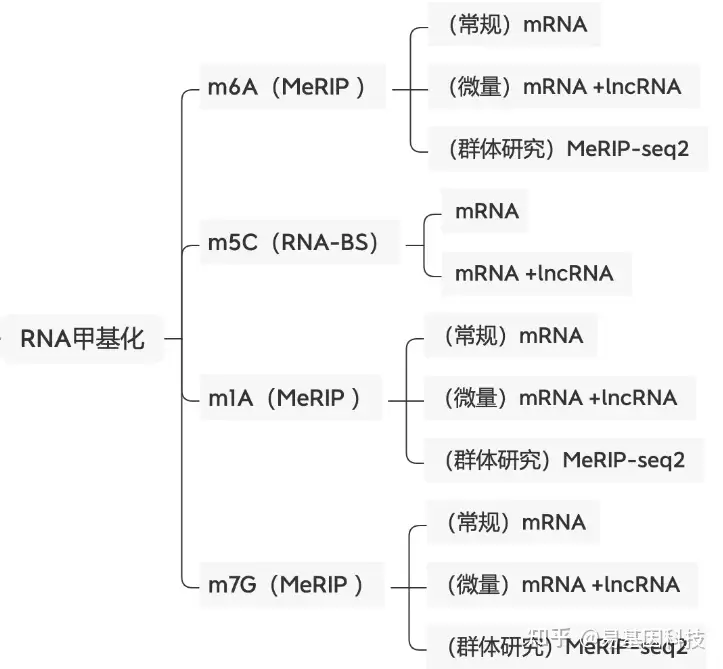
易基因提供适用于不同科研需求的MeRIP技术:
- m6A甲基化-常量mRNA 甲基化测序(MeRIP-seq)
- m6A甲基化-常量mRNA +lncRNA甲基化测序(lnc-MeRIP-seq)
- m6A甲基化-微量mRNA +lncRNA甲基化测序(Micro-lnc-MeRIP-seq)
- 高通量m6A甲基化-常量mRNA甲基化测序(MeRIP-seq2)
技术优势:
- 起始量低:样本起始量可降低至10-20μg,最低仅需1μg总RNA;
- 转录组范围内:可以同时检测mRNA和lncRNA;
- 样本要求:可用于动物、植物、细胞及组织的m6A检测;
- 重复性高:IP富集重复性高,最大化降低抗体富集偏差;
- 应用范围广:广泛应用于组织发育、干细胞自我更新和分化、热休克或DNA损伤响应、癌症的发生与发展、药物响应等研究领域。
研究方向:
m6A甲基化目前主要运用在分子机制的理论性研究
- 疾病发生发展:肿瘤、代谢疾病(如肥胖/糖尿病)、神经和精神疾病(如阿尔兹海默症/抑郁症)、炎症…
- 发育和分化:早期胚胎发育、个体/组织/器官生长发育、干细胞分化与命运决定、衰老
- 环境暴露与响应:污染、抗逆、生活方式
关于m6A甲基化研究思路
(1)整体把握m6A甲基化图谱特征:m6A peak数量变化、m6A修饰基因数量变化、单个基因m6A peak数量分析、m6A peak在基因元件上的分布、m6A peak的motif分析、m6A peak修饰基因的功能分析
(2)筛选具体差异m6A peak和基因:差异m6A peak鉴定、非时序数据的分析策略、时序数据的分析策略、差异m6A修饰基因的功能分析、差异m6A修饰基因的PPI分析、候选基因的m6A修饰可视化展示
(3)m6A甲基化组学&转录组学关联分析:Meta genes整体关联、DMG-DEG对应关联、m6A修饰目标基因的筛选策略
(4)进一步验证或后期试验
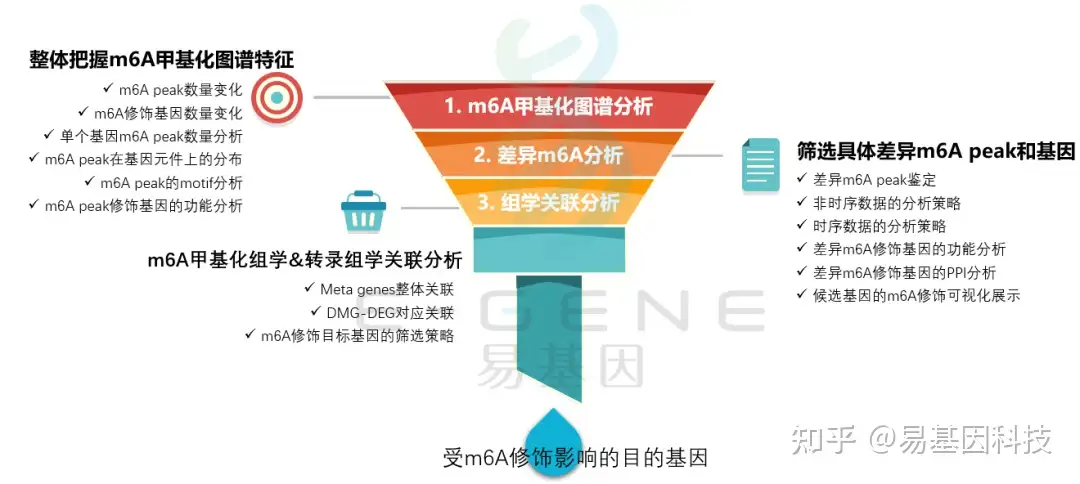
易基因科技提供全面的RNA甲基化研究整体解决方案。
参考文献:
McFadden MJ, McIntyre ABR, Mourelatos H, Abell NS, Gokhale NS, Ipas H, Xhemalçe B, Mason CE, Horner SM. Post-transcriptional regulation of antiviral gene expression by N6-methyladenosine. Cell Rep. 2021 Mar 2;34(9):108798.
相关阅读:
干货:m6A RNA甲基化MeRIP-seq测序分析实验全流程解析
项目文章 | 90天见刊,易基因m6A RNA甲基化(MeRIP)+转录组组学研究
标签:IFITM1,病毒基因,14,seq,METTL3,m6A,IFN,ISG From: https://www.cnblogs.com/E-GENE/p/17337585.html