大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
结直肠癌(Colorectal cancer,CRC)是一种复杂的多阶段疾病,由基因突变和表观遗传改变相互作用引起。组蛋白H3K36三甲基转移酶SET结构域2 (SETD2)是一种表观遗传信号分子,在结直肠癌中突变率为5%。SETD2在氧化偶氮甲烷(Azoxymethane,AOM)/葡聚糖硫酸钠盐(Dextran sodium sulfate, DSS)(AOM/DSS)处理的人类结直肠癌和小鼠发育中表达降低。SETD2缺失促进了CRC进展,SMAD家族成员4 (SMAD4)在结直肠癌中的突变率为14%,SMAD4消融导致结直肠癌。SETD2和SMAD4的共突变可预测晚期结直肠癌。然而,对于SETD2和SMAD4的潜在协同作用知之甚少。
2023年11月14日,上海交通大学生物医学工程学院李力研究员和同济大学附属东方医院鲁兵医生合作在《Clin Transl Med》杂志发表题为“Loss of SETD2 aggravates colorectal cancer progression caused by SMAD4 deletion through the RAS/ERK signalling pathway”的研究论文,该研究以小鼠结直肠癌组织和SW620细胞为研究对象,通过染色质免疫共沉淀测序(ChIP-seq)和对应的转录组测序(RNA-seq)分析揭示了SETD2缺失通过抑制RAS/ERK信号通路加剧SMAD4缺失引起的结直肠癌进展。

标题:Loss of SETD2 aggravates colorectal cancer progression caused by SMAD4 deletion through the RAS/ERK signalling pathway(SETD2缺失通过抑制RAS/ERK信号通路加剧SMAD4缺失引起的结直肠癌进展)
时间:2023-11-14
期刊:Clinical and Translational Medicine
影响因子:IF 10.6
技术平台:ChIP-seq、RNA-seq、WB等
研究摘要:
本研究以小鼠结直肠癌组织和SW620细胞为研究对象,对结直肠癌患者的临床数据库进行分析,以研究SETD2与SMAD4的相关性。研究人员首先构建了SETD2和SMAD4双敲除(double knockdown)小鼠,以进一步研究SETD2在SMAD4缺失型CRC中的作用。分离肠上皮细胞(IECs)进行RNA测序(RNA-seq)和染色质免疫沉淀测序(ChIP-seq),以研究CRC的发生机制和关键分子。通过分子实验和细胞实验分析SETD2在SMAD4缺失型CRC中的作用。最后通过挽救实验来验证SETD2在SMAD4缺失型CRC发生的分子机制。
实验结果表明,SETD2缺失促进了SMAD4缺失型CRC的恶性进展。Smad4Vil-KO;Setd2Vil-KO小鼠在经AOM/DSS诱导后,表现出更严重的CRC表型:肿瘤更大,上皮增殖率更高。进一步的机制研究结果表明,SETD2缺失导致DUSP7下调,DUSP7参与抑制RAS/ERK信号通路。最后,ERK1/2抑制剂SCH772984显著减缓了Smad4Vil-KO;Setd2Vil-KO小鼠的CRC进展,DUSP7过表达显著抑制了SETD2KO;SMAD4KO 小鼠SW620细胞的增殖率。
本研究结果表明,SETD2通过促进smad4缺失CRC中DUSP7的转录来抑制RAS/ERK信号通路,这可能为晚期CRC的治疗提供潜在的治疗靶点。
- SETD2缺失促进SMAD4缺失型CRC进展。
- SETD2缺失通过RAS/ERK信号通路加重SMAD4缺失型CRC。
- SETD2通过促进DUSP7转录抑制RAS/ERK信号通路。
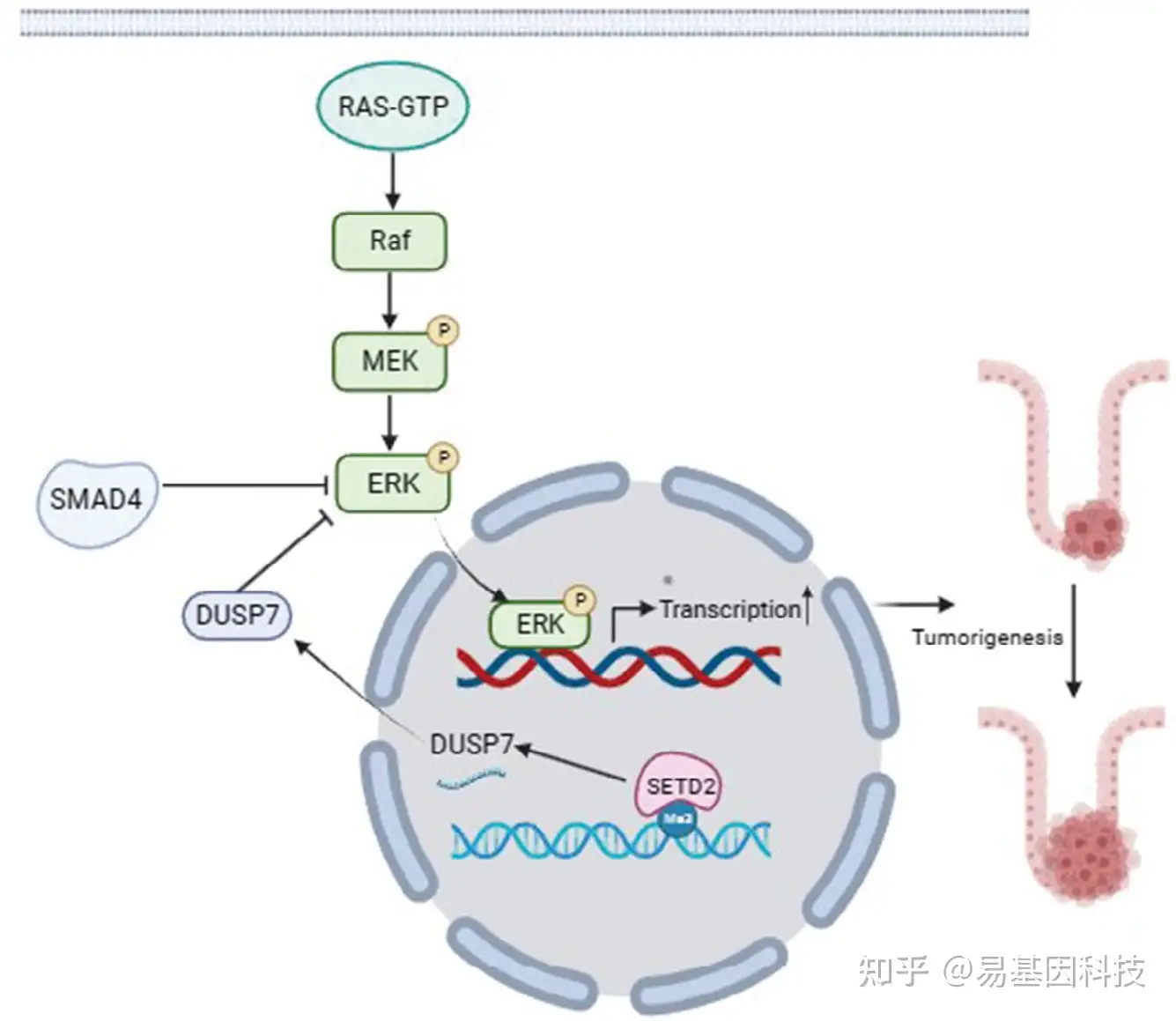
图形摘要
研究结果:
(1)在晚期CRC中,SETD2和SMAD4共突变率显著升高
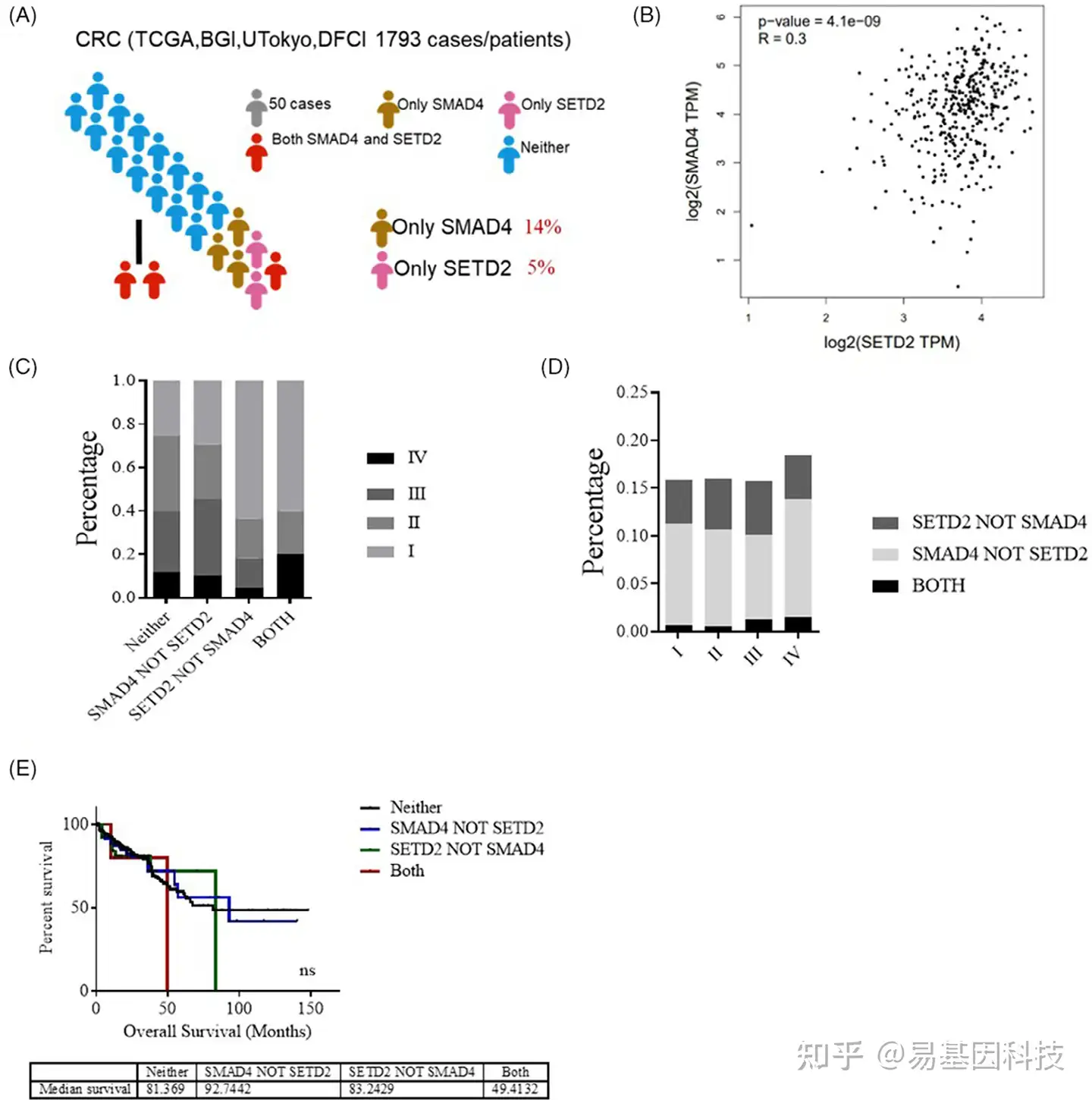
图1:晚期结直肠癌患者中SETD2和SMAD4共突变率升高。
- TCGA数据库中1793例患者样本中SMAD4和SETD2比例分析。
- TCGA数据库中CRC患者中SMAD4和SETD2 mRNA表达水平相关性分析。
- 1793例结直肠癌患者SETD2和SMAD4突变与结直肠癌分期的比例分析。
- TCGA数据库分析不同癌症分期与结直肠癌患者SETD2和SMAD4突变的相关性。
- TCGA数据库中无突变、SMAD4突变而STED2无突变、SETD2突变而SMAD4无突变、SMAD4和SETD2共突变患者的生存期统计。
(2)SETD2缺失促进Smad4Vil-KO;Setd2Vil-KO小鼠肠息肉病的形成
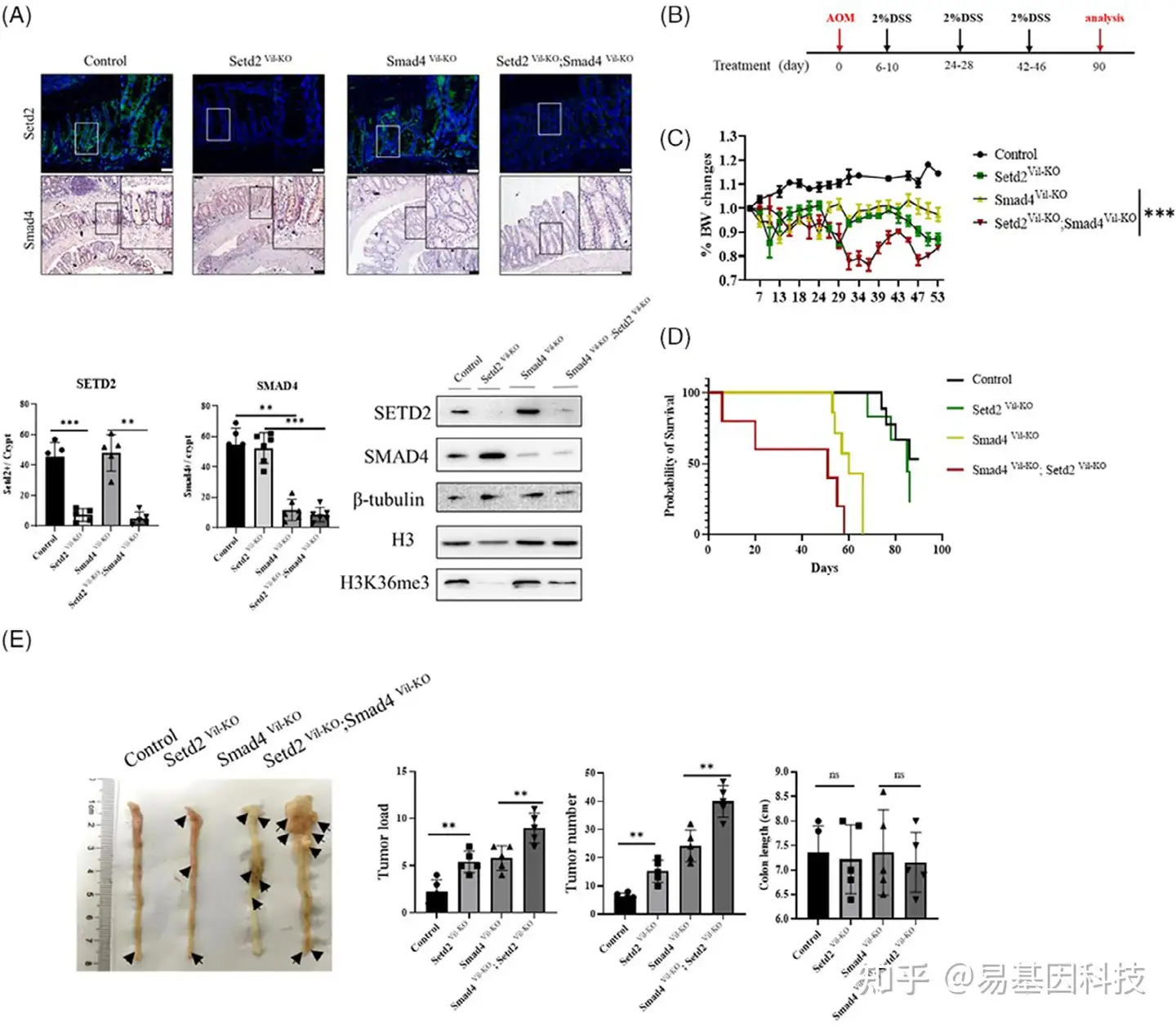
图2:SETD2缺失促进小鼠SMAD4缺失型CRC的形成。
(A) 免疫荧光和免疫组化方法分析SETD2和SMAD4的表达,Western blot分析SETD2、SMAD4和H3K36me3的表达。比例尺:200μm。数据表示平均值±S.D.,通过双尾Student t检验确定统计学显著性。*p<0.05;**,p<0.01;***,p<0.001。(B) 诱导小鼠CRC的AOM/DSS方案示意图。
(C-D) 小鼠腹膜内注射10mg/kg AOM后,在饮用水中添加2%DSS喂养小鼠,并记录体重(C)和生存期(D)的降低(每个基因型n=6)。
(E) AOM注射后90天,处死小鼠以检查肿瘤负荷和结肠长度(每个基因型n=6)。比例尺:1cm。
(3)在体内和体外实验中,SETD2沉默加剧了SMAD4缺失型CRC的恶性进展
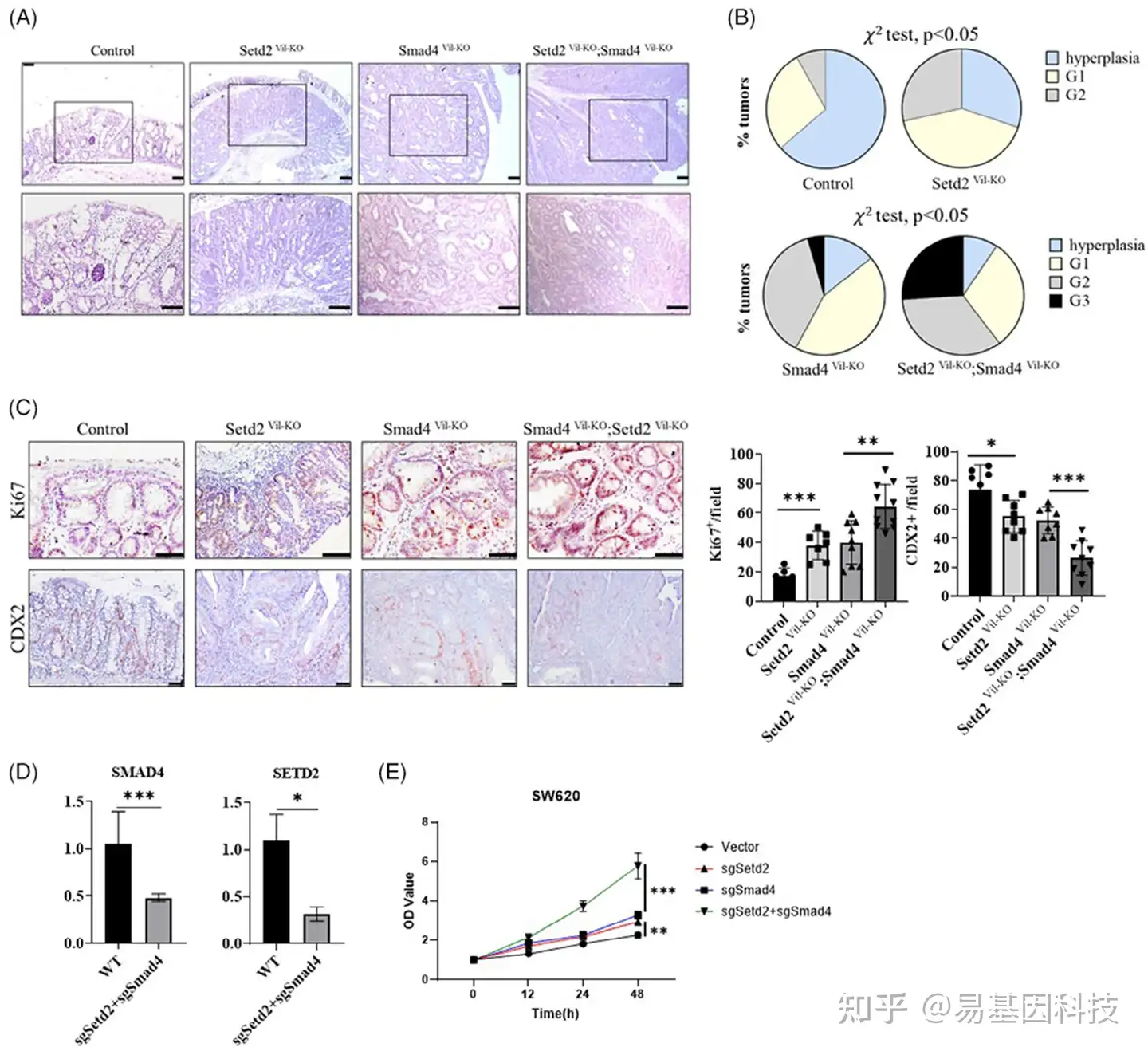
图3:在体内和体外实验中,SETD2沉默加剧了SMAD4缺失型CRC的恶性进展
(A-B) AOM/DSS处理小鼠结肠组织的H&E染色切片,各基因型结肠和肿瘤总体分级的代表性图像(χ2检验)(每个基因型n=5)。比例尺:上200 μm,下100 μm。
(C) 各基因型AOM/DSS处理小鼠Ki67和CDX2染色的代表性图像(每个基因型n=5)。标尺:50 μm(上),100 μm(下)。数据代表平均值±S.D.,统计学显著性采用双尾学生t检验。*,p<0.05;**,p < 0.01;***,p<0.001。
(D-E) 分别用空载体、sgSMAD4质粒和sgSETD2质粒转染SW620细胞,采用实时荧光定量PCR检测mRNA表达水平(每个基因型n=4)。采用CCK8进行检测。数据代表平均值±S.D.,统计学显著性采用双尾学生t检验。*,p<0.05;***,p<0.001。
(4)SETD2通过激活RAS/ERK信号通路加重SMAD4消融诱导的结直肠癌(CRC)

图4: SETD2通过激活RAS/ERK信号通路加重SMAD4消融诱导的结直肠癌。
- 比较AOM/DSS处理10 d小鼠IECs中基因表达的RNA-seq数据热图。右侧为基因表达变化的KEGG term分析。
- AOM/DSS处理(10 d)小鼠IECs中与代谢通路相关的mRNA表达水平(每个基因型n=4)。
- SETD2缺失相关差异表达基因的GSEA富集图。
- RAS/ERK信号通路相关基因表达的RNA-seq结果热图。
- AOM/DSS处理(10 d)小鼠IECs中与RAS/ERK信号通路相关的mRNA表达水平(每个基因型n=4)。
- AOM/DSS处理(10 d)小鼠IECs中目标蛋白的Western blot分析。
- AOM/DSS处理(10 d)小鼠p-p44/p42 MAPK染色。比例尺:100 μm。数据代表平均值±S.D.,通过双尾Student t检验确定统计学显著性。**,p<0.01。
(5)SETD2通过促进RAS/ERK信号通路中的DUSP7转录来抑制CRC进展
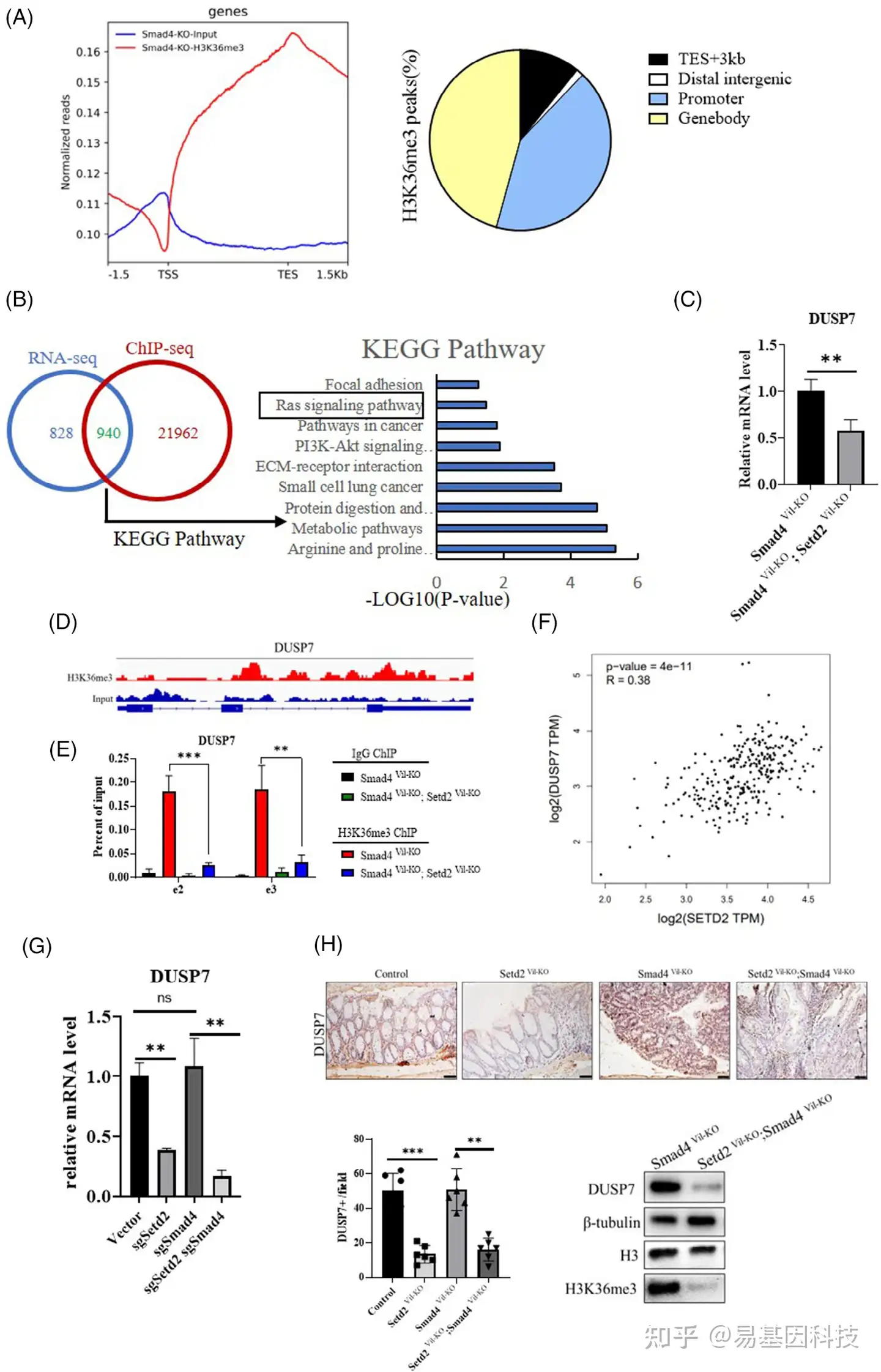
图5:SETD2通过促进RAS/ERK信号通路中DUSP7转录来抑制CRC进展。
- AOM/DSS处理10 d后的Smad4Vil-KO小鼠IECs中H3K36me3 ChIP-seq信号的归一化reads密度。H3K36me3 ChIP-seq peaks在基因体和基因间区域的分布分析。
- Venn图显示Smad4Vil-KO IECs中携带H3K36me3结合并表现出表达变化的基因数量。右图显示重叠基因的KEGG分析。
- Smad4Vil-KO小鼠和Smad4Vil-KO;Setd2Vil-KO小鼠中DUSP7表达的RT- qPCR分析。
- AOM/DSS处理(10 d) 的Smad4Vil-KO小鼠和Smad4Vil-KO;Setd2Vil-KO小鼠中分离的IECs中DUSP7位点的H3K36me3 ChIP-seq信号快照。
- AOM/DSS处理(10 d) Smad4Vil-KO小鼠和Smad4Vil-KO;Setd2Vil-KO小鼠IECs中H3K36me3结合DUSP7位点的ChIP-qPCR分析,IgG作为对照(每个基因型n=3)。
- TCGA数据库中CRC样本中SETD2和DUSP7表达水平之间的相关性分析。
- RT-qPCR分析转染sgSETD2和sgSMAD4质粒的SW620细胞中mRNA的相对表达水平(每个基因型n = 5)。
- AOM/DSS处理小鼠DUSP7染色。比例尺:50 μm。数据表示平均值±S.D.,通过双尾Student t检验确定统计学显著性。**,p<0.01;***,p<0.001。从AOM/DSS处理的小鼠分离的IEC中DUSP7蛋白的Western blot分析。
(6)抑制RAS/ERK信号通路可以延缓癌症进展
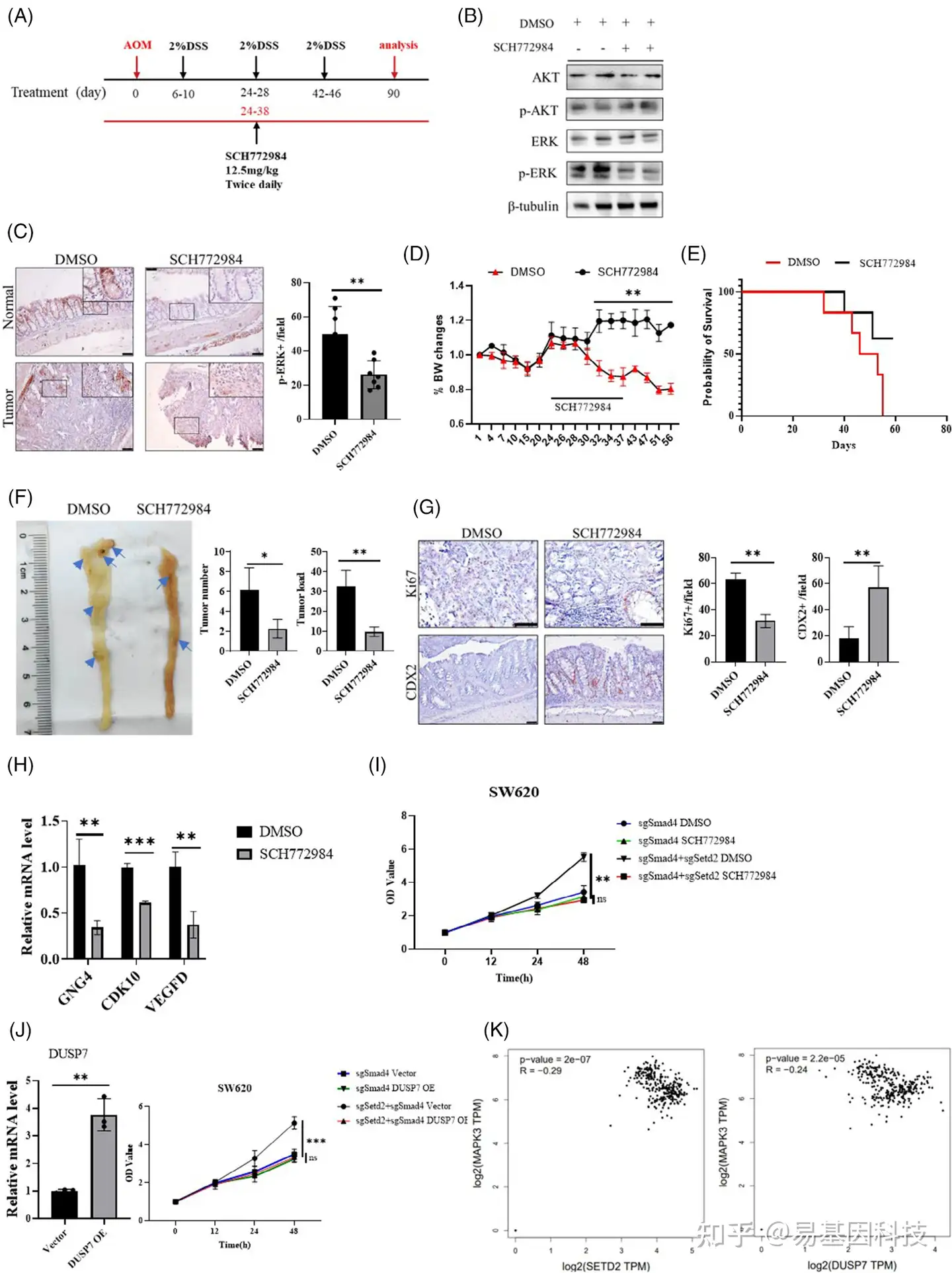
图6:抑制RAS/ERK信号通路可延缓癌症进展。
- 处理小鼠结直肠癌的SCH772984方案示意图。
- DMSO和SCH772984处理小鼠IECs目标定蛋白的Western blot分析。
- DMSO和SCH772984处理小鼠p-p44/p42 MAPK染色。比例尺:100 μm。数据代表平均值±S.D.,通过双尾Student t检验确定统计学显著性。**,p<0.01。
(D-E) 用DMSO和SCH772984处理小鼠,记录体重(D)和生存期(E)的降低(每个基因型n = 6)。
(F) AOM注射 90天后,处死小鼠以检测肿瘤负荷(每个基因型n = 6)。比例尺:1cm。
(G) AOM/ DSS诱导CRC后,DMSO和SCH772984处理的小鼠肿瘤中显示Ki67和CDX2染色的代表性图像(每个基因型n = 5)。标尺:50 μm(上),100 μm(下)。数据代表平均值±S.D.,通过双尾Student t检验确定统计学显著性。*,p < 0.05;**,p < 0.01;***, p<0.001。
(H) DMSO和SCH772984处理小鼠的IECs中与RAS/ERK信号通路相关的mRNA表达水平(每个基因型n=4)。
- 分别用DMSO和SCH772984处理SMAD4KO细胞和SMAD4KO;SETD2KO细胞(每个基因型n=4)。采用CCK8进行检测。数据代表平均值±S.D.,通过双尾Student t检验确定统计学显著性。**,p<0.01。
(J) 分别转染空载体和dup7 OE质粒的SW620细胞中DUSP7表达的RT- qPCR分析(每个基因型n=3)。采用CCK8进行检测。数据代表平均值±S.D.,通过双尾Student t检验确定统计学显著性。**,p<0.01;***,p<0.001。
(K) TCGA数据库中CRC样本中SETD2/ DUSP7与MAPK3表达水平的相关性分析。
研究意义
SETD2介导的H3K36me3通过促进SMAD4缺失型CRC中DUSP7的转录来抑制RAS/ERK信号通路,这可能为治疗晚期CRC提供潜在的治疗靶点。
关于易基因染色质免疫共沉淀测序 (ChIP-seq)
染色质免疫共沉淀(Chromatin Immunoprecipitation,ChIP),是研究体内蛋白质与DNA相互作用的经典方法。将ChIP与高通量测序技术相结合的ChIP-Seq技术,可在全基因组范围对特定蛋白的DNA结合位点进行高效而准确的筛选与鉴定,为研究的深入开展打下基础。
DNA与蛋白质的相互作用与基因的转录、染色质的空间构型和构象密切相关。运用组蛋白特定修饰的特异性抗体或DNA结合蛋白或转录因子特异性抗体富集与其结合的DNA片段,并进行纯化和文库构建,然后进行高通量测序,通过将获得的数据与参考基因组精确比对,研究人员可获得全基因组范围内某种修饰类型的特定组蛋白或转录因子与基因组DNA序列之间的关系,也可对多个样品进行差异比较。
应用方向:
ChIP 用来在空间上和时间上不同蛋白沿基因或基因组定位
- 转录因子和辅因子结合作用
- 复制因子和 DNA 修复蛋白
- 组蛋白修饰和变异组蛋白
技术优势:
- 物种范围广:细胞、动物组织、植物组织、细菌微生物多物种富集经验;
- 微量建库:只需5ng以上免疫沉淀后的DNA,即可展开测序分析;
- 方案灵活:根据不同的项目需求,选择不同的组蛋白修饰特异性抗体。
技术路线:
易基因提供全面的DNA与蛋白互作测序方案,详询易基因:0755-28317900.
参考文献:
Ma C, Liu M, Feng W, Rao H, Zhang W, Liu C, Xu Y, Wang Z, Teng Y, Yang X, Ni L, Xu J, Gao WQ, Lu B, Li L. Loss of SETD2 aggravates colorectal cancer progression caused by SMAD4 deletion through the RAS/ERK signalling pathway. Clin Transl Med. 2023 Nov;13(11):e1475. doi: 10.1002/ctm2.1475. PubMed PMID: 37962020.
相关阅读:
Nature子刊:ChIP-seq等组学研究鉴定出结直肠癌的致癌超级增强子
项目文章 | 组蛋白ChIP-seq揭示烟粉虱共生菌Hamiltonella调控宿主生殖新机制
项目文章 | NAR:ChIP-seq等揭示蛋白质酰基化与c-di-GMP协同调控放线菌发育与抗生素合成机制
项目集锦 | 易基因近期染色质免疫共沉淀测序(ChIP-seq)研究成果
标签:seq,SMAD4,ChIP,小鼠,CRC,CTM,SETD2,AOM,ERK From: https://www.cnblogs.com/E-GENE/p/17876885.html